View in English?
View in English?
Cleavage Under Targets & Release Using Nuclease (CUT&RUN) アッセイは、ChIP-qPCRやChIP-seqと比較して、より少ない細胞で、より迅速に解析できる費用対効果の高いタンパク質-DNA相互作用の解析方法です。Cell Signaling Technology® (CST®) は、CUT&RUNキットを改良するためにプロトコールの追加検証と最適化を行い、タンパク質-DNA相互作用を調べる方法の選択肢をさらに増やしました。
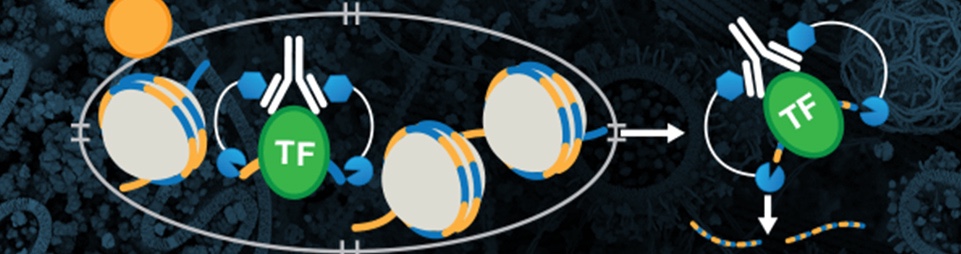
CUT&RUNは、シーケンシングコストが低く、様々な抗体や標的に対応したin vivoの手法であり、1 - 2日で結果が得られます。アッセイの柔軟性を高めるため、CSTのCUT&RUN Assay Kitには、新たに以下の利点が追加されました。
| プロトコールが改善 | 改善されたプロトコールを利用することで、存在量の少ない、あるいは結合の弱い転写因子やコファクターの濃縮が促進される |
| 必要サンプル量が低減 | ヒストン修飾因子:5,000 - 10,000個 転写因子やコファクター:10,000 - 20,000個 |
| 固定した細胞に対応 | 細胞の状態を維持するよう軽く固定することにより、細胞のシグナル伝達の保持や巨大複合体の構成成分の濃縮が促進される |
| 固定/非固定の組織サンプルから信頼性の高いデータが得られる | 検証、最適化された組織用プロトコールを利用することで、ChIPの1/20以下のサンプルから信頼性の高いデータが得られる |
| 初代培養細胞を用いてタンパク質-DNA相互作用の研究が可能 | CUT&RUNは微量サンプルの解析が可能で、初代培養細胞の解析にも適する |
さらに、CSTのCUT&RUN Assay Kitでは、これまで通り下記のことが可能です。
| 短時間でデータ取得が可能 | わずか1 - 2日で細胞から目的のDNAが得られる |
| 低深度のシーケンシング=シーケンシングコストが低減 | アッセイ時のバックグラウンドが低いため、必要な高品質リードはわずか300 - 500万リード |
| In Vivoを反映 | 未変性のクロマチンを用いた解析のため、生体内を反映しやすい |
| 多様な抗体に対応 | ラビット抗体およびマウス抗体を利用できる |
| 多様な標的に対応 | ヒストンやヒストン修飾、転写因子、コファクターのqPCRデータやシーケンシングデータが得られる |
| 直線的な定量 | Spike-In DNAを利用してサンプル間のシグナルを標準化することができる |
CUT&RUNのプロトコール開発を続けた結果、信頼性の高いデータが得られるようプロトコールが最適化され、CUT&RUN Assay Kitがより多くの種類のサンプルに使用できるようになりました。例えば、バックグラウンドを大幅に増加させることなく目的のクロマチン断片を回収するため、現在CSTはDNAの消化を0℃ではなく4℃で行うことを推奨しています。これにより、特に転写因子やコファクターを対象としたqPCRやNG-seqのデータが改善されます。
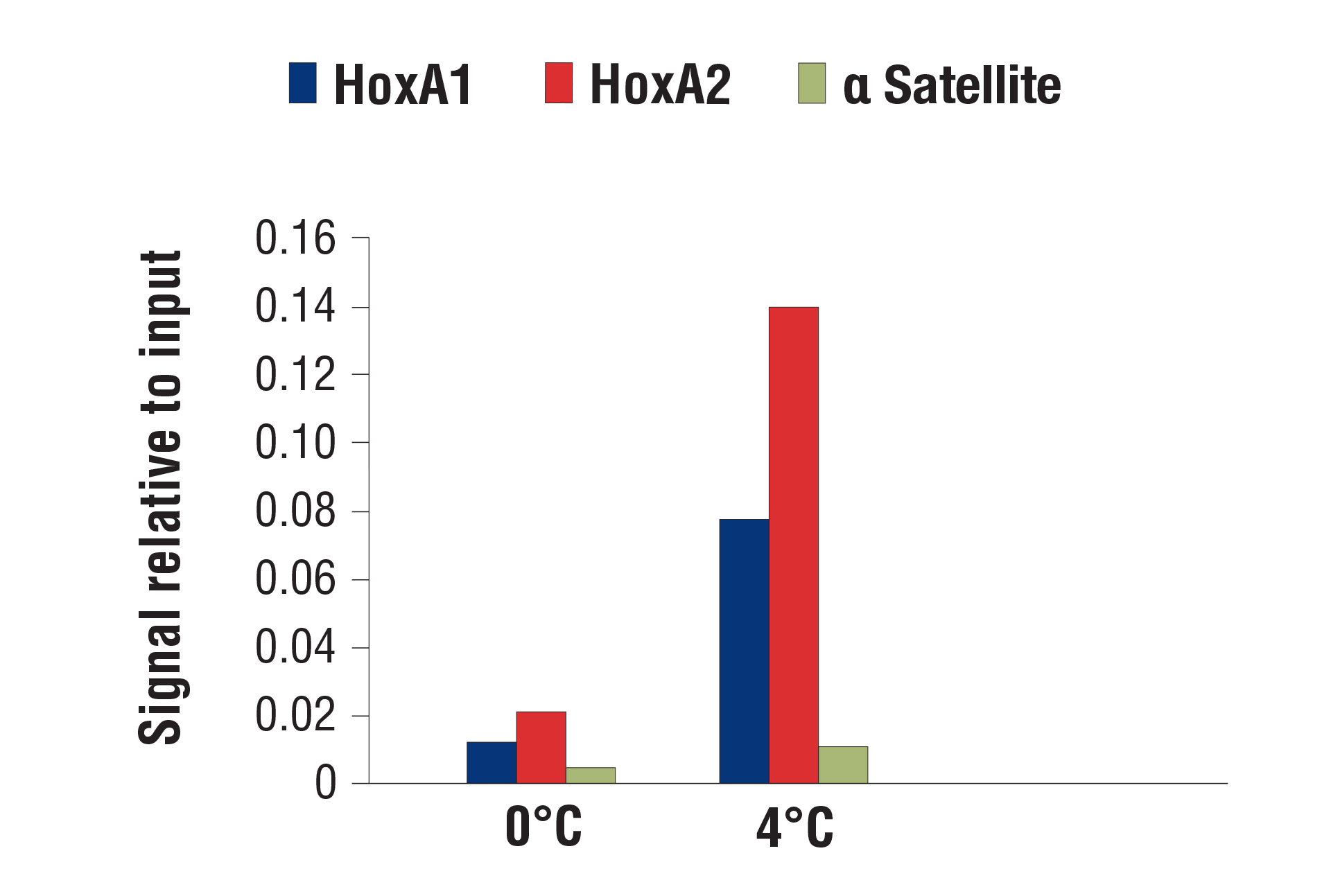
パネルA
パネルB
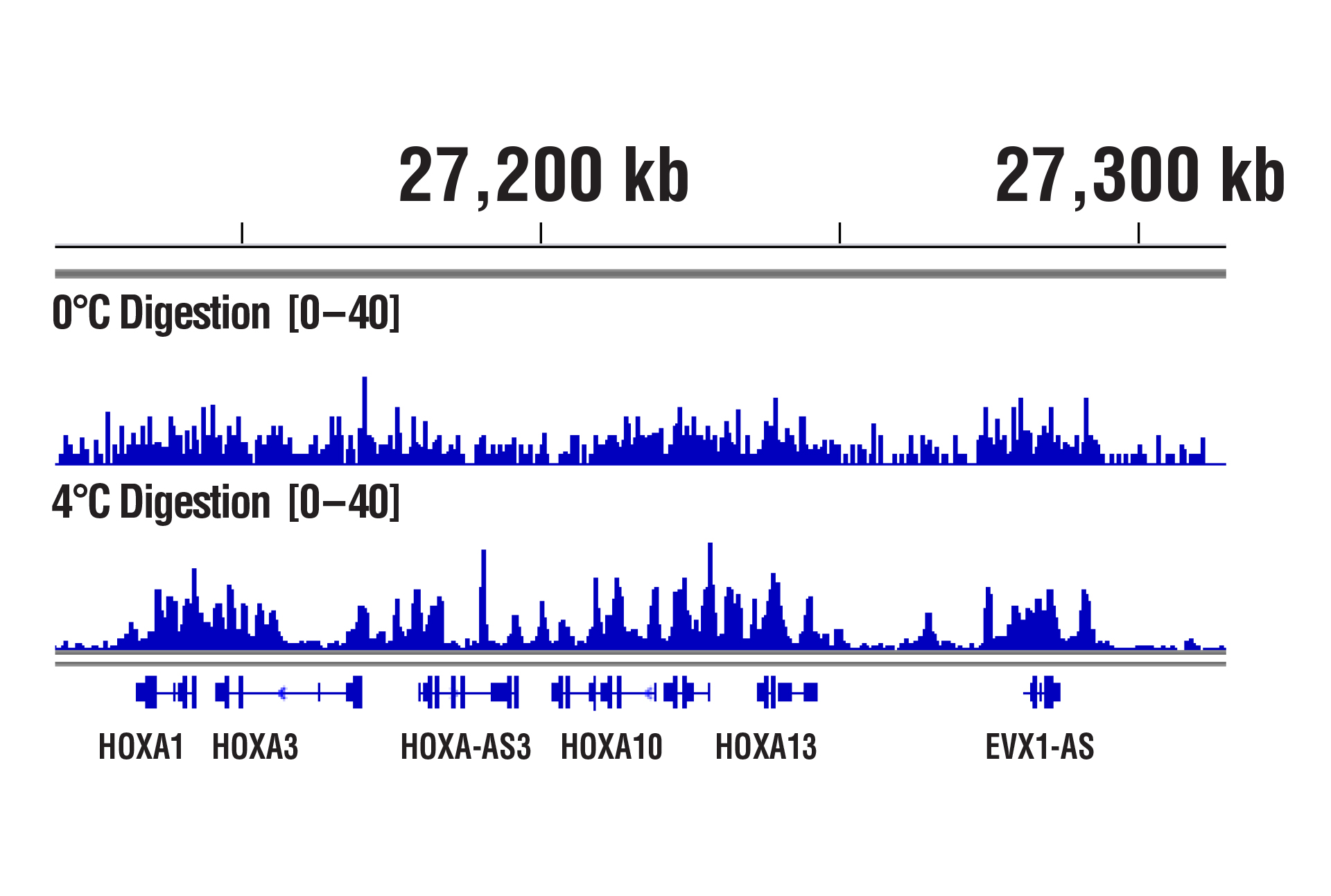
CUT&RUN Assay Kit #86652を用い、SUZ12 (D39F6) XP® Rabbit mAb #3737によるNCCIT細胞のCUT&RUNアッセイ行いました。図に示したように、DNA消化は0°Cまたは4°Cで行いました。パネルAでは、SimpleChIP® Human HoxA1 Intron 1 Primers #7707、SimpleChIP® Human HoxA2 Promoter Primers #5517、SimpleChIP® Human α Satellite Repeat Primers #4486のプライマーセットを用いて濃縮したDNAをリアルタイムPCRで定量しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。パネルBでは、DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、HoxA遺伝子全体へのSUZ12の結合を示しています。
CUT&RUNは、ChIP-qPCRやChIP-seqと比較して、信頼性の高いデータを得るために必要な細胞数が大幅に減少しています。CSTは、さらに少ないサンプルで済むようにプロトコールを最適化することで、実験時間の短縮を実現しています。
100,000個の細胞を集められない場合でも、CUT&RUNアッセイを実施したいことはありませんか?CUT&RUNでは、5,000 - 20,000個の細胞からでも同じ品質のデータが得られるため、問題ありません。
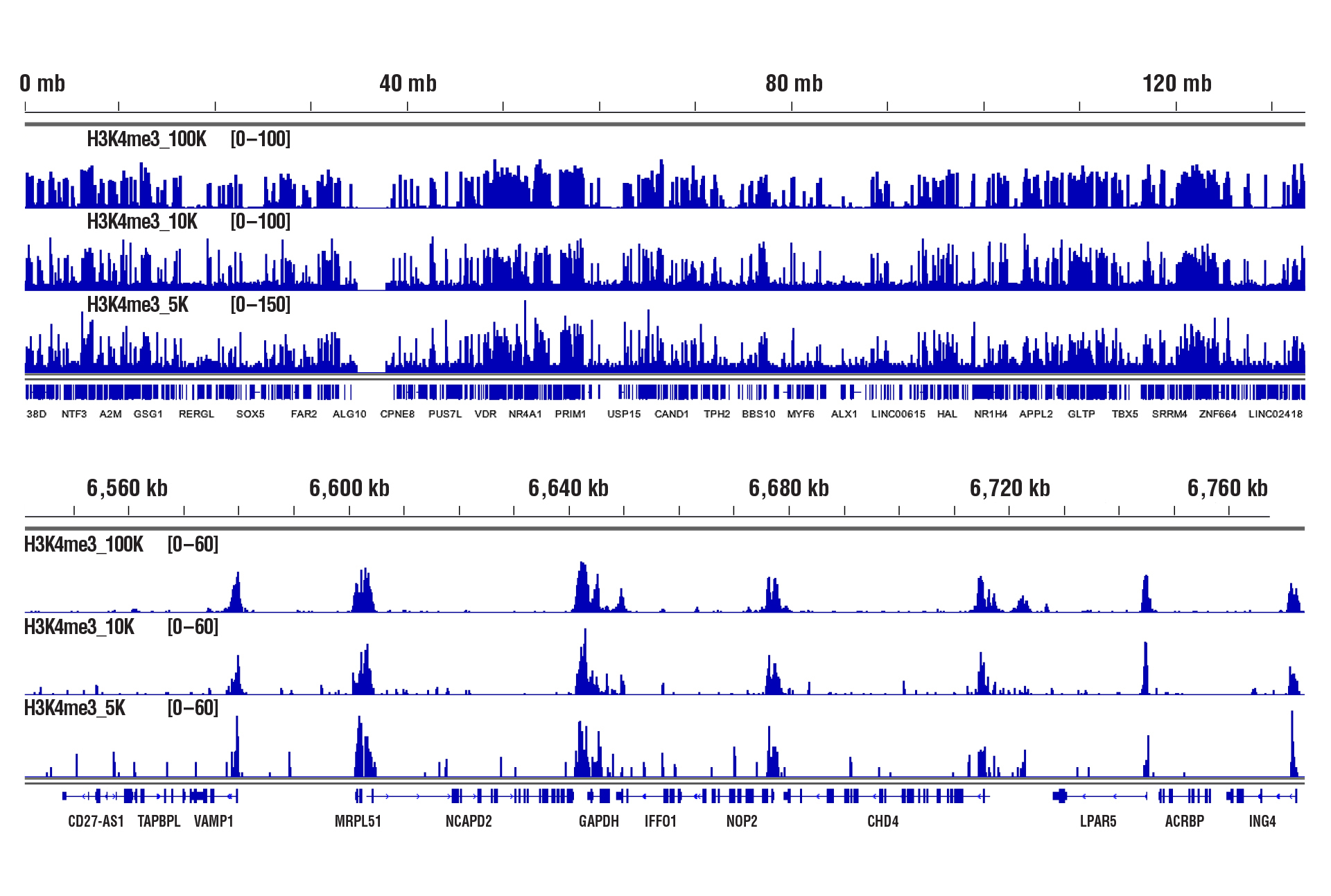
100,000個、10,000個または5,000個の細胞に対してCUT&RUN Assay Kit #86652を用いたヒストンの解析を行うと、いずれの細胞数においても同様の結合パターンが観察されました。
パネルA
パネルB
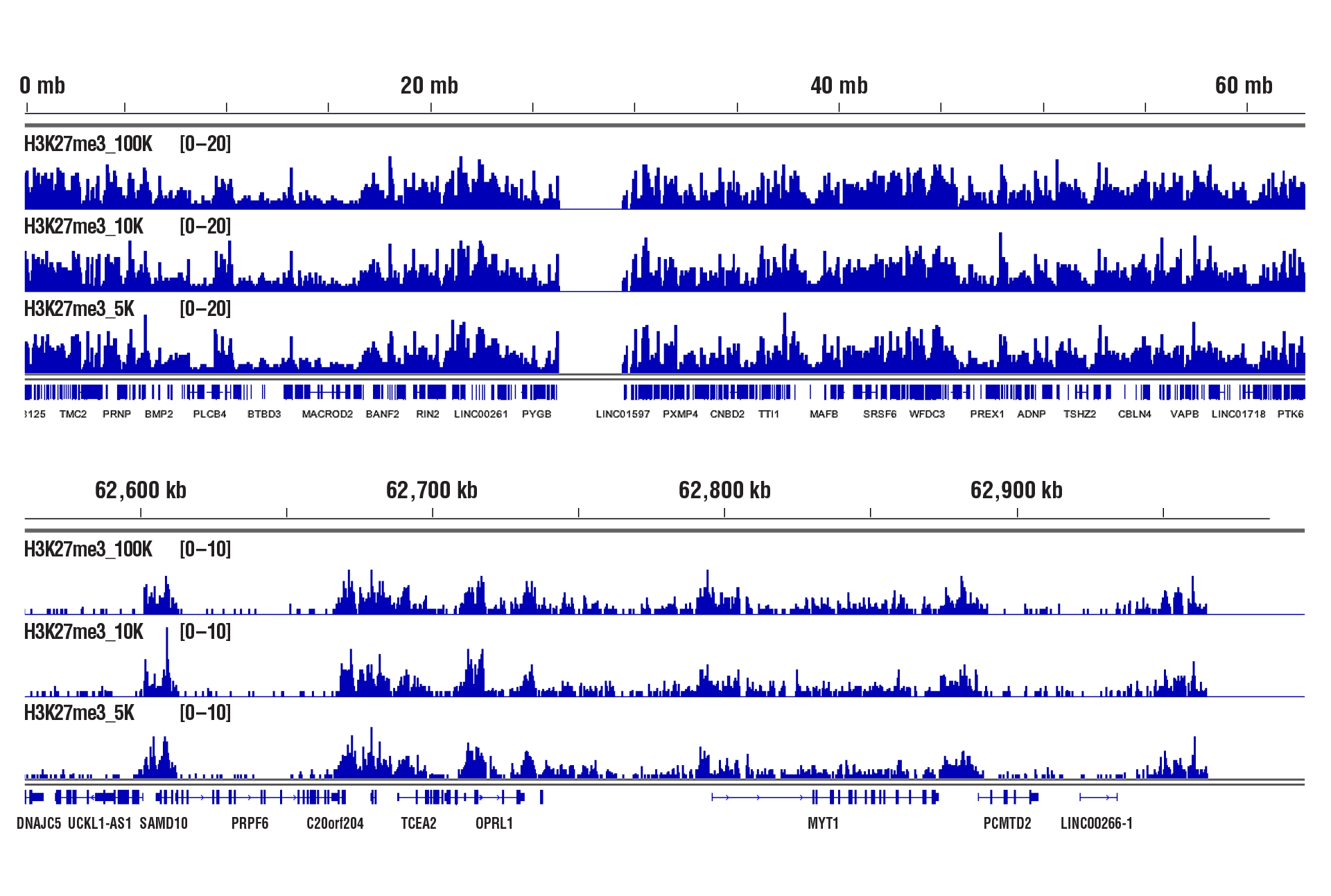
CUT&RUN Assay Kit #86652を用いて細胞数を減らしながらCUT&RUNアッセイを行いました。パネルAでは、100,000個、10,000個または5,000個のHCT 116細胞に対してTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いて解析しています。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、12番染色体全体にわたる結合 (上パネル) と、これに含まれるH3K4me3の既知の標的遺伝子であるGAPDHへの結合 (下パネル) を示しています。パネルBでは、CUT&RUNは100,000個、10,000個、または5,000個のHeLa細胞対してTri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733を用いて行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、20番染色体全体 (上) および、H3K27me3の既知の標的遺伝子であるMYT1 (下) への結合を示しています。
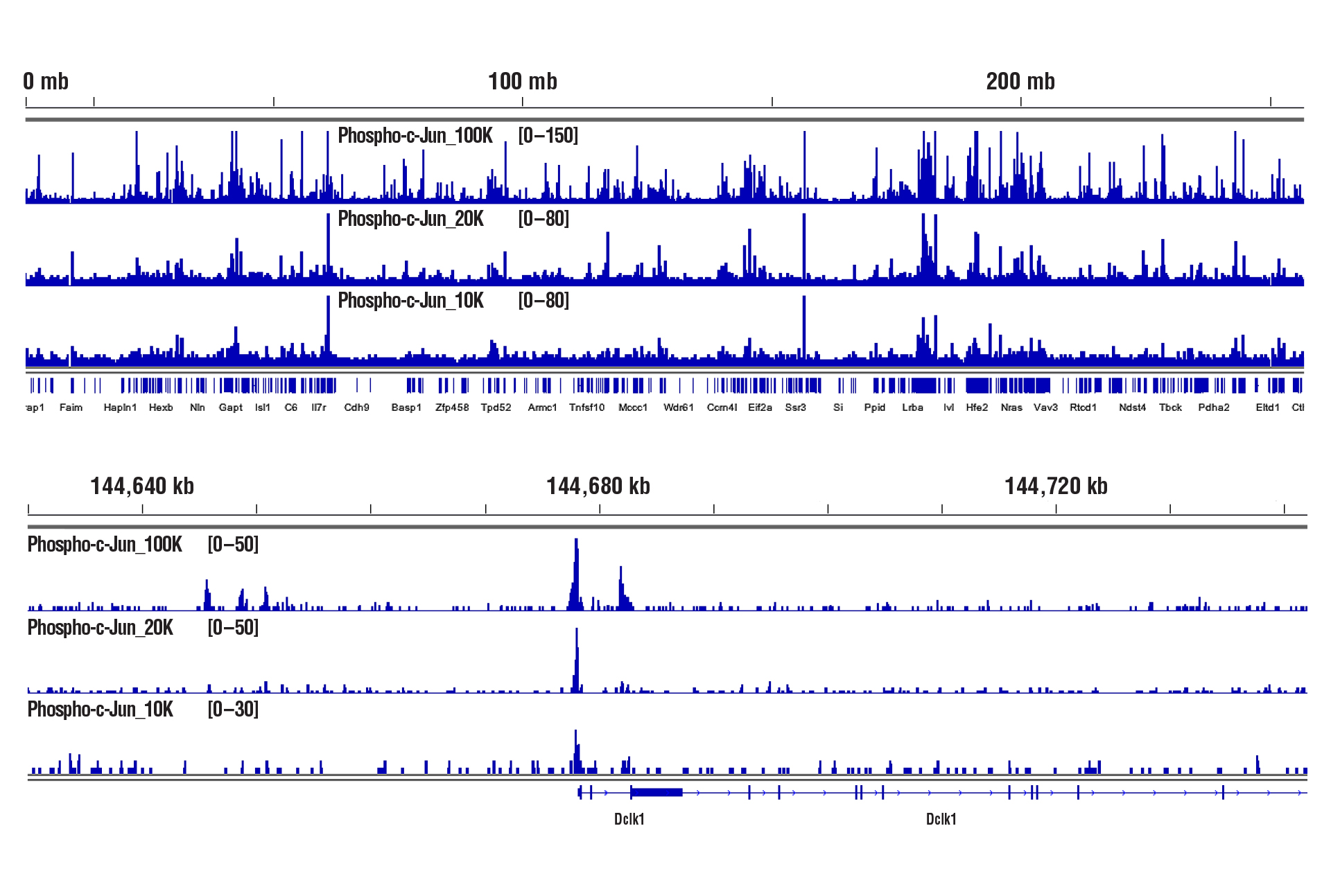
100,000個、20,000個または10,000個の細胞に対してCUT&RUN Assay Kit #86652を用いた転写因子の解析を行うと、いずれの細胞数においても同様の結合パターンが観察されました。
パネルA
パネルB
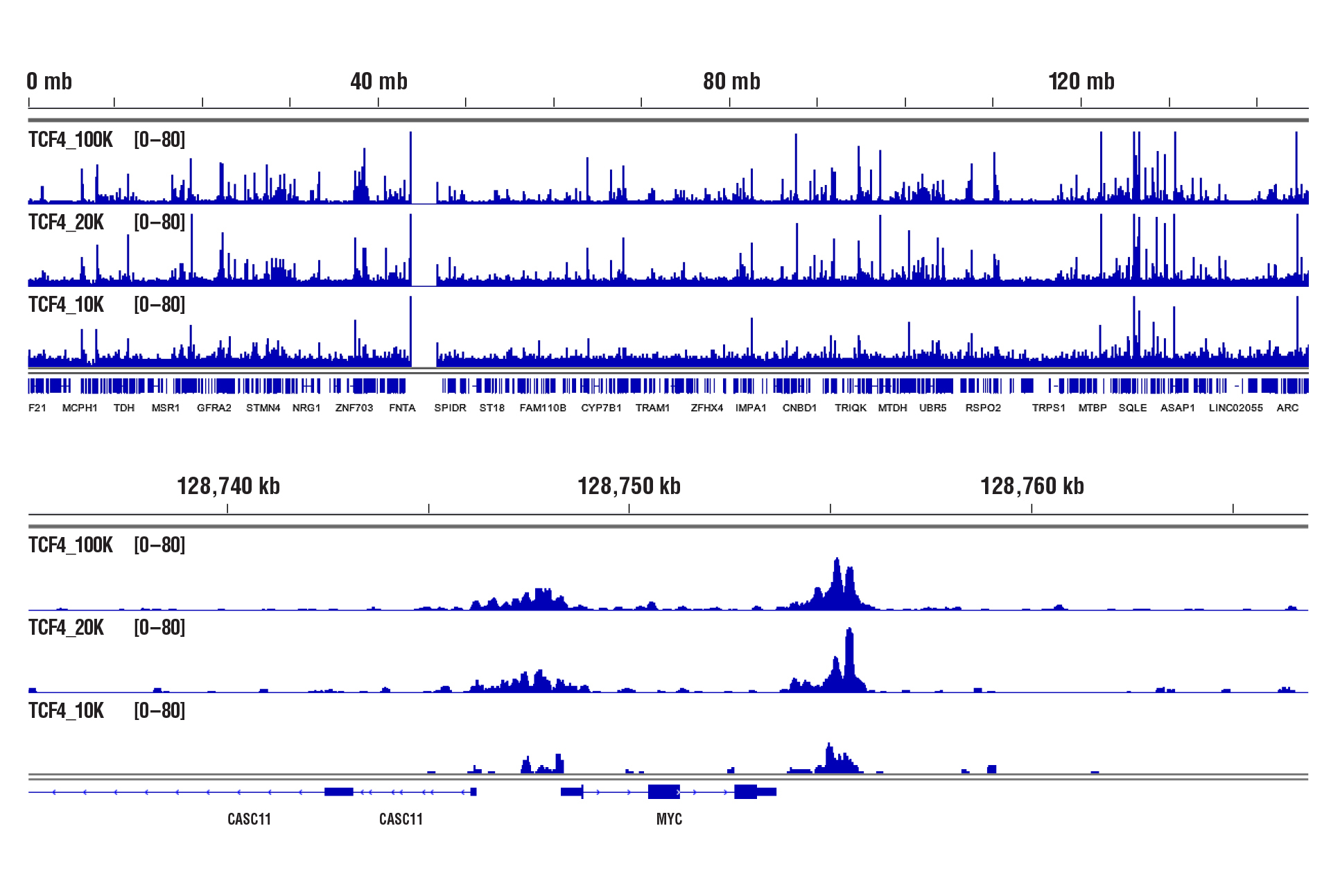
CUT&RUN Assay Kit #86652を用いて細胞数を減らしながらCUT&RUNアッセイを行いました。パネルAでは、100,000個、20,000個または10,000個のPC-12細胞を一晩飢餓状態にし、Human β-Nerve Growth Factor (hβ-NGF) #5221 (50 ng/ml) を2時間処理したのち、Phospho-c-Jun (Ser73) (D47G9) XP® Rabbit mAb #3270を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Phospho-c-Junの既知の標的遺伝子であるDclk1 (下) および、これを含む2番染色体全体への結合 (上) を示しています。パネルBでは、100,000、20,000、または10,000のHCT116細胞およびTCF4/TCF7L2 (C48H11) Rabbit mAb #2569にCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、8番染色体全体への結合 (上) および、TCF4の既知の標的遺伝子であるMYC (下) への結合を示しています。
100,000個、20,000個または10,000個の細胞に対してCUT&RUN Assay Kit #86652を用いたコファクターの解析を行うと、いずれの細胞数においても同様の結合パターンが観察されました。
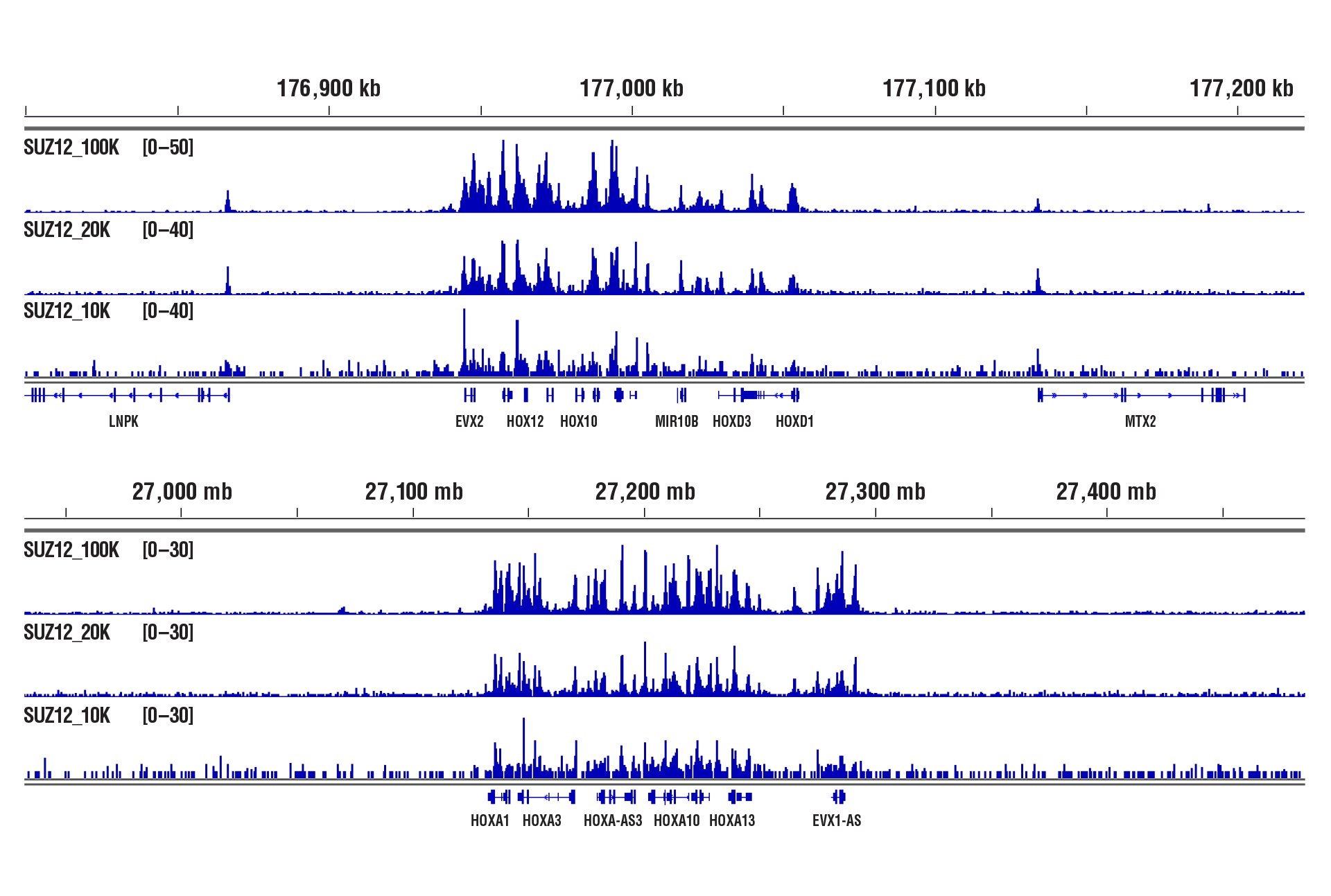
100,000個、20,000個または10,000個のNCCIT細胞に対しにSUZ12 (D39F6) XP® Rabbit mAb #3737とCUT&RUN Assay Kit #86652を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、SUZ12の既知の標的遺伝子であるHoxD (上) および、HoxAへの結合 (下) を示しています。
組織のように限られたサンプルでクロマチンのプロファイリングを行う場合、多くのサンプルを必要とするChIP実験には制限があります。CUT&RUNが培養細胞で実現した必要サンプルの削減は、組織サンプルにも当てはまります。組織により、ChIPアッセイは25-50 mgの組織サンプルを必要としますが、CUT&RUNキットは約1-2.5 mgの組織サンプルしか必要としません。
CSTのCUT&RUN Assay Kit #86652を用いたタンパク質-DNA相互作用の解析では、ChIPの1/20以下のサンプルから信頼性の高いデータが得られます。
CUT&RUNキットを用いたH3K4me3とMITFのタンパク質-DNA相互作用解析には、わずか1 mgの心臓組織が必要でした。
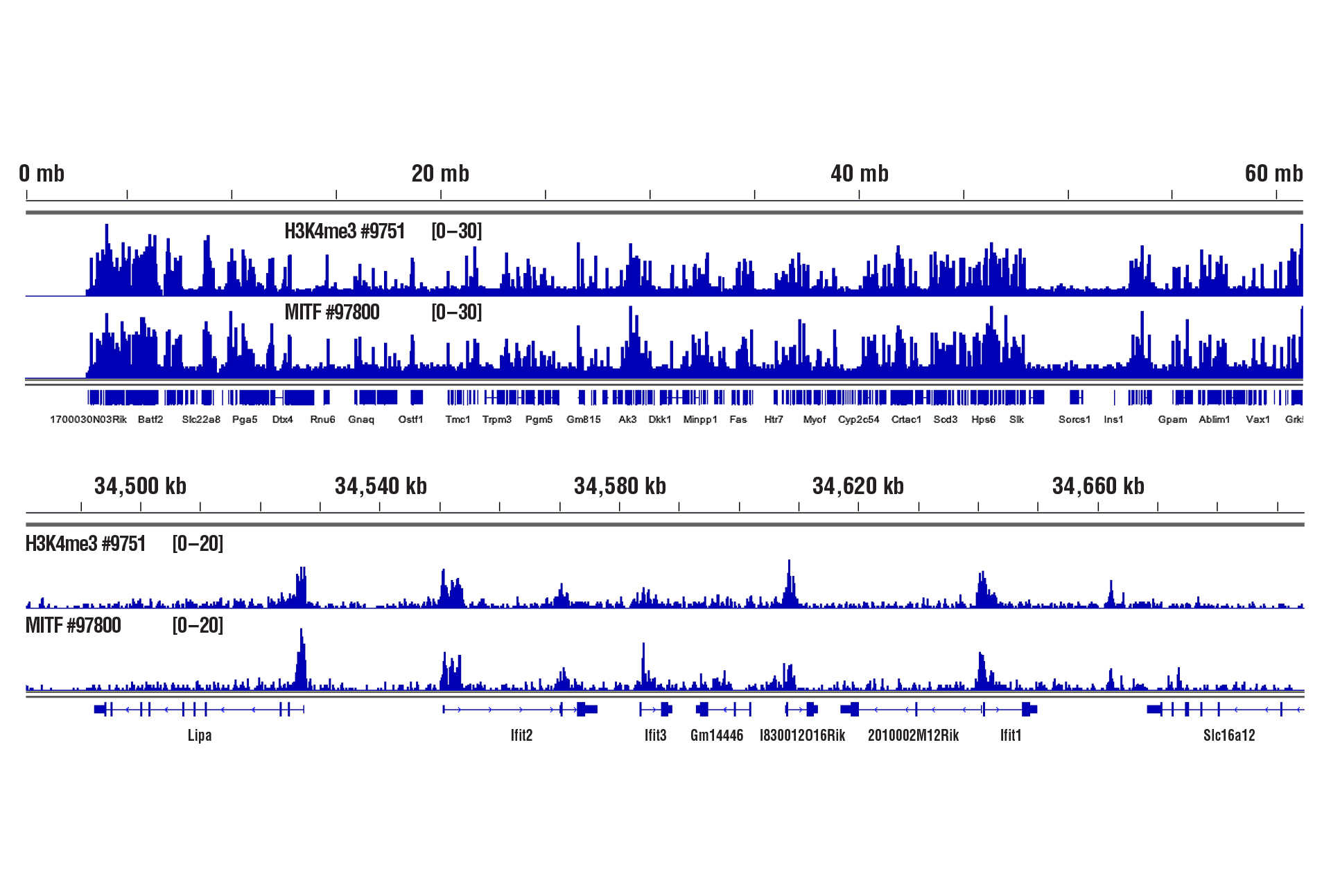
軽い固定 (0.1%ホルムアルデヒド、2分間) をした1 mgのマウス心臓組織に対してTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 またはMITF (D3B4T) Rabbit mAb #97800とCUT&RUN Assay Kit #86652を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Ifit2遺伝子 (下) および、これを含む19番染色体全体 (上) へのH3K4me3とMITFの結合を示しています。
CUT&RUNキットを用いたH3K4me3とBrn2/POU3F2のタンパク質-DNA相互作用解析には、わずか1 mgの脳組織が必要でした。
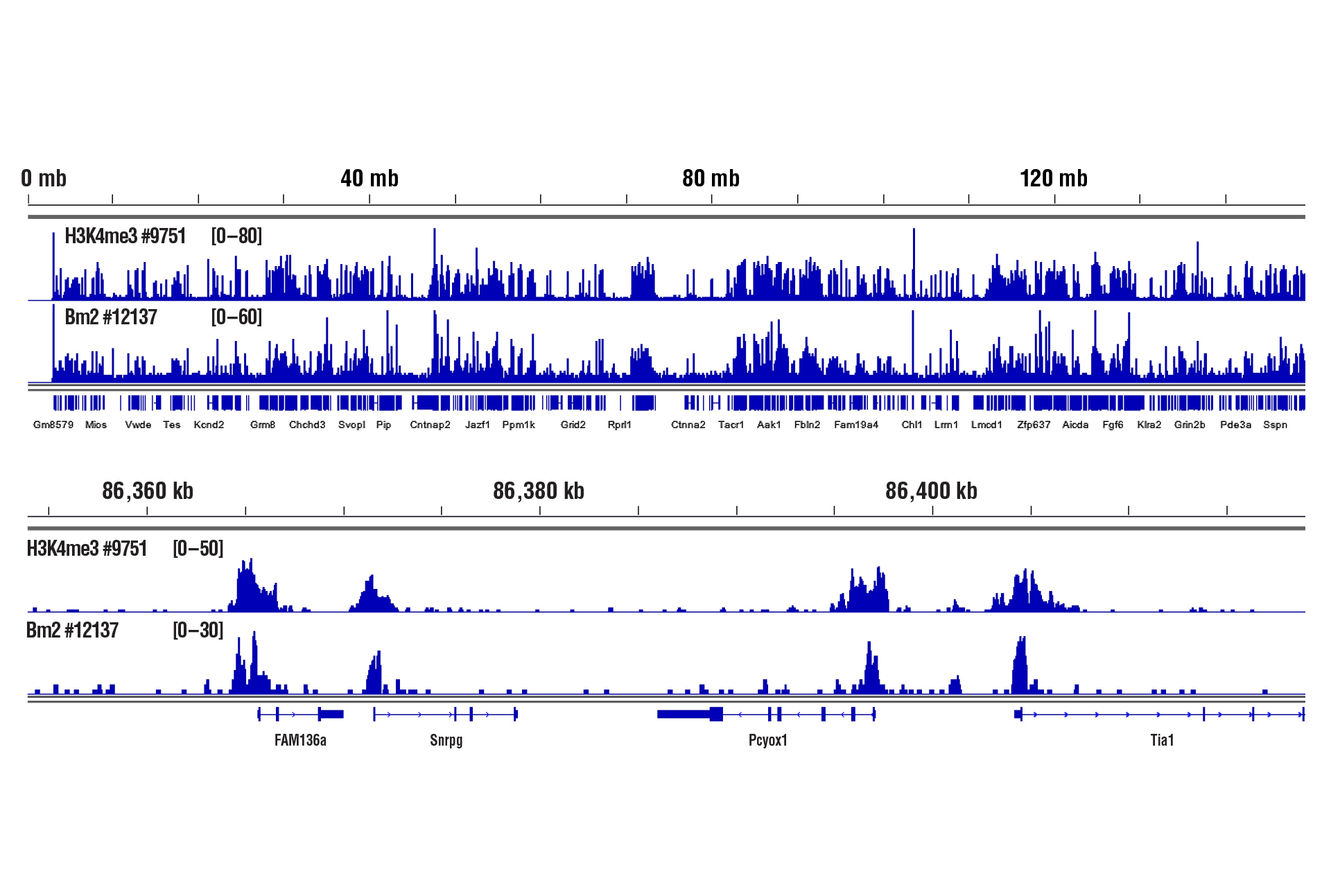
軽い固定 (0.1%ホルムアルデヒド、2分間) をした1 mgのマウス脳組織に対してTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはBrn2/POU3F2 (D2C1L) Rabbit mAb #12137とCUT&RUN Assay Kit #86652を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Snrpg遺伝子 (下) および、これを含む6番染色体全体 (上) へのH3K4me3とBrn2の結合を示しています。
CUT&RUNキットを用いたH3K4me3とグルココルチコイド受容体のタンパク質-DNA相互作用解析には、わずか1 mgの肝臓組織が必要でした。
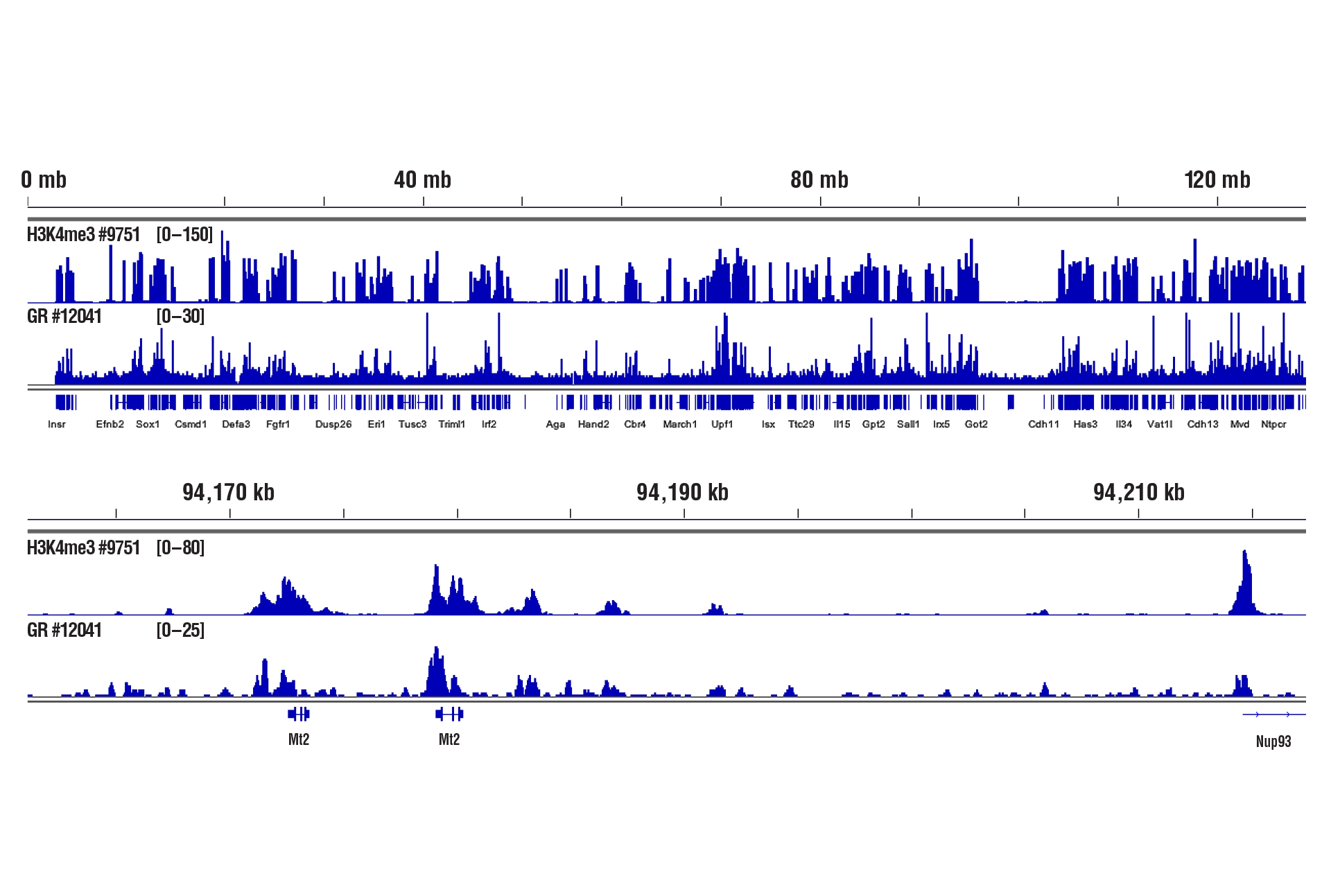
中程度の固定(0.1%ホルムアルデヒド、10分間) をした1 mgのマウス肝臓組織に対してTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 またはGlucocorticoid Receptor (D6H2L) XP® Rabbit mAb #12041とCUT&RUN Assay Kit #86652を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Mt2遺伝子 (下) および、これを含む8番染色体全体 (上) へのH3K4me3とグルココルチコイド受容体の結合を示しています。
2.5 mgの肝臓組織を用いたSUZ12とRING1Bのタンパク質-DNA相互作用解析をCUT&RUNキットは可能にします。
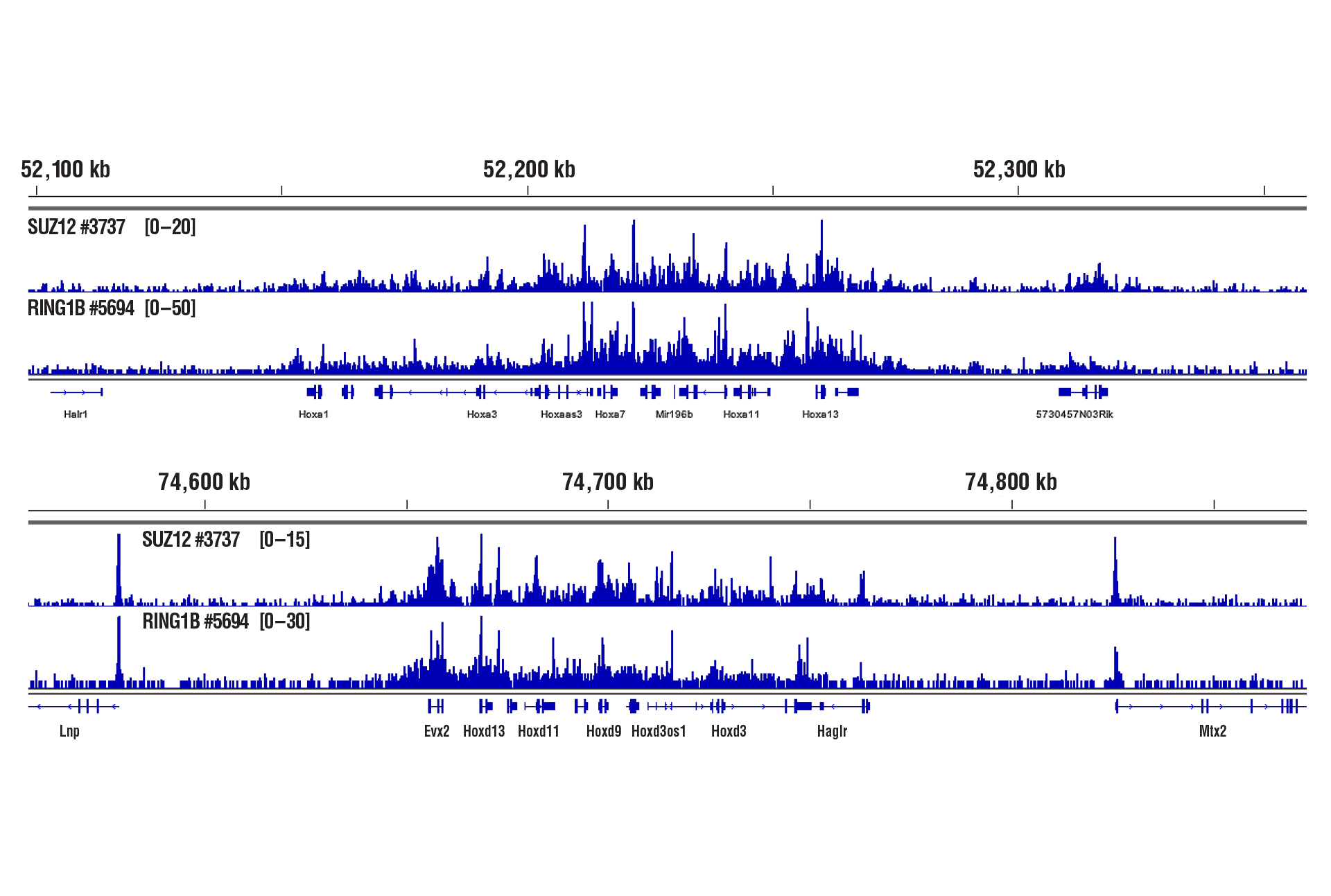
2.5mgのマウス肝臓組織に中程度の固定 (0.1%ホルムアルデヒド、10分間) を行ったのち、SUZ12 (D39F6) XP® Rabbit mAb #3737 またはRING1B (D22F2) XP® Rabbit mAb #5694とCUT&RUN Assay Kit #86652を用いてCUT&RUNアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、HoxA (上) およびHoxD (下) 遺伝子へに対するSUZ12とRING1Bの結合を示しています。
CUT&RUNが登場する以前、ChIPによる不死化されていない初代培養細胞のタンパク質-DNA相互作用解析は、アッセイに必要な細胞数を確保するのが困難なため、ほぼ不可能でした。
CUT&RUN Assay Kit #86652は、必要細胞数を大幅に削減し、初代培養細胞でのタンパク質-DNA相互作用研究を可能にします。細胞数の問題で、希望するサンプルの種類を妥協する必要はありません。
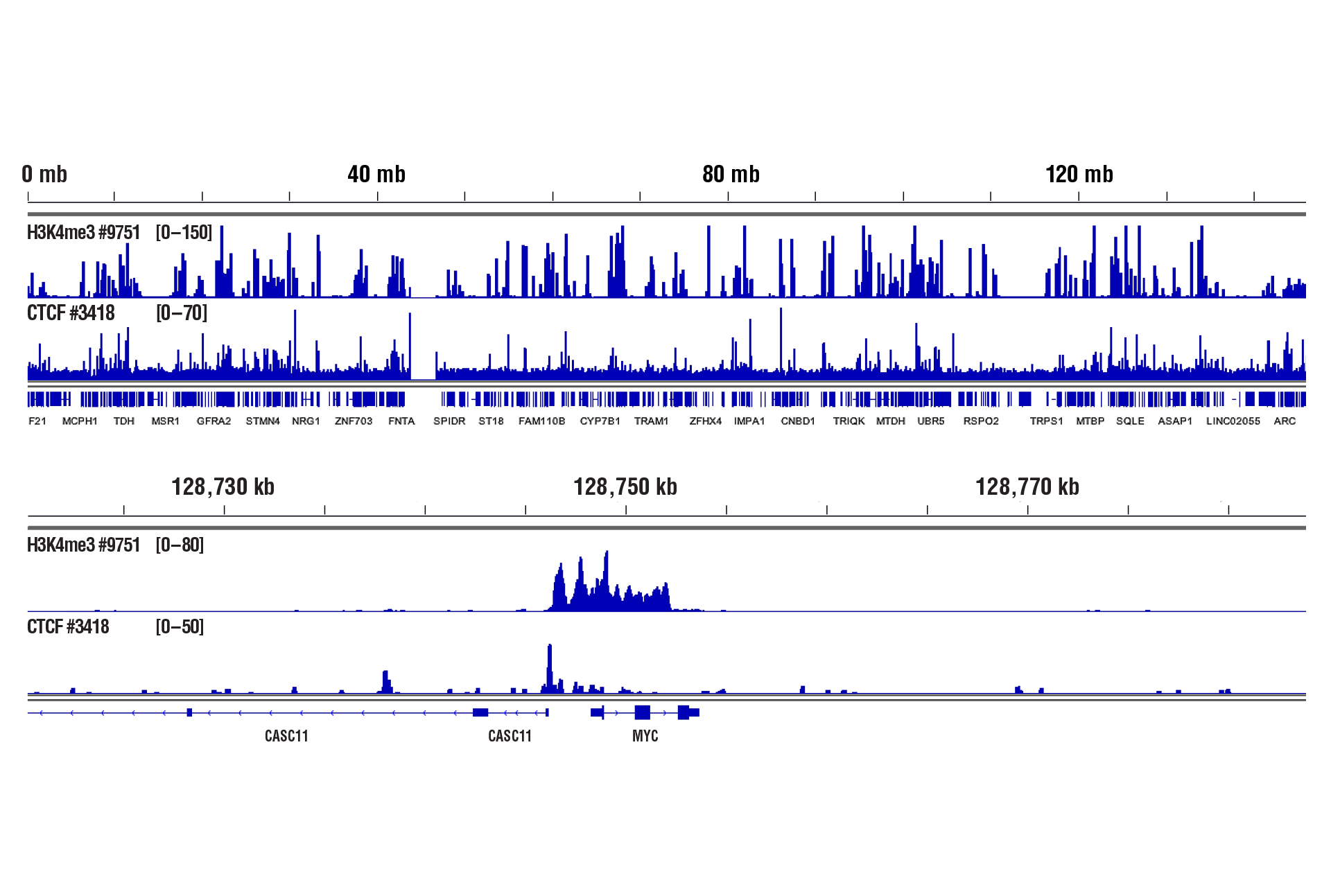
CUT&RUNは、100,000生ヒトCD8+ T細胞およびTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはCTCF (D31H2) XP3418® Rabbit mAb #86652を用い、CUT&RUN Assay Kit #により行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、8番染色体全体 (上) および、H3K4me3とCTCFの既知の標的遺伝子であるMYC (下) への結合を示しています。CUT&RUN-qPCRデータを含む追加のデータは、#3418のデータシートをご確認ください。
CSTは、CUT&RUNアッセイに必要なすべての試薬を含んだキットだけでなく、CUT&RUN関連製品を単品でも提供しており、必要な試薬だけ購入することもできます。これらの製品は、高品質であることを保証するために社内で厳格に検証されていますので、安心して実験にご使用いただけます。
| 製品番号 | 製品 |
| 86652 | CUT&RUN Assay Kit |
| 40366 | CUT&RUN pAG-MNase and Spike-in DNA |
| 14209 | DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN) |
| 88989 | SimpleChIP® Universal qPCR Master Mix |
| 56795 | DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 |
| 47538 | Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) |
| 29580 | Multiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) |
| 66362 | Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) |
| 93569 | Concanavalin A Magnetic Beads and Activation Buffer |
| 16359 | Digitonin Solution |
| 27287 | 100X Spermidine |
| 7012 | Protease Inhibitor Cocktail (200X) |
| 7013 | RNAse A (10 mg/ml) |
| 10012 | Proteinase K (20 mg/ml) |
| 31415 | CUT&RUN 10X Wash Buffer |
| 15338 | CUT&RUN Antibody Binding Buffer |
| 48105 | CUT&RUN 4X Stop Buffer |
| 42015 | CUT&RUN DNA Extraction Buffer |