次世代シーケンシング (NG-seq) は、クロマチン免疫沈降 (ChIP) やCUT&RUN (Cleavage Under Targets and Release Using Nuclease) アッセイのデータを利用し、領域の特定やゲノム全体における標的DNAの定量などのハイスループット解析に用いられます。Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) には、Illumina platform (Illumina, Inc.) でNG-seq解析を行うための、マルチプレックスサンプルの調製に最適なアダプターとプライマーが含まれています。このキットは、最大96種類のバーコード標識されたChIP-seqまたはCUT&RUN DNAライブラリー調製に使用することができ、これらのライブラリーをまとめて1回のシーケンシング反応で解析することができます。
各キットの構成品は、厳密な品質管理基準を満たしています。新しいロットは、キット全体を用いてIlluminaシーケンシングプラットフォームでのインデックスライブラリーの構築およびシーケンシングにより検証されています。
この製品には、最大96回分のDNA配列ライブラリーを調製するための試薬が含まれます。また、この製品はDNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795と組み合わせて使用する必要があります。
適合するアッセイキット:
- CUT&RUN Assay Kit #86652
- SimpleChIP® Enzymatic Chromatin IP Kit (Magnetic Beads) #9003
- SimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005
- SimpleChIP® Plus Sonication Chromatin IP Kit (Magnetic Beads) #56383
適合しないアッセイキット:
注意: アガロースビーズはソニケーションされたサケ精子DNAでブロッキングされており、DNAライブラリーやNG-seqに混入します。
- SimpleChIP® Enzymatic Chromatin IP Kit (Agarose Beads) #9002
- SimpleChIP® Plus Enzymatic Chromatin IP Kit (Agarose Beads) #9004
DNAライブラリーの調製に必要なその他の製品:
- DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795
必要な試薬:
キットに含まれている試薬:
- • (赤) Adaptor for Illumina #42436
- • (赤) USER Enzyme #59713
- º (白) Index 501 Primer for Illumina #86676
- º (白) Index 502 Primer for Illumina #92596
- º (白) Index 503 Primer for Illumina #29576
- º (白) Index 504 Primer for Illumina #46325
- º (白) Index 505 Primer for Illumina #67343
- º (白) Index 506 Primer for Illumina #89663
- º (白) Index 507 Primer for Illumina #27649
- º (白) Index 508 Primer for Illumina #40566
- • (オレンジ) Index 701 Primer for Illumina #60797
- • (オレンジ) Index 702 Primer for Illumina #79999
- • (オレンジ) Index 703 Primer for Illumina #18697
- • (オレンジ) Index 704 Primer for Illumina #26125
- • (オレンジ) Index 705 Primer for Illumina #39467
- • (オレンジ) Index 706 Primer for Illumina #51808
- • (オレンジ) Index 707 Primer for Illumina #58724
- • (オレンジ) Index 708 Primer for Illumina #65787
- • (オレンジ) Index 709 Primer for Illumina #75272
- • (オレンジ) Index 710 Primer for Illumina #99422
- • (オレンジ) Index 711 Primer for Illumina #10812
- • (オレンジ) Index 712 Primer for Illumina #38569
- Index Primer Orange Tube Caps #54631
- Index Primer White Tube Caps #70942
キットに含まれない試薬:
- ChIPまたはCUT&RUN Illuminaライブラリー調製に適した酵素とバッファー:DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795に含まれています
- Nuclease-free Water #12931
- AMPure XP Beads (Beckman Coulter, Inc. #A63881) またはSPRIselect Reagent Kit (Beckman Coulter, Inc. #B23317)
- 用時調製した80%エタノール
- 1X TE (10 mM Tris-HCl pH 8.0、1 mM EDTA)
- 10 mM Tris-HCl (pH 8.0-8.5)
- Magnetic Separation Rack
- BioanalyzerとAgilent High Sensitivity DNA Kit (Agilent Technologies, Inc.) 、PCRチューブとPCR
Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) のプロトコール
I. ロープレックスでサンプルをプールする場合のガイドライン:
デュアルインデックスプライマー方式では、それぞれに8塩基のインデックス配列をもつ、2種類のプライマーを使用します。Index 7 primerは、P7シーケンスに隣接するインデックスを含み、一方、Index 5 primerは、P5シーケンスに隣接するインデックスを含みます。デュアルインデックスでは、シーケンスサンプルの両末端に固有のインデックスを付加します。12種類のIndex 7 primerと、8種類のIndex 5 primerとを組み合わせることで、最大96種類の異なるサンプルを固有にインデックス化することができます。
Illumina NG-seqプラットフォームでは、A/Cのシーケンスには赤色レーザー/LEDを、G/Tのシーケンスには緑色レーザー/LEDを使用しています。各サイクルにつき、赤と緑の両方のチャンネルで読み取り、適切な画像のレジストレーションを行う必要があります (すなわち、各サイクルでAかCが存在し、各サイクルでGかTが存在する必要があります)。インデックスリードのシーケンシングの際、この色のバランスが保たれていないと、失敗の原因となります。各インデックスのシーケンスをチェックして、各サイクルに対して赤と緑両方のチャネルでシグナルを持つインデックスが使用されているかどうかを確認してください。以下の例をご参照ください:
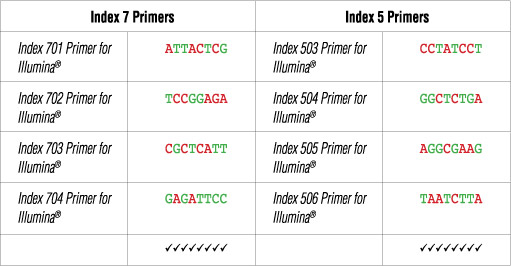

下の表は、一緒にシーケンスすることができる、有効なインデックスの組み合わせの一部 (すべてではありません) を、リスト化したものです。

その他の有効な組み合わせを下の表にまとめています。Index 7 primnerと有効なセットと、Index 5 primerの有効なセットを選択してください。Index 5 primerとIndex 7 primerを組み合わせて、必要とするライブラリー数に応じて、PCRに必要なプライマーセットを選定してください。

II. Index 5 Primers for Illumina:
各Index 5 Primer for Illuminaは、60 µLの容量で提供されています。

-s-はホスホロチオエート結合を示しています。
III. Index 7 Primers for Illumina:
各Index 7 Primer for Illuminaは 40 µLの容量で提供されています。
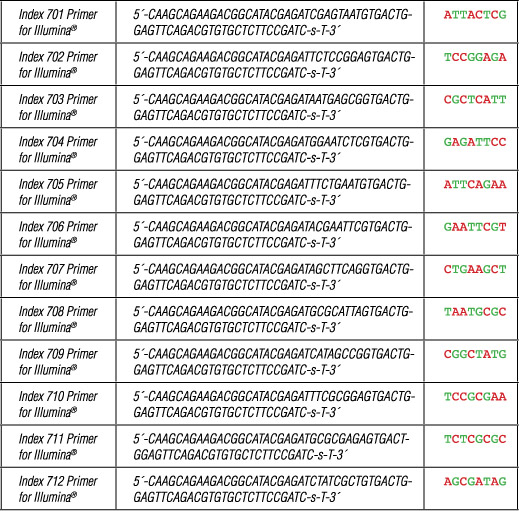
-s-はホスホロチオエート結合を示しています。
IV. PCR反応の設定
- Index 7 primerとIndex 5 primerの有効な組み合わせが成されていることを確認してください。正しいプライマー選択が行われていることの確認はセクションIとIIをご参照ください。
- 各PCRチューブには、Index 5 primer (º) (5 µL) とIndex 7 primer (•) (5 µL) を、それぞれ1種づつのみ加えてください。クロスコンタミネーションを避けるために、チューブ間で異なるチップを使用することが重要です。必要に応じて、元のインデックス5の白キャップ、またはインデックス7のオレンジキャップを破棄して、クロスコンタミネーションを避けるために新しいキャップを使用してください。
- 各PCRチューブに加えたIndex 5 primerとIndex 7 primerを記録してください。
- Q5 PCR Master Mix (•) 25 µLをプライマーを含む各チューブに加えてください。
- アダプターを連結したDNA 15 µLを、最終容量50 µLになるように対応するチューブに加えてください。ピペッティングにより、5–10回穏やかに上下させて、混ぜ合わせてください。クロスコンタミネーションを避けるために、サンプル間でチップ交換することが重要です。
- PCRチューブに、加えたDNAサンプル (アダプターを連結したDNA) を記録しておいてください。
- スピンダウンし、推奨のサイクル条件に従ってPCRを実行してください (ChIP-DNAまたはCUT&RUNに用いるDNAの初期サンプル量については、DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795のプロトコールを参照してください)。
参考:キット構成品の品質管理
Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538の構成品は、以下に記載された機能テストによって検証されており、厳格な品質管理基準を満たしています。さらに、インデックス化されたライブラリーの調製を行い、Illuminaシーケンシングプラットフォームで シーケンシング解析を行うことで、構成品の各セットが機能することを確認しています。
I. Adaptor for Illumina (15 µM) (•)
5´-/5Phos/GAT CGG AAG AGC ACA CGT CTG AAC TCC AGT C/ideoxyU/A CAC TCT TTC CCT ACA CGA CGC TCT TCC GAT C-s-T-3´
品質管理アッセイ
- 16時間のインキュベーション:アダプターとHindIII消化したLambda DNA 1 μgを含む反応液50 μLを、37℃で16時間インキュベートした場合に、アガロースゲル電気泳動で検出可能な非特異的ヌクレアーゼ分解はみられません。1X 濃度の反応バッファーとT3 DNA 1 μgを含む反応液50 μLを37℃で16時間インキュベートした場合も同様に、アガロースゲル電気泳動で検出可能な非特異的ヌクレアーゼ分解はみられません。
- エンドヌクレアーゼ活性:本アダプター最低5 µLとφX174 RF 1 DNA 1 µgを混合し、37℃で4時間インキュベートした場合 (反応液50 µL)、アガロースゲル電気泳動で検出可能なRF II (ニック分子) への変換は10%未満です。
- ホスファターゼ活性:本アダプター最低10 µLと、2.5 mM p-Nitrophenyl phosphateを混合し、タンパク質ホスファターゼアッセイバッファー (1 M Diethanolamin (pH 9.8)、0.5 mM MgCl2) 中でインキュベート (37℃、4時間) した場合、分光分析法 (405 nm) で検出可能なp-Nitrophenylene anionの産生はみられません。
- RNase活性:アダプターとFAMラベルしたRNA転写物40 ngを混合しインキュベート (37℃、16時間) した場合、ポリアクリルアミド電気泳動で検出可能なRNase活性はみられません。
II. USER Enyzme (•)
溶液組成:50 mM KCl、5 mM NaCl、10 mM Tris-HCl (pH 7.4 @ 25°C)、0.1 mM EDTA、1 mM DTT、175 µg/mL BSA、50% Glycerol
品質管理アッセイ
- 非特異的DNase活性 (16時間):Uracil DNA Glycosylaseを最低50ユニットとLambda DNA 1 µgを混合し、 50 µLのNEBuffer 1中でインキュベート (37℃、16時間) した場合、アガロースゲル電気泳動で検出可能なヌクレアーゼ活性はみられません。また、Endonuclease VIIIを最低25ユニットと、Lambda-HindIII DNA 1 µgを混合し、Endonuclease VIII反応バッファー50 µL中でインキュベート (37℃、16時間) した場合、アガロースゲル電気泳動で検出可能なヌクレアーゼ活性はみられません。
- エキソヌクレアーゼ活性 (放射線の放出):Uracil DNA Glycosylaseを最低50ユニットと [3H] ラベルした単鎖および二本鎖E. coli DNAの混合物 1 µgを混合し、 50 µLのNEBuffer 1中でインキュベート (37℃、4時間) した場合に放出される放射線は、全放射線の0.1%未満です。また、Endonuclease VIIIを最低10ユニットと[3H] ラベルした単鎖および二重鎖E. coli DNAの混合物 1 µgを混合し、50µLのEndonuclease VIII反応バッファー中でインキュベート (4℃、37時間) した場合、放出される放射線は、全放射線の0.5%未満です。
- エンドヌクレアーゼ活性 (ニッキング):Uracil DNA Glycosylaseを最低50ユニットとスーパーコイルφX174 DNA 1 µg を混合し、50µLのUDG反応バッファー中でインキュベート (37℃、4時間) した場合、アガロースゲル電気泳動で検出可能なニック型への変換は 10%未満です。
- ホスファターゼ活性:1X 濃度のUSER最低10 µLと2.5 mM p-Nitrophenyl phosphateを混合し、タンパク質ホスファターゼアッセイバッファー (1 M Diethanolamine (pH 9.8)、 0.5 mM MgCl2) 中でインキュベート (37℃、4時間) した場合、分光分析法 (405 nm) で検出可能なp-Nitrophenylene anionの産生はみられません。
III. Index 5 および Index 7 Primers for Illumina (10 µM) (º•)
品質管理アッセイ
- 16時間のインキュベーション:Index [X] Primer for Illumina1 μLとHindIII消化したLambda DNA 1 μgを含む反応液50 μLを、37℃で16時間インキュベートした場合、アガロースゲル電気泳動で検出可能な非特異的ヌクレアーゼ分解はみられません。Index [X] Primer for IlluminaとT3 DNA 1 μgを含む反応液50 μLを、37℃で16時間インキュベートした場合も同様に、アガロースゲル電気泳動で検出可能な非特異的ヌクレアーゼ分解はみられません。
- エンドヌクレアーゼ活性:Index [X] Primer for Illumina 1 μLとφX174 RF IスーパーコイルDNA 1 μgを混合し、37℃で4時間インキュベートした場合 (反応液50 μL)、アガロースゲル電気泳動で検出されるRF II (ニック分子) への変換は10%未満です。
- RNase活性:Index [X] Primer for Illumina 1 μLとRNA転写物40 ngを混合し、37℃で16時間インキュベートした場合 (反応液10 μL)、ゲル電気泳動で検出可能なRNA分解はみられません。
- ホスファターゼ活性:Index [X] Primer for Illuminaと2.5 mM p-Nitrophenyl phosphateを混合し、タンパク質ホスファターゼアッセイバッファー (1 M Diethanolamine (pH 9.8)と0.5 mM MgCl2) 中でインキュベート(37℃、4時間) した場合、分光分析法 (405 nm) で検出可能なp-Nitrophenylene anionの産生はみられません。






