View in English?
View in English?
抗体検証のための直交的戦略では、抗体ベースの結果を抗体ベースではない方法を用いて得られたデータに相互参照します。この方法は、既存の抗体検証データを確認し、その抗体に直接関連する効果やアーティファクトを特定するのに非常に重要です。ここで記述された他の戦略によって生成された結果を裏付ける、さらに高いレベルの詳細を提供する際に、直交的検証は、しばしばパブリックドメインにて利用可能なデータを使って行われます。
標的となる抗原によって、非抗体ベースの方法には、以前に発表された結果のマイニング、「オミクス」技術 (ゲノミクス、トランスクリプトミクス、プロテオミクス) による発現解析、そしてin situハイブリダイゼーションやRNA-seqなどのその他の確立された抗体非依存的な方法の採用、などが含まれます。それに応じて、直交的戦略は社内で行われた抗体検証すべてが、所期の標的に最も関連のある生物学的モデルを使用して行われることを確実にするためにも使うことができます。
最も簡易な形態の直交的戦略は、他の保証方法で得られた結果には非抗体ベースの検出方法による裏付け証拠が必要であるということを決定します。ただ1つの例として、バイナリーあるいはレンジ戦略で観察された標的のポジティブまたはネガティブ発現は、ノックアウトを確認するための遺伝子シーケンシング、あるいは発現を確認するためのmRNAのトランスクリプトミック解析などの直交的戦略を用いて確認することが必要です。
ここに記述した他の戦略と同様に、1つの情報源または個々の結果のみに依存するのはよい考えとは言えません。標的によっては、検証戦略のガイドとなる大量の発現や生物学的データ (相反するものもありますが) が利用できます。そうでない標的の場合は、抗体試験を裏付けるため適切な実験を社内で行う必要があるかもしれません。複数の信頼できるデータ源がある場合は、もちろん時間とリソースの節約になりますが、効果的な直交的検証を行うにはさらなる努力が必要です。
ウェスタンブロッティングや免疫組織化学染色など確立されている免疫染色の技術は、抗体の特異性を迅速に視覚化しますが、このような方法で生成された抗体検証データはすべて、直交的試験で裏付けをとることが非常に重要です。これを行う1つの方法としては、観察される免疫染色の結果が関連性のあるものかどうか、あるいは抗体に関連するアーチファクトによるものなのかを判断する助けとして、公表されているデータベース (CCLE、BioGPS、Human Protein Atlas、DepMap Portal、COSMICなど) からゲノムおよびトランスクリプトームのプロファイル情報を探し出すことがあります。
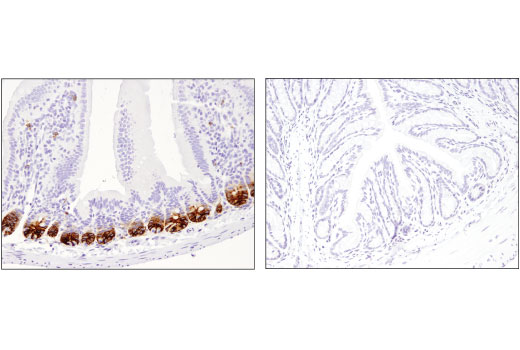
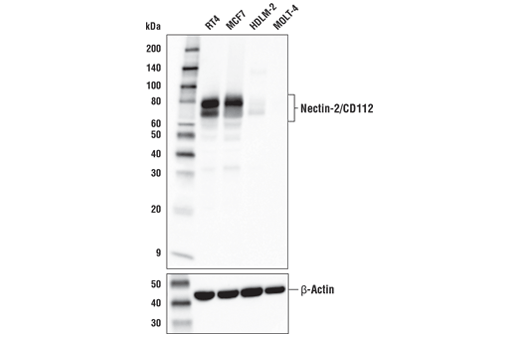
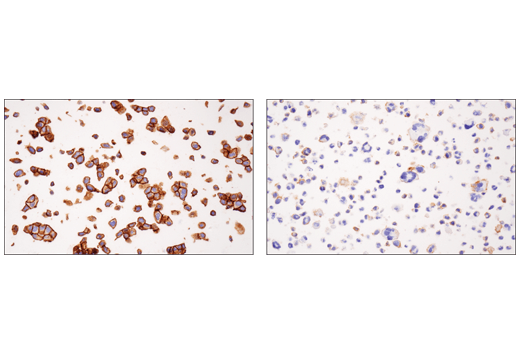
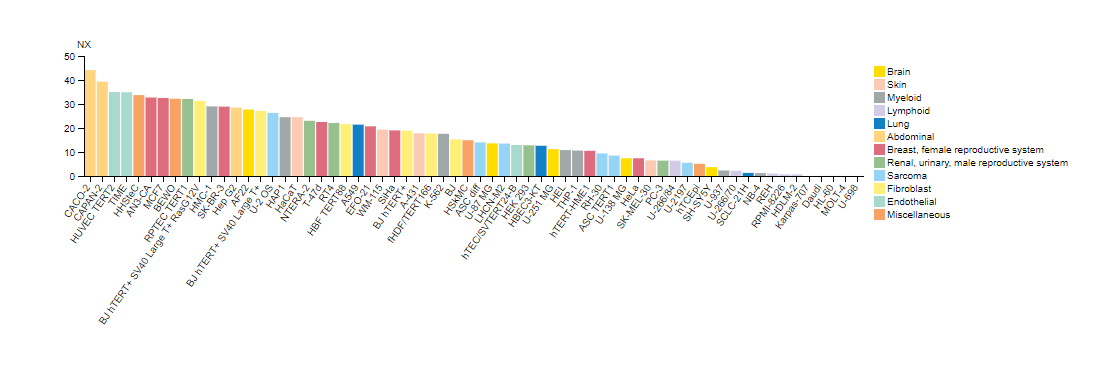
1つの例として、図1はNectin-2/ CD112 (D8D3F) rabbit mAbを用いた、多様な細胞株内のNectin-2のウェスタンブロッティングによる検出を示しています。RT4とMCF7では発現の上昇が目立っているのに対し、HDLM-2とMOLT-4での発現は最小限です。これらの結果は、RT4およびHDLM-2細胞ペレットの免疫組織化学染色による解析とよく似ており (図2)、Nectin-2の予測される発現は、Figure 3に示すように、ゲノミクスとトランスクリプトミクスのデータに基づいており、2つの異なるアプリケーションからのデータが非常によく相関していることを示しています。
様々なオミクス技術を補完するin situハイブリダイゼーション、RNA-seq、RNAscopeなどの直交的手法により、組織におけるタンパク質の発現や局在を検出できます。これらの方法は、免疫細胞化学染色または免疫組織化学染色などのイメージング技術を用いて生成された抗体データを検証するのに特に有用です。
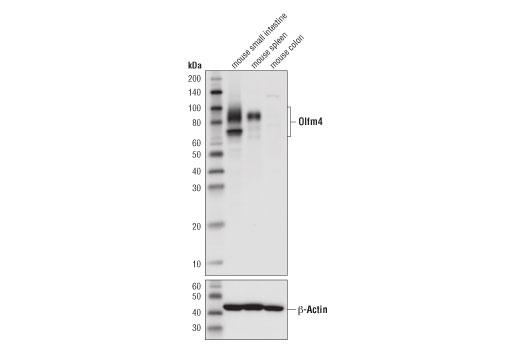
図4は、Olfm4 (D6Y5A) rabbit mAbを用いたマウスOlfm4の免疫組織化学染色による解析を示しています。ここではマウス小腸のポジティブ染色と大腸のネガティブ染色を示しており、この結果はウェスタンブロッティングで裏付けられています。(図5)。観察される染色パターンは、in situハイブリダイゼーションなど多数の公表済みの直交的戦略と一致しています。1,2
直交的戦略の成功の最も明確な基準は、遺伝子や標的タンパク質の既知あるいは予測される生物学的な役割と局在、およびその結果みられる抗体染色の間の一貫性です。これは、下流の実験で使われるモデルやアプリケーション内のすべての試薬の特異性と機能性を確認することの重要性を示しています。
ここに記述した他の保証方法と同様に、1つの検証戦略がそれだけで十分ということはありません。直交的戦略は、抗体の挙動が予測通りであるというエビデンスを提供するものですが、直交的戦略を他の方法と組み合わせ、抗体の性能に対する信頼を確実に得ることが重要です。