View in English?
View in English?
クロマチン免疫沈降 (ChIP) は抗体ベースの技術で、特定のDNA結合タンパク質とそれに結合するDNA断片を選択的に濃縮します。ChIPは特定のタンパク質とDNAの相互作用、複数のタンパク質とDNAの相互作用、タンパク質とゲノム全体あるいは遺伝子サブセットの横断的な相互作用の解析に利用されます。
ChIPでは、ヒストン、ヒストン修飾、転写因子、コファクターなどに選択的に結合する抗体を用い、クロマチンの状態や遺伝子の発現に関する情報を得ます。プロテオミクス解析や分子生物学的手法をChIPと組み合せて用いることで、解析対象の細胞や組織における遺伝子の発現や制御を解析することができます。
ChIPは通常、ゲノムの特定の領域上の、特定のタンパク質や特定のタンパク質修飾の相対量を特定するために用います。ChIPによってタンパク質とクロマチンの相互作用に関する多くの科学的な疑問に答えを得ることができます。例えばChIPを用いることで、特定のタンパク質の様々な遺伝子座上の存在量を比較することができます。これによって様々なタンパク質を解析対象の遺伝子領域上にマッピングしたり、刺激に応答する遺伝子上に結合するタンパク質を経時的に定量することができます。
ChIPの原理は比較的単純で、抗体を用いて特定のタンパク質 (ヒストン、転写因子、コファクター) とそれに結合するクロマチンを、細胞や組織の抽出物から単離 (沈降) します。これがクロマチン免疫沈降 (Chromatin ImmunoPrecipitation)という名前の由来です。免疫濃縮されたDNA断片は、広く普及しているPCRやqPCR試薬、次世代シーケンシング (NGS) 技術を利用してChIP-PCRやChIP-seqを行い、特定することや定量解析することができます。
ChIP技術には次の2つのタイプがあります:1) ネイティブChIP (N-ChIP)、2) クロスリンクChIP (X-ChIP)。これらは実験目的や実験の出発材料によって使い分けられます。どちらのタイプのChIPにも利点と欠点があります。
クロマチン免疫沈降そのものを終えた後、精製したクロマチンやその結合タンパク質 (ヒストン、転写因子、コファクター) を解析する方法はいくつかあります。単一遺伝子解析で最もよく利用される方法はqPCRで、ゲノム全体の解析の場合はChIP-seqがよく利用されます。PCRやChIP-chipを選択することもできます。
ChIP-PCRは、ゲノム上の既知の標的遺伝子座のサブセットのヒストン修飾やタンパク質の結合を解析するために行います。ChIP-PCRでは、広く普及しているPCRやqPCRの試薬や技術を用いて免疫濃縮されたDNA断片の特定と定量を行います。ChIP-PCRでは、ゲノムの特定領域について複数のサンプルの定量比較を速やかに行うことができます。これは全ゲノムのシーケンシングを行う手法より安価で時間効率の良い手法です。
ChIPで免疫濃縮したDNA断片の解析にマイクロアレイを利用する手法をChIP-chipと呼びます。ゲノムタイリングアレイ技術を用いることで、濃縮されたDNAに結合するタンパク質の全ゲノム解析を行い、タンパク質結合やタンパク質修飾の高解像度ゲノムマップを作成することができます。ChIP-chipは基礎研究から臨床研究まで多くの用途があります。例えば転写因子、エンハンサー、リプレッサーの結合部位を特定し、この種のタンパク質の結合を患者とコントロールで比較することができます。しかし、NGSのコストが大幅に低下し、ChIP-seqを用いることで同様の結果が得られるため、現在はChip-chipの代わりにChIP-seqを選択する人が増えています。
ChIP-chipと同様に、ChIP-seqによりゲノムワイドのタンパク質結合の情報が得られます。しかし、ChIP-chipと異なり、NGS技術を用いてDNA断片を特定し、全ゲノムに対してこれらをマッピングします。
DNA増幅技術の進歩により少量のDNAから安定した解析を数日で行うことができます。また、ライブラリー調製法の技術進歩により微量な出発物質からもChIP-seq解析が可能になりました。
さらに、DNAサンプルをユニークな短い配列 (バーコード配列) で標識する新技術を用いて、個々のサンプルを1つにまとめて同時にシーケンシングするマルチプレックス解析が可能になりました。これによりDNAシーケンシング実験の大幅な効率化とコストの低下が実現し、ChIP-seq実験はより身近なものとなっています。
つまり、ChIP-seqの利点は多くのChIP濃縮DNAサンプルをシーケンシングできることで、DNAシーケンシング技術の進歩によりChIP-chipより安価で時間効率良く、高感度で精密な結果が得られるようになりました。
ChIPアッセイは一般に次のような手順で行います。
各ステップが成功していることを評価するための、ポジティブコントロールとネガティブコントロールが重要です。
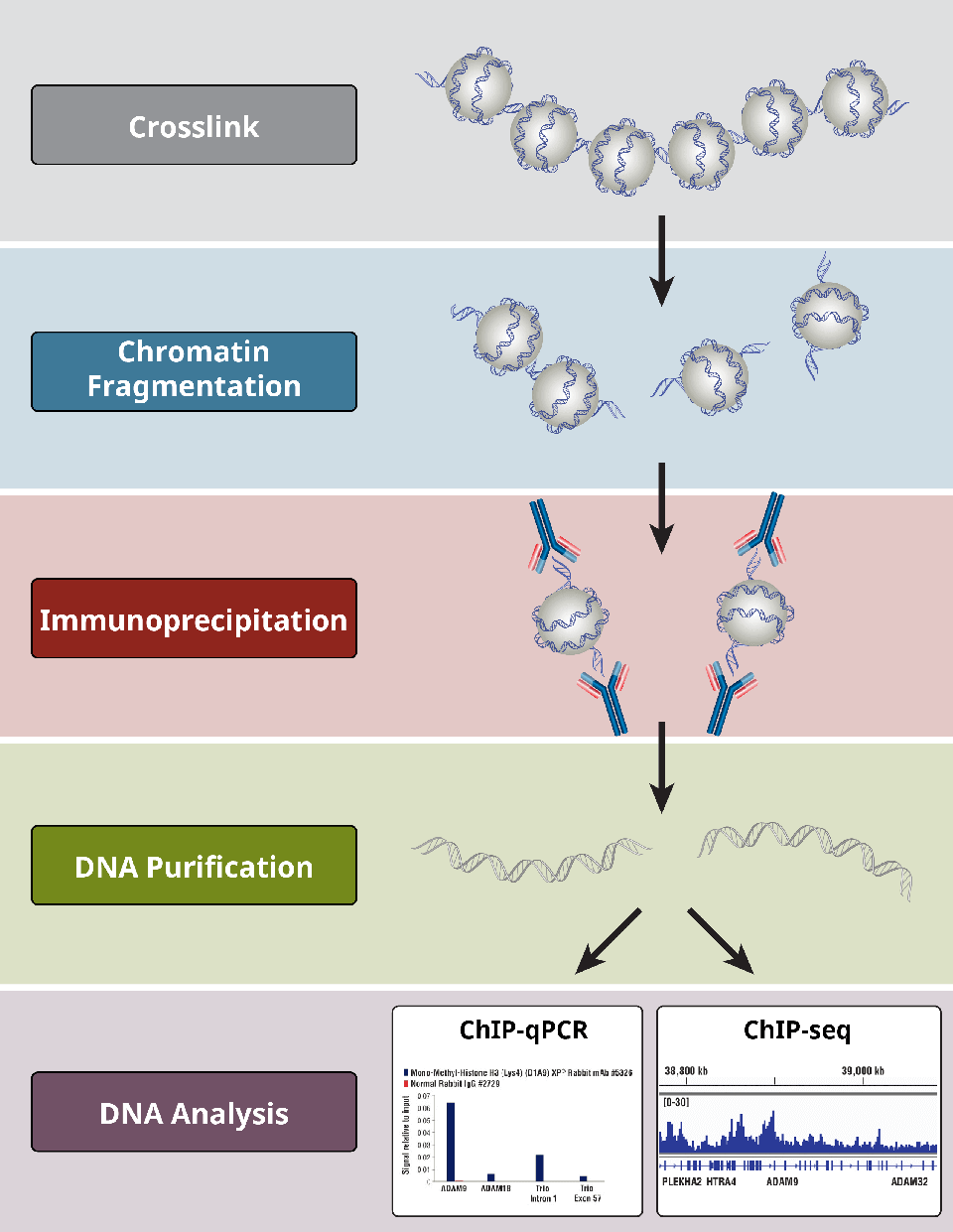
ChIPプロトコールの最も重要なステップの概要
クロスリンク試薬はタンパク質を結合したDNAに「固定」するために用います。この固定には多くの場合、ホルムアルデヒドベースの試薬を用います。細胞と組織は一般に同様の方法で固定されますが、組織の場合はより長時間の固定が必要で、また組織の劣化が始まる前に速やかに固定試薬を浸透させる必要があります。
過度のクロマチンの固定はソニケーションによる断片化の効率を低下させ、また、抗体の標的タンパク質への結合が低下する原因にもなります。したがって、抗体と抗原の結合を最大限に保持しつつ、タンパク質と標的DNAを理想的にクロスリンクする固定時間を経験的に決定する必要があります。
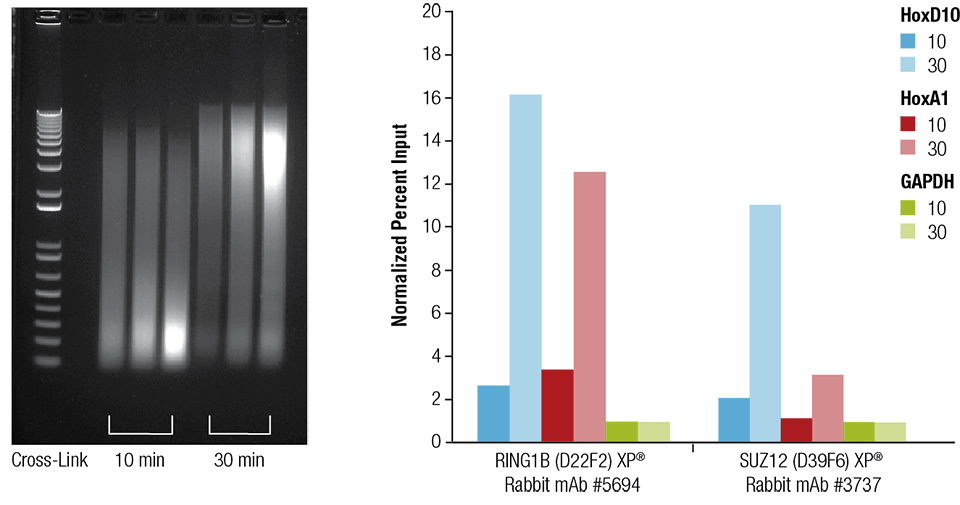
図中に示したように、マウスの心臓 (H)、脳 (B)、肝臓 (L) を10分間または30分間クロスリンクしました (左パネル)。クロマチンサンプルを調製し、4分間ソニケーションを行いました。心臓組織由来のクロマチンと図中し示した抗体を用いて、SimpleChIP® Plus Sonication Chromatin IP Kit #56383でChIPを行いました。図中に示した遺伝子のプライマーセットでリアルタイムPCRを行い、濃縮されたDNAを定量しました。各サンプルの免疫沈降で濃縮されたDNAは、GAPDH遺伝子座 (ネガティブ遺伝子座) のシグナルを「1」とした場合の相対値で示しました。
クロマチンの断片化はChIP実験の成功に必要不可欠です。クロマチンの可溶化にクロマチンの断片化が必要で、これによって免疫沈降で共沈させることが可能になります。また、DNA断片のサイズがChIPアッセイの解像度を左右するため、DNAの断片化がChIPアッセイの解像度を規定すると言えます。
酵素消化にはMNase (micrococcal nuclease) を用います。この酵素はヌクレオソームの間の2本鎖DNAを切断してクロマチンを断片化します。MNaseで完全消化した場合には150 bp (モノ-ヌクレオソーム) のDNA断片が生じますが、不完全消化によって150-750 bp (モノ-ヌクレオソーム、ジ-ヌクレオソーム、トリ-ヌクレオソーム) のDNA断片が生じます。ソニケーションは力学的な力でクロマチンを断片化します。ソニケーションではヌクレオソームの間やヌクレオソーム内が切断され、150-1000 bpの様々なクロマチン断片が生じます。
X-ChIPの場合は酵素消化かソニケーションでクロマチンを断片化します。ソニケーションChIPプロトコールでは、最適なソニケーション条件が細胞のタイプや実験条件によって異なるため、経験的に決定する必要があります。酵素消化の条件は異なる細胞や組織で比較的一貫していますが、IPの前にクロマチン断片のサイズを確認しておく必要があります。
N-ChIPの場合は非固定サンプルのタンパク質結合を保持するため、ヌクレアーゼを用いてクロマチンを断片化します。ヌクレアーゼによる断片化も、過剰な消化を最小限に抑えるように経験的に最適条件を決定する必要があります。
N-ChIPの場合、タンパク質はDNAにクロスリンクされておらず、ソニケーションによる過酷な条件に晒されることでクロマチンタンパク質がDNAから解離することがあるため、ヌクレアーゼ消化を用います。ヒストンとDNAの結合は非常に強く安定であるため、N-ChIPはヒストンとDNAの相互作用の解析に理想的です。しかし、N-ChIPは転写因子やコファクターのクロマチン結合の解析にはうまく機能しません。
X-ChIPの場合は酵素消化とソニケーションのどちらを用いることもできます。酵素消化の利点には、安定した断片化ができることや、断片化条件が穏やか (加熱や界面活性剤) であることがあります。このためクロマチンや抗体エピトープの完全性が保持され易く、転写因子やコファクターの免疫濃縮が増加します。
酵素消化によるクロマチンの断片化と異なり、ソニケーションは力学的な力に依存じてクロマチンをより小さな断片に断片化します。免疫濃縮に理想的なクロマチン断片のサイズは200-1000 bpです。ソニケーションはクロマチンを断片化するための従来法で、従来のプローブソニケーターを用いることもできますが、より進歩したウォーターバスソニケーターを用いてより集中的にソニケーションを行うこともできます。ソニケーションでは完全にランダムなクロマチン断片が得られます。しかし、異なる細胞株や組織の間で大掛かりな最適化が必要で、実験ごとに再現することが容易ではありません。強い界面活性剤バッファーやソニケーション中の発熱によって、クロマチンやクロマチンタンパク質の抗体エピトープの完全性が損なわれる可能性もあります。
ソニケーションによるクロマチンの断片化は、従来的に強い界面活性剤バッファーを用い、熱を発生します。これらはどちらもクロマチンや抗体エピトープの完全性を損なう原因になります。したがって、クロマチンを断片化するソニケーションの時間を、細胞株や組織ごとに経験的に決定する必要があります。クロマチンの損傷が最小限に抑えられるように、150-1000 bpのDNA断片が得られる最短のソニケーション時間を決定し、この条件で実験を行う必要があります。
下流のqPCR、DNAチップ、NGSを用いた解析を含む完全なChIPアッセイに着手する前に、ソニケーションを様々な時間で行ったクロマチンサンプルをゲル電気泳動で解析してください。断片のサイズはソニケーション時間に依存し、ソニケーション時間が長いほど断片のサイズは小さくなります。しかし、長時間のソニケーションが必ずしも良好な結果に繋がらないことがデータから示されています。したがって、免疫沈降したDNAを精製し、電気泳動で理想的な断片サイズを決定することが、目的のDNAサイズを得るために必要な最小のソニケーション時間を決定し、クロマチンの無用な損傷を避けるための簡単な方法です。

酵素消化したクロマチンをアガロースゲルで電気泳動しました。レーン1は、消化が不十分なクロマチンを示しています。レーン2は、適切に消化されたクロマチンを示し、レーン3は過剰に消化されたクロマチンを示しています。
ChIP実験を成功させるには、適切な抗体を選択することが大変重要です。ChIPに用いる抗体は標的タンパク質に特異的で、抗原に高い親和性をもつ必要があります。ChIPまたはChIP-seqに用いる最適な抗体は、ChIPまたはChIP-seq検証済み抗体です。解析対象のChIP検証済み抗体が見付からない場合は、IP検証済み抗体が次善の選択です。IP検証済み抗体が必ずしもChIPで機能するとは限らず、ChIP検証済み抗体が必ずしもChIP-seqで機能するとは限らないことに注意することが重要です。また、WB、IP、IF、flow、IHCなど、より多くのアプリケーションで抗体が検証されているほど、抗体の性能と特異性への信頼性が高まります。抗体の検証に引き続き、抗体の最適濃度とIPの洗浄条件を経験的に決定する必要があります。
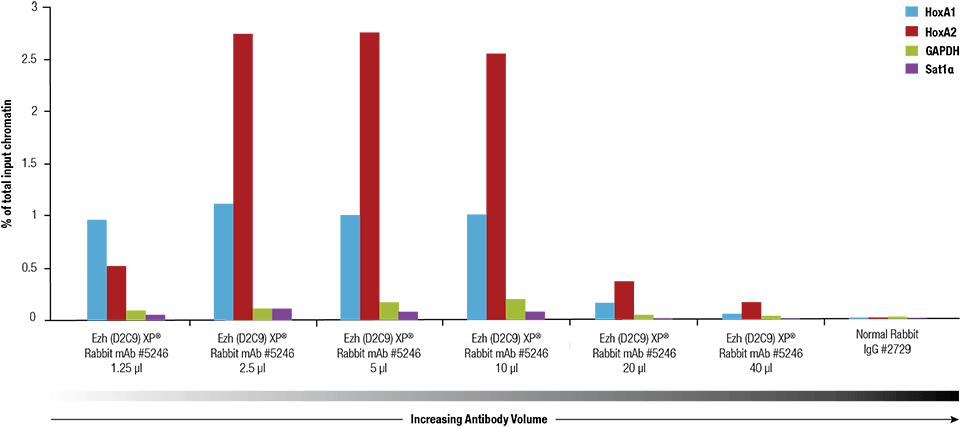
SimpleChIP®Plus Enzymatic Chromatin IP Kit #9005 を用い、4x106の NCCIT細胞から調製したクロスリンククロマチンで Ezh2 (D2C9) XP® Rabbit mAb #5246のタイトレーションを行いました。
標的タンパク質とそれに結合するDNAを捕捉するために抗体を利用します。抗体濃度は経験的に決定します。通常は10 μgのクロマチンDNA (約4 x 10 6細胞に相当) 当たり0.5-2.0 µgから始めます。バッファー条件と洗浄回数も、抗体と標的抗原の親和性に依存するため、経験的に決定する必要があります。通常、抗体とクロマチンのインキュベーションは2時間から一晩行います。
抗体と抗原 (とDNA) の複合体は抗体結合レジンに捕捉します。ChIP実験では通常、Protein AやProtein Gを結合させたChIP品質の磁気ビーズやsepharoseビーズ、アガロースビーズを用います。抗体はProtein AやProtein Gビーズに結合しますが、抗体の免疫動物種やIgG重鎖のサブタイプによって親和性は異なります。通常、ビーズと抗体-クロマチン複合体のインキュベーションは2-4時間行います。
抗体に特異的に結合していないクロマチンを洗浄ステップで除去し、脱クロスリンク (X-ChIPの場合) とDNAの精製を行います。また、バックグラウンド (S/N比) を決定するため、IgGコントロールIPを行う必要があります。非特異結合を決定するには、ポジティブコントロール抗体 (総ヒストンH3抗体など) や、ポジティブコントロールqPCRプライマー (標的タンパク質の既知陽性遺伝子座と陰性遺伝子座) も必要です。最適な結果を得るには、下流のNGS解析の前に、qPCRによるクロマチンIPのQCを行う必要があります。
クロマチンは界面活性剤を用いて加熱することでProtein A/Gビーズから溶出します。ビーズの懸濁状態を保ち、クロマチンの溶出効率を高めるため、低速の「ボルテックス」または攪拌が必要です。
高塩濃度、高温の加熱 (この両方が重要な要素です) によって脱クロスリンクを行います。プロテイナーゼKを加えてクロマチン結合タンパク質や免疫沈降に用いた抗体を消化することで、下流のDNA精製が効率化されます。
クロマチンのクロスリンクを除去した後、古典的なフェノール-クロロホルム、エタノール沈殿法か、カラムベースのDNA精製キットでDNAを精製しま
精製したDNAは、ChIP-PCR、ChIP-qPCR、ChIP-chip、ChIP-seqなど、いくつかの手法で解析することができます。
ChIP-PCRおよびChIP-qPCRは単一の遺伝子の解析に最適な手法で、特定のDNA断片を迅速かつ安価に増幅して定量することができます。
タイリングDNAマイクロアレイを用いたChIP-chip解析で、タンパク質結合やタンパク質修飾の高解像度ゲノムマップを作成することができます。
標準的なNGS技術を利用したChIP-seq解析で、精製したDNAと注釈付きの全ゲノムの配列を比較し、ゲノムワイドなタンパク質結合プロファイリングを行うことができます。