View in English?
View in English?
CSTのシグナル伝達パスウェイ図で個々のタンパク質名をクリックすると、それに関連した研究リソースや製品情報を検索することができます。さらに、教育・研究のために、パスウェイ図をダウンロードすることも可能です。
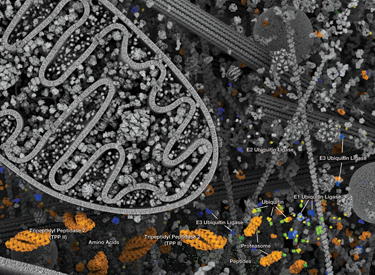
ユビキチンプロテアソーム系 (Ubiquitin-Proteosome System:UPS) は細胞タンパク質を分解する主要なプロセスで、折りたたみに異常があるタンパク質や損傷したタンパク質だけでなく、シグナル伝達経路によってその活性の急速な制御が行われるタンパク質も除去する、高度に制御されたシステムです。このシステムは細胞増殖や転写の制御、アポトーシス、免疫、および発生において中心的な役割を果たします。ユビキチンは、高度に保存された76個のアミノ酸から成るタンパク質であり、ユビキチン化プロセスを通じて、多くの細胞タンパク質と共有結合します。ユビキチン化はATP依存性のプロセスで、3種類の酵素によって実行されます。「ユビキチン活性化酵素」(E1) であるUBA1は、ユビキチンとチオエステル結合を形成します。この反応によって、それに続くユビキチンと「ユビキチン結合酵素」 (E2) の結合が可能となり、さらにユビキチンのカルボキシ末端と基質タンパク質上のε-アミノ基リジン残基との間にイソペプチド結合を形成します。後者の結合は「ユビキチンリガーゼ」 (E3) を必要とし、真核細胞には数百種類のE3リガーゼが存在し、それぞれのリガーゼが修飾できるのは、ある1種類の基質タンパク質のサブセットのみです。このように、UPSは基質特異性を有しています。ユビキチン化されたタンパク質は、その後、26Sプロテアソームの標的となって分解されるか、またはその局在や活性の変更が行われます。
ユビキチンは、モノユビキチン化によって単体で、またはポリユビキチン化によって分枝鎖として、基質と結合できます。基質タンパク質は、ユビキチンの七つのリジン残基 (Lys6、Lys11、Lys27、Lys29、Lys33、Lys48、およびLys63) によってユビキチンに結合します。ポリユビキチン鎖は、ユビキチンのリジン残基が別のユビキチンのカルボキシ末端のグリシンに結合して形成されます。ポリユビキチン化されたタンパク質は、その結合を媒介するユビキチン結合の性質次第で異なる運命を辿ります。K48結合ポリユビキチン鎖は主にプロテアソーム分解のタンパク質を標的とし、他方、K63結合ポリユビキチン鎖は通常、タンパク質機能や細胞内局在、タンパク質相互作用を制御しますが、この結合も、時にはプロテアソーム分解をもたらします。
脱ユビキチン化酵素 (DUB) は、ユビキチンをその基質タンパク質から除去することで、ユビキチン化プロセスを逆行させます。DUBの活性はユビキチンのリサイクルを維持し、細胞内におけるユビキチン分子の貯蔵量を安定させます。DUBは5つのサブファミリー、すなわちUSP、UCH、OTU、MJD、JAMMに分類され、それぞれに固有の組織と基質特異性を有しています。26Sプロテアソームは非常に巨大な ~2 MDaの複合体であり、ユビキチンプロテアソームシステムのタンパク質分解アームとして機能します。主に19S調節粒子 (Regulatory Particle: RP) と20S触媒コア粒子 (Core Particle: CP) との2種類のサブ複合体から構成されます。CPは、円柱状の触媒チャンバーを形成します。RPは、基底部と蓋を含みます。この基底部は、基質の折り畳みをほどき、ゲートを開きます。これによりほどかれた基質が触媒チャンバーに入っていきます。蓋は、主にユビキチンシグナルの特異的な認識に関与します。19S capに加えて、他のタンパク質や複合体が20S cylinderの末端に結合してゲートを開くことで活性化させます。更に、プロテアソームに結合したDUBおよびE3は、基質に固定されたポリユビキチン鎖を再構成し、それらの分解に対する感受性を調節します。
Small Ubiquitin-related modifier 1、2、および3 (SUMO-1、-2、-3) およびNEDD8は、ユビキチン様タンパク質ファミリーのメンバーです。SUMOとNEDDは、E1、E2、E3の結合システムを用いたユビキチン化と類似した方法でタンパク質に共有結合することができます。この共有結合をそれぞれSUMO化、NEDD化と呼びます。しかし、ユビキチン化とは異なり、一般的に基質タンパク質のSUMO化とNEDD化により、タンパク質分解は起こりません。その代わりに、SUMO修飾およびNEDD修飾は、細胞内局在、タンパク質機能、あるいはタンパク質間相互作用に影響を及ぼします。SUMO化は、核輸送、転写活性の制御、タンパク質の安定性など、細胞に対して数多くの作用を有しています。SUMO化と、ユビキチン化、リン酸化およびアセチル化などの翻訳後修飾間のクロストークは、活発に研究されている分野です。