View in English?
View in English?
| キナーゼ | 説明 |
|---|---|
| AGC | PKA、PKG、PKCファミリーを含む |
| CAMK | カルシウム/カルモジュリン依存性プロテインキナーゼ |
| CK1 | Casein kinase 1 |
| CMGC | CDK、MAPK、GSK3、CLKファミリーを含む |
| STE | 酵母のSterile 7、Sterile11、Sterile20キナーゼのホモログ |
| TK | チロシンキナーゼ |
| TKL | チロシンキナーゼ様 |
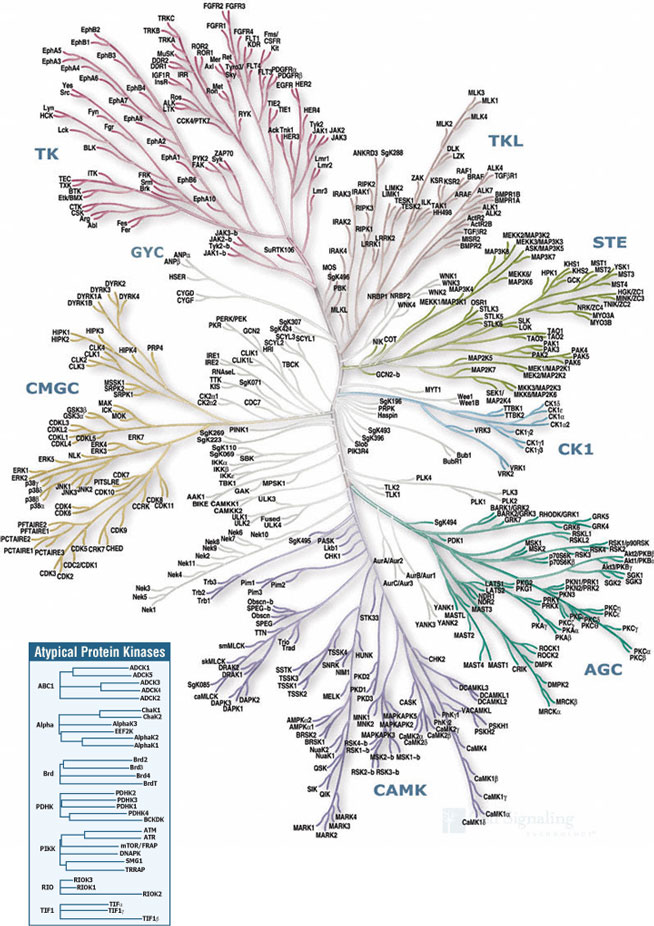
図中のキナーゼグループをクリックすると、各キナーゼグループの詳細ページに移動します。
Cell Signaling Technology (CST) のThe Human Kinomeの使用リクエストのために、リクエストフォームを記入してください。
こちらの樹状図 は、プロテインキナーゼドメイン間の配列類似性を示しています。これは公開配列および著者らが詳述した遺伝子予測法によって導き出されました。(Science, 298, 1912-1934)。ドメインは、隠れマルコフモデルのプロファイル分析や多重配列のアラインメントによって定義されました。初期の分枝パターンは、ドメインのclustalWタンパク質配列アラインメントから導き出された近隣結合樹から構築されました。そして他のアラインメント法や樹状図構築法 (hmmalignやparsimony trees)、そしてキナーゼドメインの広範なペアシーケンスアラインメントを参照して、広範囲に渡りこれが修正されました。曲線型レイアウトは手書きでおこなわれました。多くの枝長は完全に定量的ではありませんが、この枝分かれパターンは単一の自動方式で描くよりも多くの情報を提供します。詳細な樹状図は、全長タンパク質配列のclustalWアライメントの後に近隣結合系統樹の構築によって自動生成されました。未発表のキナーゼは、できる限りファミリーの命名法に従って命名されます。一部の分岐キナーゼは、SgK (SuGenキナーゼ) の数字で表した登録番号をそのまま保持します。二重ドメインキナーゼの2番目のドメインは、「~b」の接尾語を付けて命名されます。