View in English?
View in English?
様々な細胞タイプ・発生過程において、それぞれに特有の、ユークロマチンおよびヘテロクロマチンの個々のドメインからなる、エピジェネティックなランドスケープがみられます。遺伝子発現は、ヒストンの翻訳後修飾を介して、エピジェネティックな制御を受けます。例えば、ヒストンH3のLys4のトリメチル化は、活性化クロマチン (ユークロマチン) にみられる修飾で、一方、ヒストンH3のLys27のトリメチル化は、不活性化ヘテロクロマチンでみられる修飾です。
ヒストン修飾の局在化領域や転写因子の結合部位を、ゲノムワイドに解析する強力な手法が、クロマチン免疫沈降 (Chromatin ImmunoPrecipitation:ChIP) 解析です。ChIPアッセイでは、ホルムアルデヒドを使用して、生細胞のタンパク質をその標的とするDNAに共有結合でクロスリンクします。その後、このタンパク質-DNA複合体を、特定のヒストン修飾または転写因子を認識する抗体を用いて免疫沈降します。免疫沈降で共沈したDNAを精製し、オリゴヌクレオチドマイクロアレイへのハイブリダイゼーション (ChIP-chip) や、ハイスループットDNAシーケンシング (ChIP-seq) で解析します。これらの手法を用いることで、生細胞内のDNA-タンパク質相互作用の「スナップショット」を解析することができ、例えば、細胞周期や発生の特定の時期や、組織から直接採取した検体を解析できます。ChIP-chipは、DNA-タンパク質相互作用のゲノムワイド解析で、はじめに用いられた手法です。しかし、ヒトゲノム全体を網羅するためには、複数のDNAマイクロアレイチップが必要で、全ゲノム解析には高いコストがかかります。一方、ChIP-seq技術は、ゲノムワイド解析のもう一つの手法で、一度のシーケンシング反応でゲノム全体に渡る解析が可能です。この方法では、Illumina GA2シーケンサーを用いて短いシーケンスリードを作成し、濃縮されたDNA断片をゲノム上へ正確にマッピングするのに必要な情報を得ることができます。
CSTのSimpleChIP® Enzymatic Chromatin IP Kit (Magnetic Beads) #9003を用いたChIP-seq実験により、K562赤白血病細胞株におけるエピジェネティックマーク、ヒストンH3 (Lys4) のトリメチル化、ヒストンH3 (Lys27) のトリメチル化を解析しました。SimpleChIP®プロトコールに従って、K562細胞からクロマチンサンプルを調製し、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733、Normal Rabbit IgG #2729 (ネガティブコントロール) を用いて免疫沈降を行いました。H3 (Lys4) のトリメチル化修飾、H3 (Lys27) のトリメチル化修飾が濃縮する、ゲノム上の既知領域について、通常のPCR解析で確認を行いました (図1)。Illumina GA2シークエンシングプラットフォームで解析するため、免疫沈降したDNAを用いてシーケンシングライブラリーを調製しました。得られたシーケンスをUCSCヒトゲノムアセンブリ (HG18) へマッピングし、ユニークにマッピングされたシーケンスを解析に用いました。結合部位の濃縮はシーケンスリード数に反映され、ピークが高くなっています (図2)。ラビットモノクローナル抗体は、ピークの高さが平均17リードと高く、かつ非常に低バックグラウンドで、優れた濃縮結果が得られていることが分かります。

図 1 K562細胞のクロマチンサンプルを、SimpleChIP® Enzymatic Chromatin IP Kit (Magnetic Beads) #9003と、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 (レーン1)、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733 (レーン2)、Normal Rabbit IgG #2729 (レーン3) を用いた免疫沈降に使用しました。共沈したDNAを精製し、転写が活性化しているDHFR遺伝子とHIST1H2AC遺伝子クラスター、または不活性化しているMYT1遺伝子それぞれに特異的なプライマーを用いて、標準的なPCR法で解析しました。
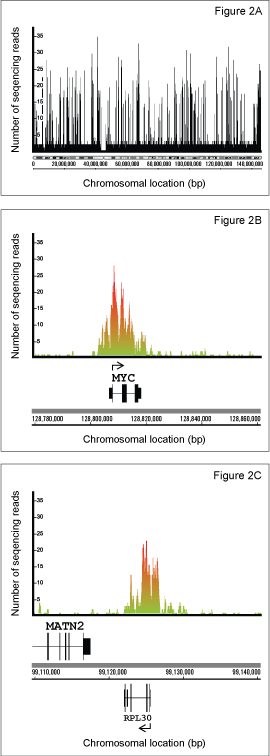
図 2 Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いてK562細胞のクロマチンサンプルを免疫沈降し、濃縮されたDNAを、Illumina GA2シーケンサーでシーケンシング解析しました。得られたシーケンスをUCSCヒトゲノムアッセンブリ (HG18) にマッピングし、IGB (Integrated Genome Browser) を用いてデータを可視化しました。(図2A) 第8染色体におけるH3 (Lys4) トリメチル化修飾の濃縮が示されています。メチル化が濃縮された部位では、シーケンシングリード数が多くなり、ピークが高くなります。c-MYC (図2B) やRPL30 (図2C) などの活性化された遺伝子において、トリメチル化修飾されたH3 (Lys4) が濃縮されていることが分かります。
CSTは、ChIPシーケンシングデータを共有していただいた、カリフォルニア州、カリフォルニア大学デイビス校のH. O'geen氏とP.J. Farnha氏に心より感謝いたします。