View in English?
View in English?
バイナリーアプローチは、抗体の特異性を評価する最良の方法の1つです。生物学的に関連するポジティブ発現系とネガティブ発現系で抗体を試験することにより、サンプルに存在するほかの生体分子と交差反応することなく、ネイティブな環境で標的抗原の認識を確認できます。バイナリーモデルには、標的タンパク質の発現がポジティブ (高い) あるいはネガティブ (低い) ことが知られているまたは予測される細胞や組織の内因性標的を利用するもの、遺伝的ノックアウトを利用するもの、標的タンパク質の発現や修飾を誘導あるいは阻害する処理を利用するものがあります。
バイナリー試験を効果的に行うためには、遺伝子シーケンシングによるノックアウトの確認やプロテオミクスプロファイリングによる発現レベルの確認など、直交的な手法を用いてデータを常に確認する必要があります。抗体の性能に最大限の信頼を持たせるため、その他の検証戦略も採用する必要があります。また、抗体のバイナリー検証に使用するモデルはそれぞれ、抗体の使用を予定しているアプリケーションごとに試験する必要があります。ある抗体がウェスタンブロットで特異的であるからと言って、同じ抗体が免疫組織化学染色 (IHC) でも同様に特異的であるとは限りません。
目的の標的を内在的に発現する細胞や組織は、最も単純な形のポジティブコントロールです。同様の材料に由来する標的の発現が無いサンプルと並べて用いることによって、抗体による抗原の特異的な認識の有無が迅速に分かります。
この戦略によって意義ある知見を確実に得るには、真のポジティブコントロールあるいはネガティブコントロールとなるサンプル材料を選択することが重要です。これは、ゲノムデータベース、トランスクリプトームデータベース、プロテオミクスデータベースを利用して大量の文献を検索することで実現できます。 あるいは、試験する抗体によって得られた免疫染色を、同じ標的の異なる抗原に対する完全に検証されている抗体の免疫染色と比較します。
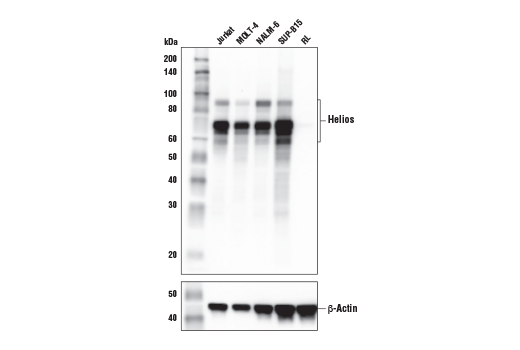
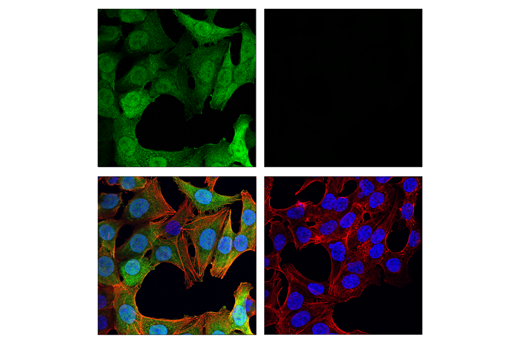
図1および2は、内因性コントロールを使用した抗体検証の例です。抗体の検証に使用するモデルは、抗体の使用を予定しているアプリケーションごとに試験する必要があることが明確に分かります。図1はJurkat細胞とRL細胞を用い、Helios (E4L5U) rabbit mAbをウェスタンブロッティングで評価した結果です。Heliosの発現はJurkat細胞でポジティブ、RL細胞でネガティブとなっています。一方、 図2は同じ細胞モデルを用いて免疫組織化学染色 (IHC) で抗体を評価した結果です。ローディングコントロールを用いてサンプルの品質に問題が無いことを確認しており、ウェスタンブロットとIHCのデータがお互いを裏付けています。

普遍的に発現している一部のタンパク質を検証する場合や、発現をサポートするデータが矛盾していたり信頼性を欠く場合など、内因性バイナリーモデルが常に利用できるとは限りません。このような場合、マウスのノックアウトモデルは抗体の特異性のバイナリー評価に広く使用されているコントロールです。通常、共同研究者から提供されるマウスのノックアウトサンプルは、野生型およびノックアウト型の細胞や組織で構成されています。これらはそれぞれ、標的抗原のネイティブな発現の有無を示しており、利用が可能であれば理想的な試験パラダイムとなります。
遺伝的ノックアウトを実現するもう1つの方法として、CRISPRを介した遺伝子破壊を利用する方法がありますが、これは近年、直感的で柔軟性の高いシステムに簡略化され、その有用性が高まってきています。このアプローチの利点は、同じ細胞株でポジティブおよびネガティブの発現モデルが得られることです。ただし、遺伝子の破壊は常に完全であるとは限らず、遺伝子の部分的な欠失や断片化が起こり、これに由来するシグナルが得られることがあるので注意が必要です。標的のノックダウンは、抗体に依存しない直交的な方法で確認することが重要です。また、細胞のCRISPRノックアウトは、細胞ベースのアッセイで抗体の性能を検証する場合は非常に有効ですが、様々なタイプの細胞が混在する、免疫組織化学染色などの組織ベースのアッセイには必ずしも有効では無いことに注意が必要です。
CRISPR以外にも、合成2本鎖RNA (siRNA [small/short interfering RNA] ) を用い、タンパク質をコードする遺伝子の発現を一過性に抑制するノックダウンも一般的な方法です。さらに別の選択肢として、DNAベクターに組み込んだshRNA (short/small hairpin RNA) をトランスフェクションやウイルス感染で細胞に導入する方法もあります。ベクターの選択マーカーを利用して、トランスフェクションまたはウイルス感染した細胞を選択することで、宿主ゲノムにshRNAが安定に組み込まれ、長期にわたって遺伝子がサイレンシングされた細胞を得ることもできます。
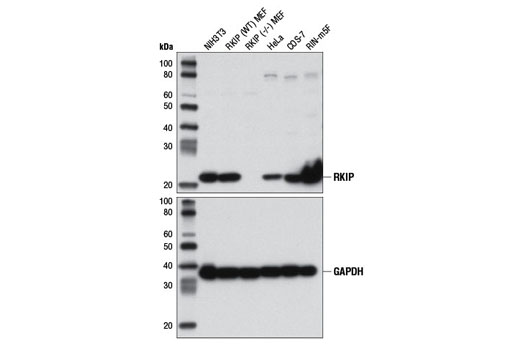
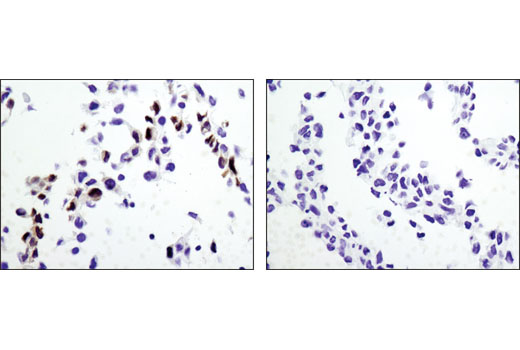
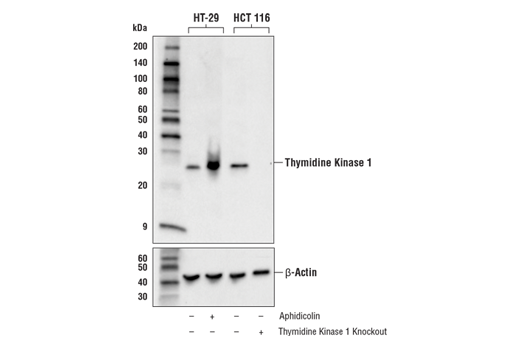
この場合も、抗体検証に使用する遺伝子ノックアウトモデルは、抗体の使用を予定しているアプリケーションごとに試験することが非常に重要です。図3および4は、野生型マウスおよびRKIPノックアウトマウスの胚性線維芽細胞 (MEF) を用い、RKIP (D42F3) Rabbit mAbをウェスタンブロッティングと免疫組織化学染色で評価したものです。図5および6は、Thymidine Kinase 1 (E2H7Z) Rabbit mAbを用い、野生型およびThymidine Kinase 1ノックアウトHCT116細胞を、ウェスタンブロッティングと免疫組織化学染色で評価したものです。どちらの場合も、2つのアプリケーションのデータがお互いを裏付けています。
バイナリーモデルの別の形として、タンパク質標的の発現、局在、翻訳後修飾 (PTM) を誘導あるいは阻害する処理 (アゴニストあるいはアンタゴニスト) の使用する方法があります。これらの処理で、ほぼ完全なポジティブのシグナルとネガティブのシグナルが得られる場合、バイナリー検証に利用することができます。標的抗原の修飾は、タンパク質のリン酸化やアセチル化など、翻訳後修飾された標的に対する抗体を検証するのに適したアプローチです。
標的抗原の修飾に依存する検証法の場合も、他の検証法と同様に、適切なローディングコントロール、発現コントロールは非常に重要です。これらによって、処理の有効性や、評価対象のサンプルが質、量ともに同等であることが確認できます。処理ができる限り標的に特異的であることも重要です。広範な標的から非特異的に翻訳後修飾を除去する一般的なホスファターゼや脱アセチル化酵素で処理を行う前に、例えば、タンパク質やパスウェイに特異的なアゴニストやインヒビターについて検討すべきです。
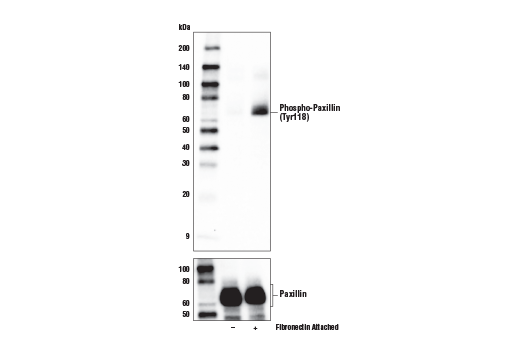
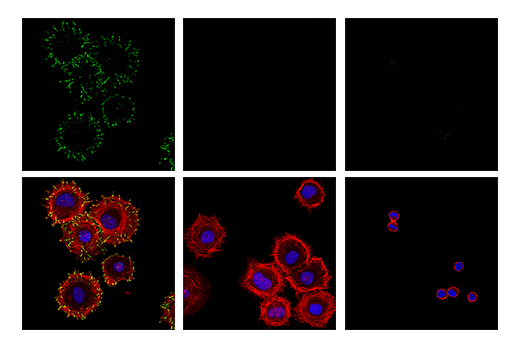
図7および8は、HeLa細胞を用い、Fibronectinでコーティングしたプレート表面への細胞接着を利用してチロシン残基のリン酸化を誘導し、免疫細胞化学染色でPhospho-Paxillin (Tyr118) 抗体の検証を行ったものです。
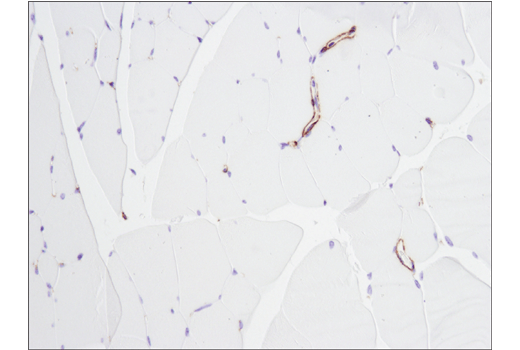
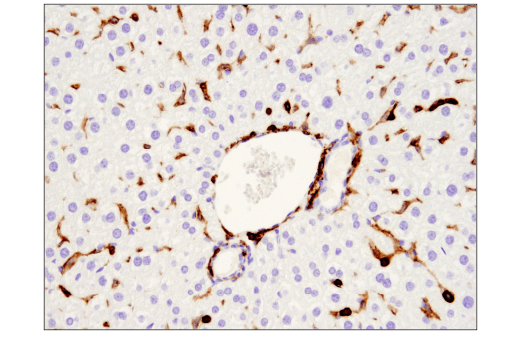
免疫組織化学染色で最も頻繁に使用されるその他のバイナリー戦略として、同じ切片にポジティブ細胞とネガティブ細胞の両方を含む組織サンプルを用いて抗体を検証する方法があります。この例を図9および10に示しました。観察された染色パターンは、標的の既知の発現プロファイルに一致しています (すなわち、発現の無い細胞は染色されず、適切な細胞が陽性染色されています)。
バイナリーモデルは抗体試薬の検証に有効な手段ですが、必ずしも材料を容易に入手できたり、速やかに準備ができる訳ではありません。バイナリー試験のデータをサポートするため、また抗体の性能を評価する代替の手段として、追加の検証方法を常に利用する必要があります。