View in English?
View in English?
クロマチンにおけるタンパク質-DNA相互作用を少ない細胞数で解析可能な、ChIP-qPCRやChIP-seqに代わるCUT&RUNをご紹介します。
クロマチン免疫沈降 (ChIP) やChIP-seqといったタンパク質とDNAの相互作用をマッピングする技術が開発されたことで、エピジェネティックな制御の異常が様々な疾患の原因となることが分かってきました。CUT&RUN (Cleavage Under Target & Release Using Nuclease) は、タンパク質とDNAの相互作用を解析する新技術です。
CUT&RUNはin vivoにて、標的に特異的な一次抗体とProtein A-Protein G-Micrococcal Nuclease (pAG-MNase) を用いて、特定のタンパク質-DNA複合体を分離します1,2,3。この手法によって細胞から目的のDNAをわずか1 - 2日で回収することができ、自動化してスループットと再現性を最大限に高めることも可能です4。
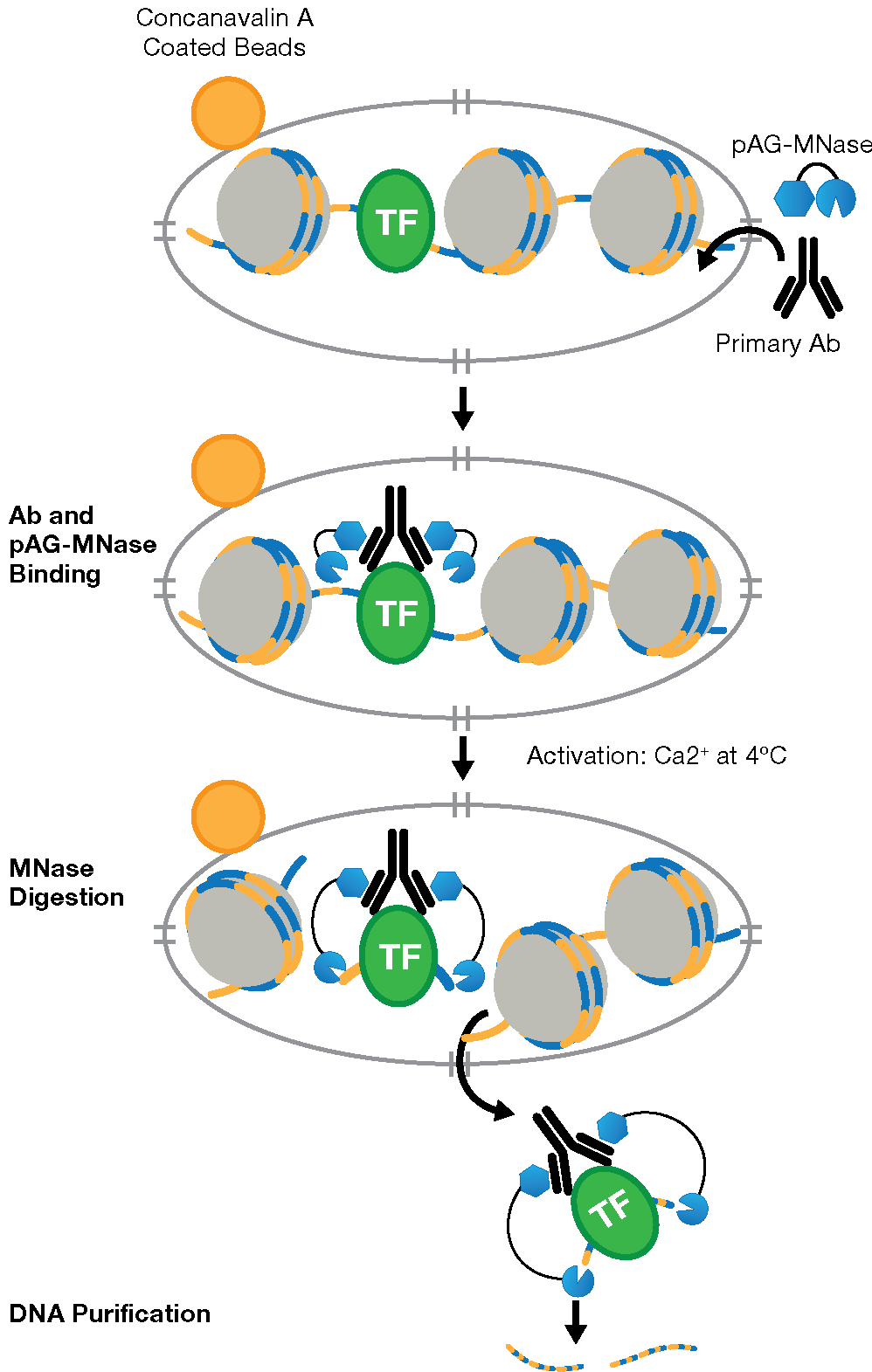
目的のタンパク質とDNAの複合体を得るため、はじめに細胞をコンカナバリンAでコーティングした磁気ビーズに結合させます。これによって細胞の取り扱いを簡便化し、後の洗浄操作での脱落を最小限に抑えます。細胞膜をジギトニンで透過化処理して一次抗体の核への進入を促進し、目的のヒストン、転写因子あるいはコファクターに結合させます。次に、pAG-MNase融合タンパク質のpAGドメインが一次抗体の重鎖に結合し、目的のクロマチン領域に酵素を配置します。Ca2+を加えることでpAG-MNaseが活性化され、DNAの消化が起こります。これによって切り出されたクロマチン複合体はゲノムクロマチンから解離して核外に拡散します。こうしてサンプルの上清に拡散したDNAをスピンカラム、あるいはフェノール/クロロホルム抽出とエタノール沈殿で回収します。
精製、濃縮されたDNAはqPCRによる同定や定量、あるいはライブラリーを構築して次世代シーケンサー (NGS) による全ゲノム解析に用いることができます。
Cell Signaling Technology® (CST) は、ほかの全ゲノムマッピング技術が抱える多くの課題を克服するCUT&RUNキットを提供しています。CST CUT&RUNキットには次のような利点があります:
| 短時間でデータ取得が可能 | わずか1-2日で細胞から目的のDNAが得られます |
| 少量のサンプルで解析可能 | 推奨必要細胞数は100,000ですが、5,000 - 20,000細胞でも検証済みです (標的のタイプごとに異なります) |
| シーケンシングコストの低減 | 必要な高品質リードの数はわずか300-500万です |
| 「クロスリンクアーティファクト」の回避 | 未変性のクロマチンを用いたin vivoの解析法です |
| 多様な抗体に対応 | ラビット抗体およびマウス抗体を利用できる |
| 多様な標的に対応 | ヒストンやヒストン修飾、転写因子、コファクターのqPCRデータやシーケンシングデータが得られます |
| 再現性の高い結果 | Spike-In DNAを利用してサンプル間のシグナルを標準化することができます |
10万個の細胞からタンパク質-DNA相互作用の解析が可能なので、培養の時間を節約することができ、存在量の少ない細胞からもエピジェネティックな情報を得ることができます。
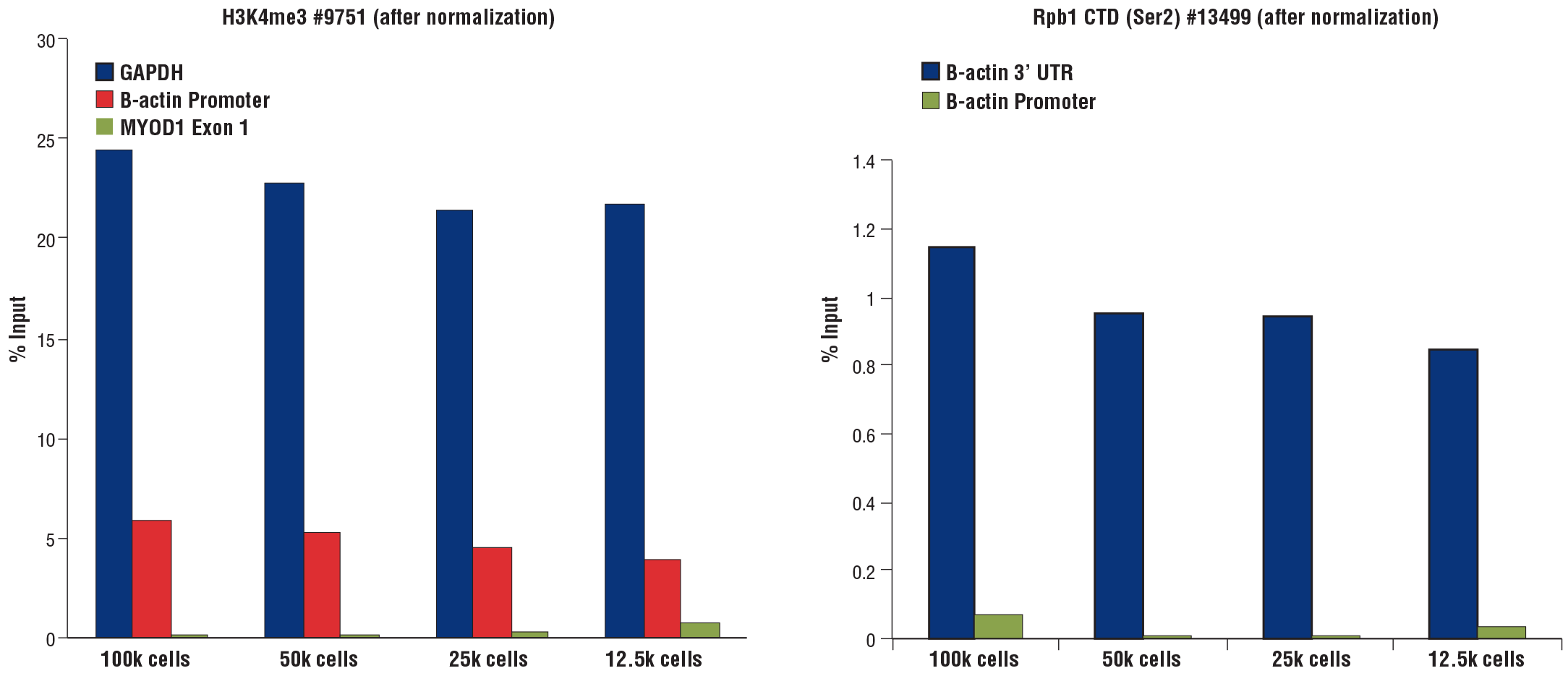
CUT&RUN Assay Kit #86652を用いてCUT&RUNを実施したところ、サンプルであるHCT116細胞の数を徐々に減らしても同様の結果が得られました。左のパネルにはTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いた解析の結果、右のパネルにはPhospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499を用いた解析の結果を示しました。SimpleChIP® Universal qPCR Master Mix #88989を用いて濃縮したDNAをリアルタイムPCRで解析しました。PCRに用いたプライマーセットはそれぞれ、SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human β-Actin Promoter Primers #13653、SimpleChIP® Human β-Actin 3' UTR Primers #13669、SimpleChIP® Human MyoD1 Exon 1 Primers #4490です。CUT&RUNで回収されたDNA量を、Inputクロマチンに対する相対量 (100,000細胞の総クロマチン量を100%とした相対値) で示しました。それぞれの反応系に加えたSpike-In DNAでデータを標準化しました。
標的クロマチンを濃縮する時間を節約できます。わずか1 - 2日で細胞から目的のDNAが得られます。
バックグラウンドが低いので、低深度のシーケンシングでもゲノムのバックグラウンドノイズとシグナルを区別できます。シーケンシングデータを得るのに必要な高品質リードの数はわずか300 - 500万で、シーケンシングコストが大幅に低減されます。また、バックグラウンドが低いので、シグナルの弱いゲノム特性のプロファイルを作成することも可能です。
精製、濃縮されたDNAをシーケンシングやqPCRに用いて、ヒストン修飾や転写因子、コファクターを解析できます。
ヒストン修飾の解析において、CUT&RUN Assay Kit #86652はChIP-qPCRおよびChIP-seqと同等に機能し、わずか100,000細胞からデータが得られます。
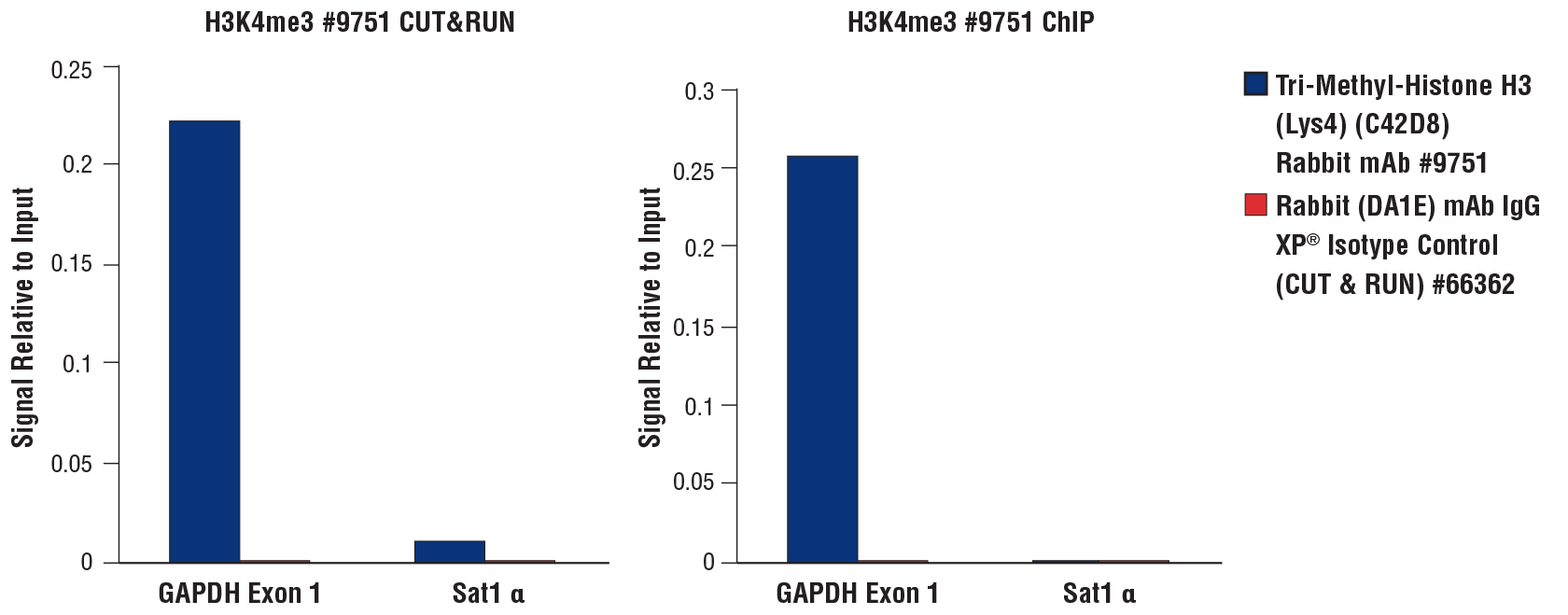
CUT&RUN Assay Kit #86652あるいはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用いて、CUT&RUN (左のパネル) およびChIPアッセイ (右のパネル) を実施しました。それぞれTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751あるいはRabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362を用いて、HCT116細胞 (CUT&RUNは1x105細胞、ChIPは4x106細胞を使用) を解析しました。SimpleChIP® Universal qPCR Master Mix #88989を用いて濃縮したDNAをリアルタイムPCRで定量しました。PCRに用いたプライマーセットはそれぞれ、SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human α Satellite Repeat Primers #4486です。各サンプルで回収されたDNA量を、インプットクロマチンの総量 (1に相当) に対する相対量で示しました。
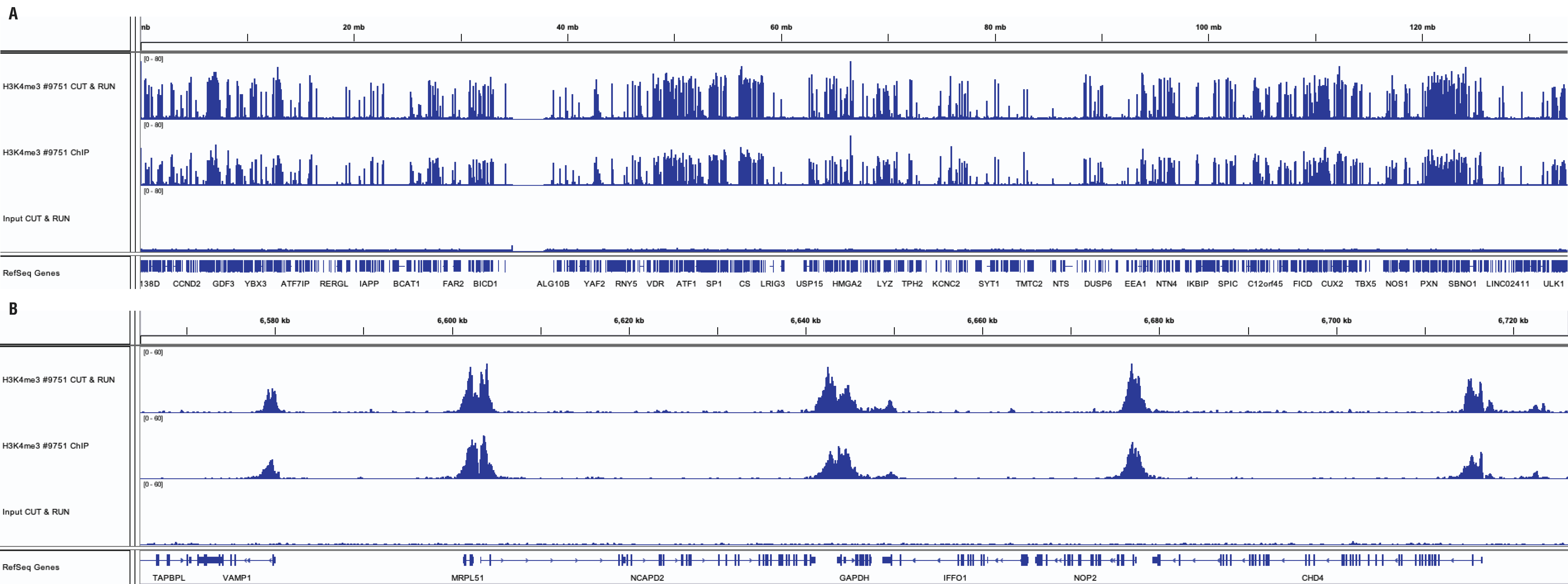
CUT&RUN Assay Kit #86652あるいはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用いて、CUT&RUNおよびChIPアッセイを実施しました。Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いて、HCT 116細胞 (CUT&RUNは1x105細胞、ChIPは4x106細胞を使用) を解析しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。12番染色体の広域に渡る濃縮の比較をパネルA、H3K4me3修飾されることが知られるGAPDH遺伝子の濃縮の比較をパネルBに示しました。InputトラックにはCUT&RUNのInputサンプルから得られたデータを示しました。
転写因子のDNA-タンパク質相互作用の解析において、CUT&RUN Assay Kit #86652はChIP-qPCRやChIP-seqと同等に機能し、わずか100,000細胞からデータが得られます。
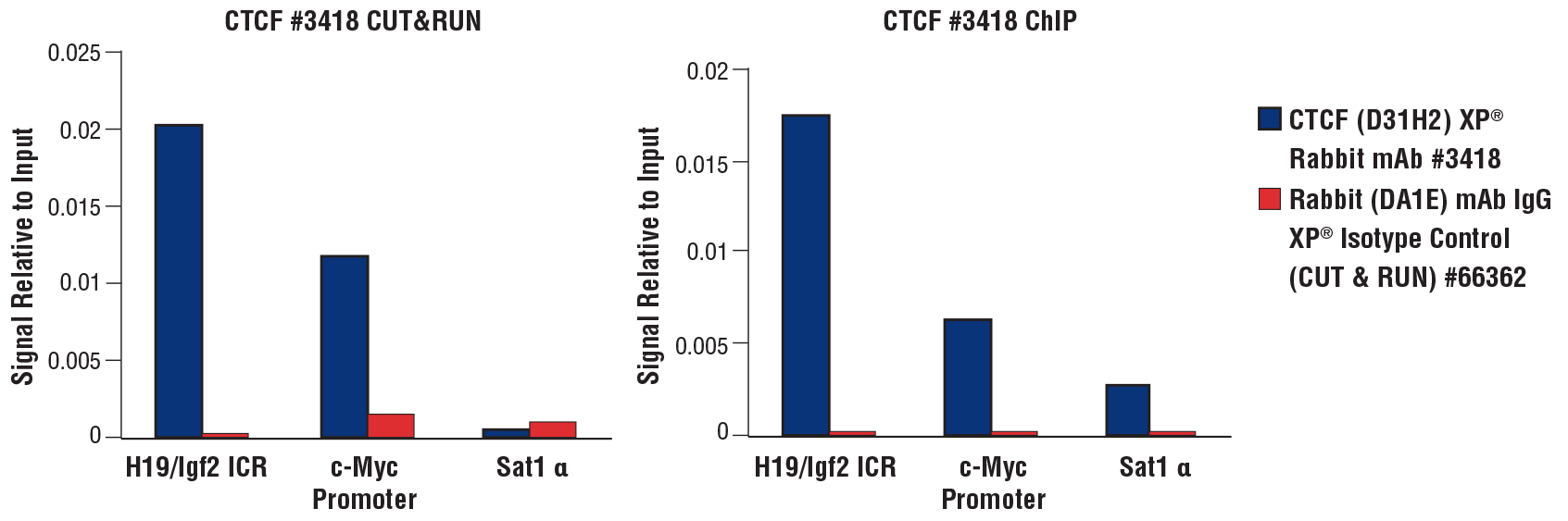
CUT&RUN Assay Kit #86652あるいはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用いて、CUT&RUN (左のパネル) およびChIPアッセイ (右のパネル) を実施しました。それぞれCTCF (D31H2) XP® Rabbit mAb #3418あるいはRabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362を用いて、HCT116細胞 (CUT&RUNは1x105細胞、ChIPは4x106細胞を使用) を解析しました。SimpleChIP® Universal qPCR Master Mix #88989を用いて濃縮したDNAをリアルタイムPCRで定量しました。PCRに用いたプライマーセットはそれぞれ、ヒトc-Mycプロモータープライマー、SimpleChIP® Human H19/Igf2 ICR Primers #5172、SimpleChIP® Human α Satellite Repeat Primers #4486です。各サンプルで回収されたDNA量を、インプットクロマチンの総量 (1に相当) に対する相対量で示しました。
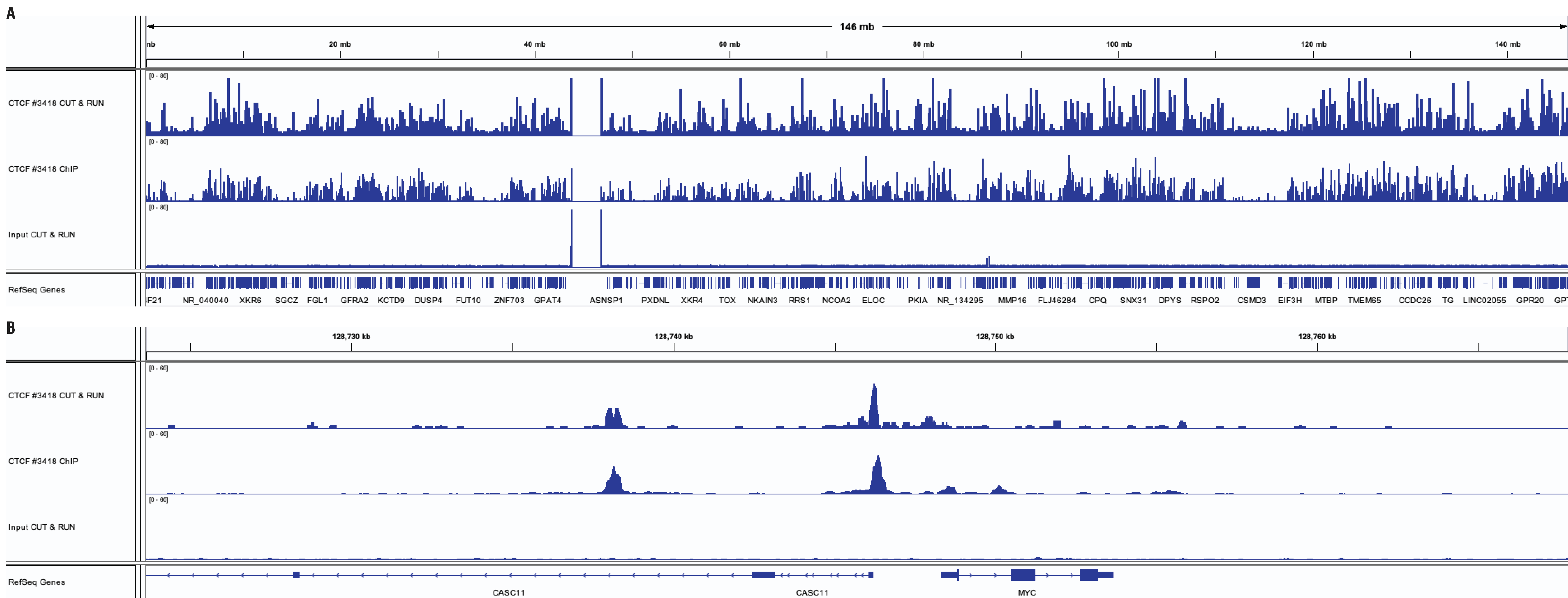
CUT&RUN Assay Kit #86652あるいはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用いて、CUT&RUNおよびChIPアッセイを実施しました。CTCF (D31H2) XP® Rabbit mAb #3418を用いて、HCT116細胞 (CUT&RUNは1x105細胞、ChIPは4x106細胞を使用) を解析しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。8番染色体の広域に渡る濃縮の比較をパネルA、CTCFの既知の標的であるMYC遺伝子の濃縮の比較をパネルBに示しました。InputトラックにはCUT&RUNのInputサンプルから得られたデータを示しました。
コファクターのDNA-タンパク質相互作用の解析において、CUT&RUN Assay Kit #86652はChIP-qPCRやChIP-seqと同等に機能し、わずか100,000細胞からデータが得られます。
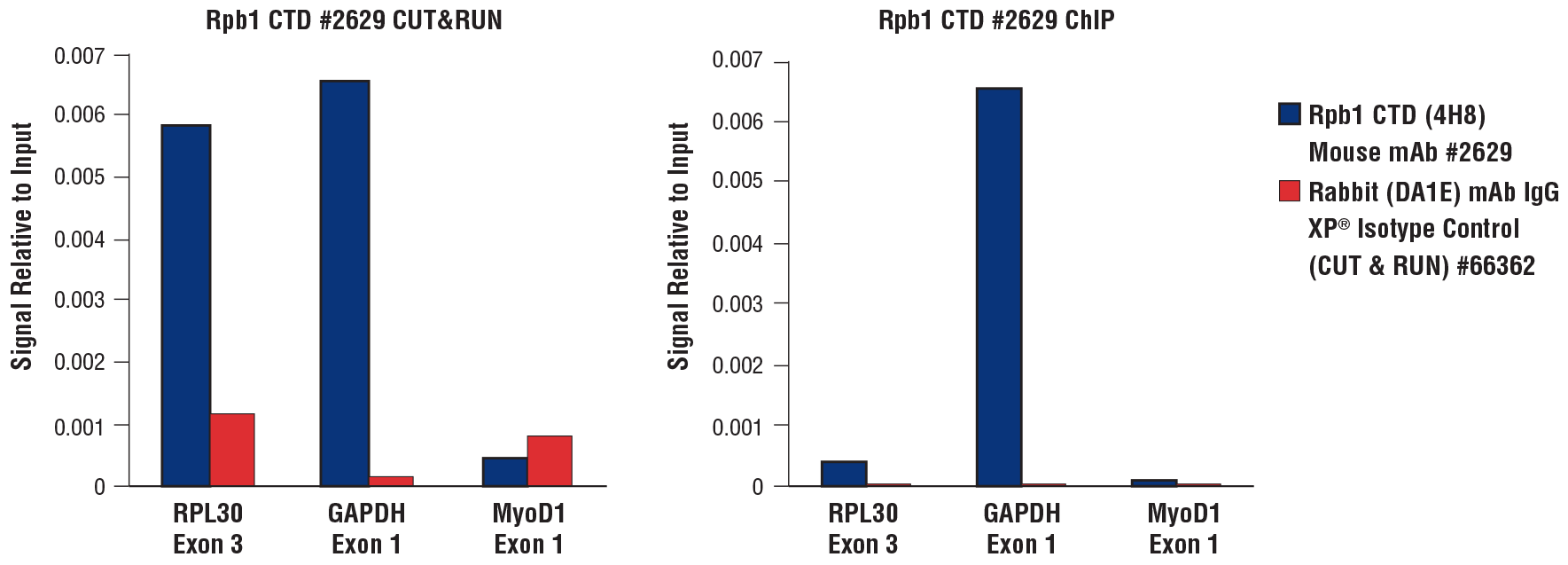
CUT&RUN Assay Kit #86652あるいはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用いて、CUT&RUN (左のパネル) およびChIPアッセイ (右のパネル) を実施しました。それぞれRpb1 CTD (4H8) Mouse mAb #2629あるいはRabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362を用いて、HeLa細胞 (CUT&RUNは1x105細胞、ChIPは4x106細胞を使用) を解析しました。SimpleChIP® Universal qPCR Master Mix #88989を用いて濃縮したDNAをリアルタイムPCRで定量しました。PCRに用いたプライマーセットはそれぞれ、SimpleChIP® Human RPL30 Exon 3 Primers #7014、SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human MyoD1 Exon 1 Primers #4490です。各サンプルで回収されたDNA量を、インプットクロマチンの総量 (1に相当) に対する相対量で示しました。
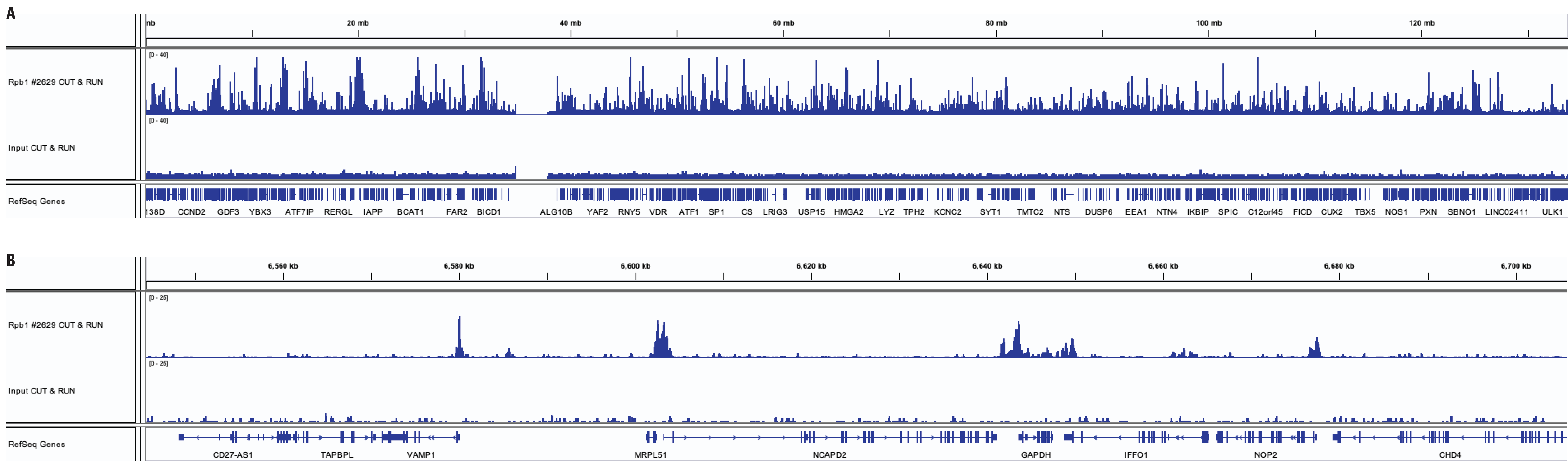
CUT&RUN Assay Kit #86652を用いて、CUT&RUNおよびChIPアッセイを実施しました。Rpb1 CTD (4H8) Mouse mAb #2629を用いて、HeLa細胞 (CUT&RUNは1x105細胞、ChIPは4x106 細胞を使用) を解析しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。12番染色体の広域に渡る濃縮の比較をパネルA、Rpb1の既知の標的であるGAPDH遺伝子の濃縮の比較をパネルBに示しました。InputトラックにはCUT&RUNのInputサンプルから得られたデータを示しました。
pAG-MNaseはマウス抗体とラビット抗体の両方に結合するので、非常に多くの抗体をCUT&RUNアッセイに用いることができます。
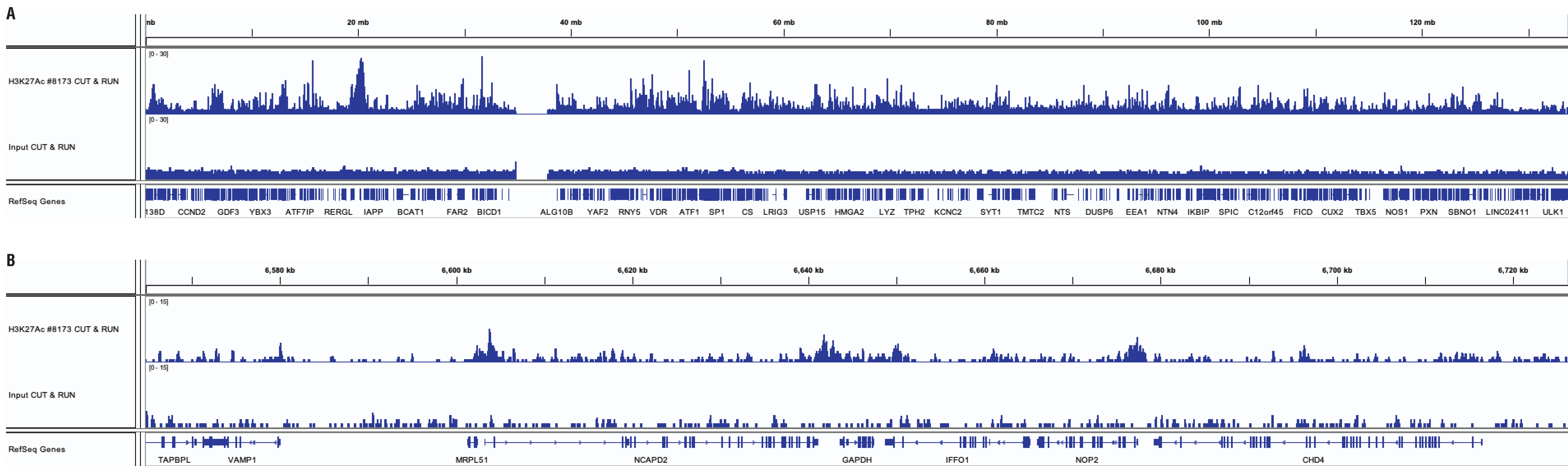
CUT&RUN Assay Kit #86652を用いて、HeLa細胞をAcetyl-Histone H3 (Lys27) (D5E4) XP® Rabbit mAb #8173によるCUT&RUNで解析しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。12番染色体の広域に渡る濃縮の比較をパネルA、H3K27Ac修飾されることが知られるGAPDH遺伝子の濃縮の比較をパネルBに示しました。
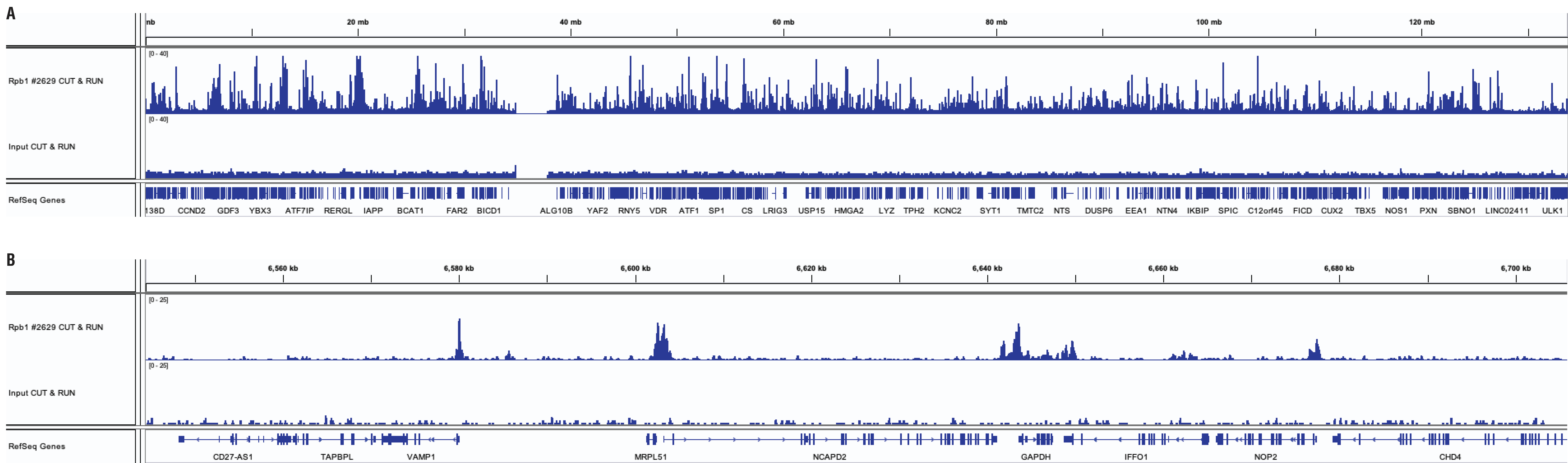
CUT&RUN Assay Kit #86652を用いて、HeLa細胞をRpb1 CTD (4H8) Mouse mAb #2629によるCUT&RUNで解析しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。12番染色体の広域に渡る濃縮の比較をパネルA、Rpb1の既知の標的であるGAPDH遺伝子の濃縮の比較をパネルBに示しました。
異なる生物種のSpike-In DNAを用いてデータを標準化することで、実験の再現性が向上します。既定の実験や異なる実験のサンプル間で標準化ができます。
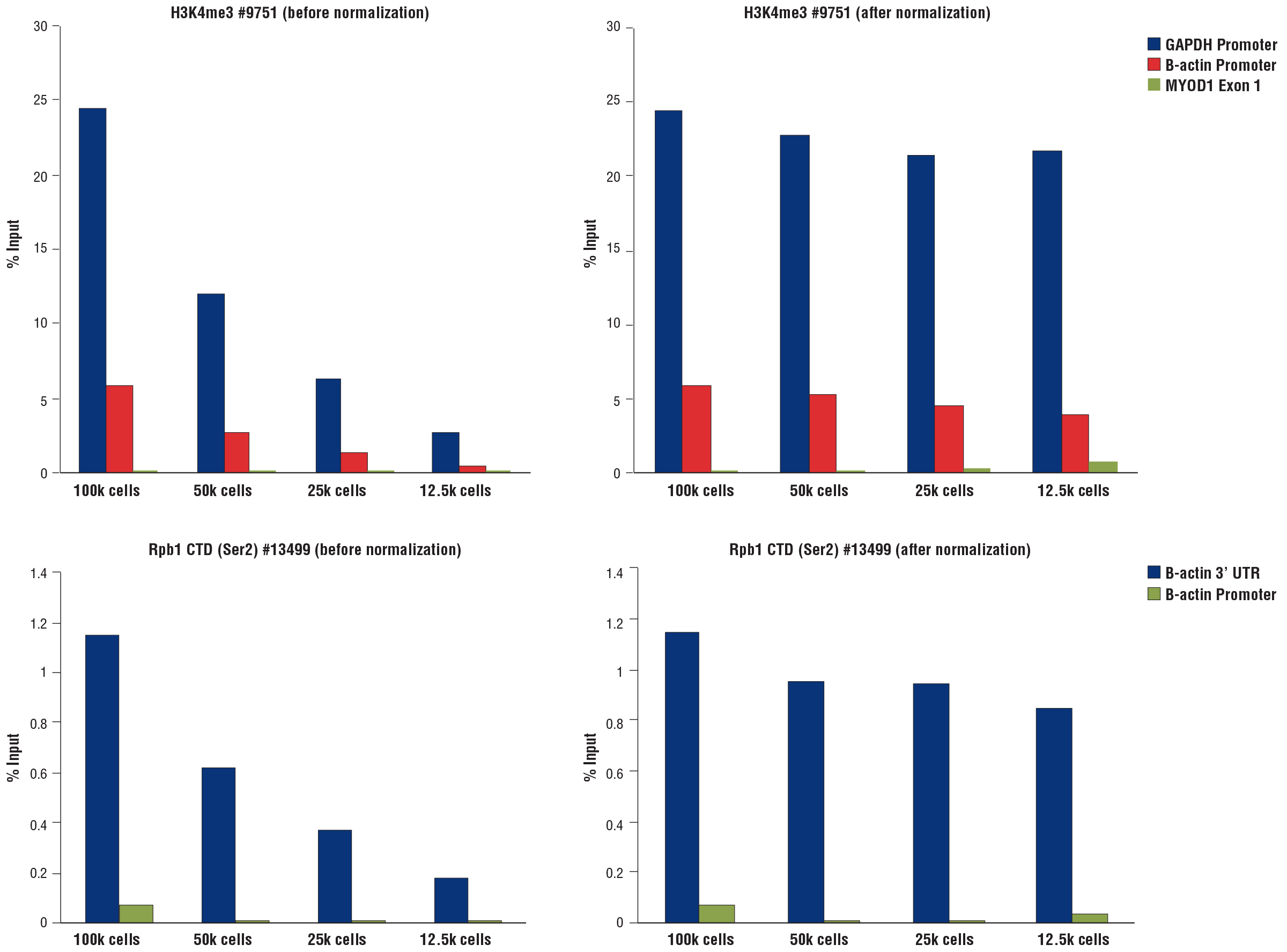
Spike-In DNAを用いて、qPCR解析でのCUT&RUNシグナルを標準化しました。CUT&RUN Assay Kit #86652を用いて、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 (上のパネル) あるいはPhospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499 (下のパネル) による、異なる細胞数のHCT 116細胞のCUT&RUNアッセイを実施しました。SimpleChIP® Universal qPCR Master Mix #88989を用いて濃縮したDNAをリアルタイムPCRで解析しました。PCRに用いたプライマーセットはそれぞれ、SimpleChIP® Human GAPDH Exon 1 Primers #5516、SimpleChIP® Human β-Actin Promoter Primers #13653、SimpleChIP® Human β-Actin 3' UTR Primers #13669、SimpleChIP® Human MyoD1 Exon 1 Primers #4490です。CUT&RUNで回収されたDNA量を、Inputクロマチンに対する相対量 (100,000細胞の総クロマチン量を100%とした相対値) で示しました。標準化前の濃縮結果を左のパネルに示しました。各反応にSpike-In DNAを最初の細胞数に比例して加えました。各サンプルのSpike-In DNAからのqPCRシグナルに基づき、CUT&RUNシグナルを100,000細胞のサンプルに標準化しました。標準化後の濃縮結果を右のパネルに示しました。
CUT&RUNは未変性クロマチンに対して作用し、クロスリンクや免疫沈降を必要としないので「クロスリンクアーティファクト」が起こりません。
CSTは、エピジェネティクス解析の初心者から専門家まで柔軟にご利用いただけるCUT&RUNアッセイ製品を提供しています。お客様ご自身で別途ご用意いただく必要があるのは、標的タンパク質に対する一次抗体のみです。そのほか必要なバッファーや試薬、詳細なプロトコールはすべて、CSTが簡単に注文できるCUT&RUN キットとして提供します。お客様の必要に応じて、pAG-MNaseとSpike-In DNAのみをご購入いただくこともできます。
CUT&RUNアッセイ製品は、CSTの他の製品と同様に社内で厳格に検証されていますので、お客様の実験に安心してご利用いただけます。
CSTが、より良い信号対雑音比を産み出し、タンパク質-DNA相互作用解析のための、より少ない必要細胞量、および組織サンプルのための適合性のために、どのようにCUT&RUNキットを検証して最適化したかを、知ってください。CSTのCUT&RUN Assay Kitプロトコールの全ての改善点について情報を得て最大限に生かしてください。
| 製品番号 | 製品 |
|---|---|
| 86652 | CUT&RUN Assay Kit |
| 40366 | CUT&RUN pAG-MNase and Spike-in DNA |
| 14209 | DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN) |
| 88989 | SimpleChIP® Universal qPCR Master Mix |
| 56795 | DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795 |
| 47538 | Multiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) |
| 29580 | Multiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) |
| 66362 | Rabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) |
| 93569 | Concanavalin A Magnetic Beads and Activation Buffer |
| 16359 | Digitonin Solution |
| 27287 | 100X Spermidine |
| 7012 | Protease Inhibitor Cocktail (200X) |
| 7013 | RNAse A (10 mg/ml) |
| 10012 | Proteinase K (20 mg/ml) |
| 31415 | CUT&RUN 10X Wash Buffer |
| 15338 | CUT&RUN Antibody Binding Buffer |
| 48105 | CUT&RUN 4X Stop Buffer |
| 42015 | CUT&RUN DNA Extraction Buffer |
| 20533 | 10% SDS Solution |
| 7005 | Glycine Solution (10X) |
| 12606 | 16% Formaldehyde Methanol Free |
| 9872 | PBS-1X pH 7.2 (Sterile) |