View in English?
View in English?
社内検証では、複数のヒストン修飾、転写因子、コファクターにおいて、1アッセイにつき100,000個の細胞を用いることで標的遺伝子座を濃縮できることが分かっています。しかしながら、ビーズや抗体、バッファーをスケールアップすることなく、アッセイごとの細胞数は250,000まで増やすことができます。
CSTのCUT&RUN Assay Kit #86652を用い、わずか5,000 - 10,000細胞でヒストン修飾、10,000 - 20,000細胞で転写因子やコファクターを解析できることを示しました (図参照)。
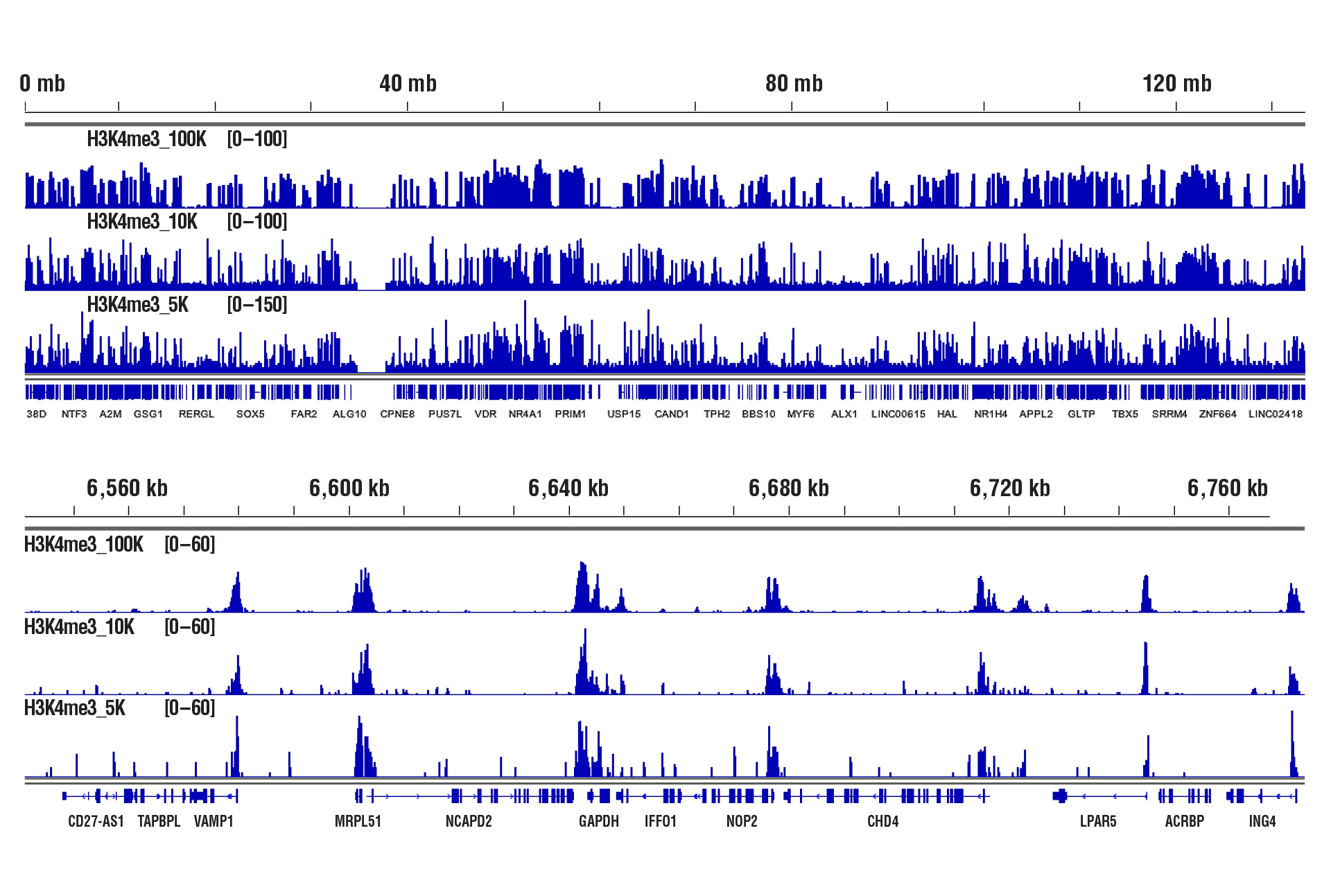
CUT&RUN Assay Kit #86652を用い、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるHCT116細胞のCUT&RUNを行いました。図中に示したように、アッセイには100,000細胞、10,000細胞、5,000細胞を用いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、H3K4me3の既知の標的遺伝子であるGAPDH (下パネル) および、これを含む12番染色体全体への結合 (上パネル) を示しています。
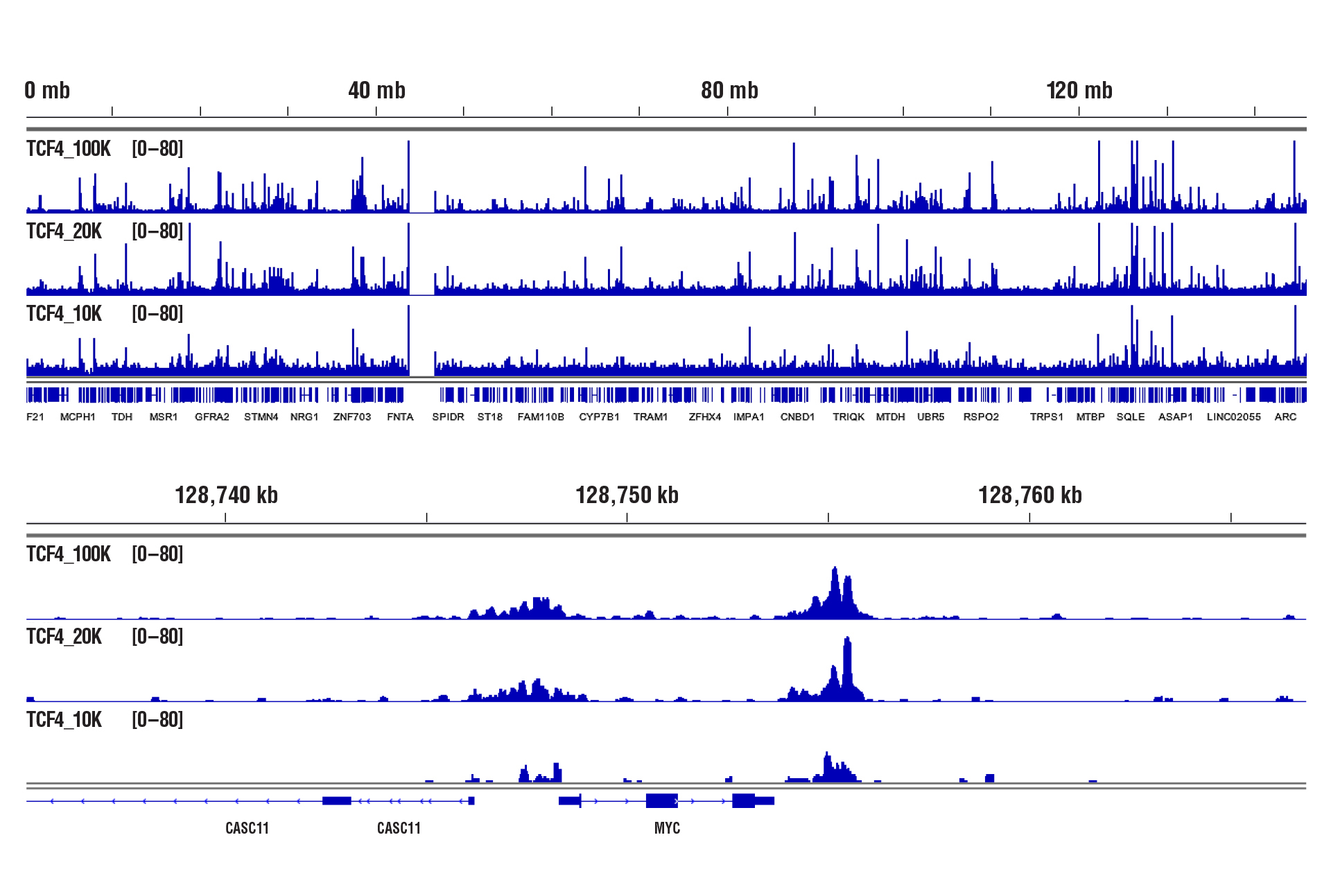
CUT&RUN Assay Kit #86652を用い、TCF4/TCF7L2 (C48H11) Rabbit mAb #2569によるHCT116細胞のCUT&RUNを行いました。図中に示したように、アッセイには100,000細胞、20,000細胞、10,000細胞を用いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、TCF4の既知の標的遺伝子であるMYC (下パネル) および、これを含む8番染色体全体への結合 (上パネル) を示しています。
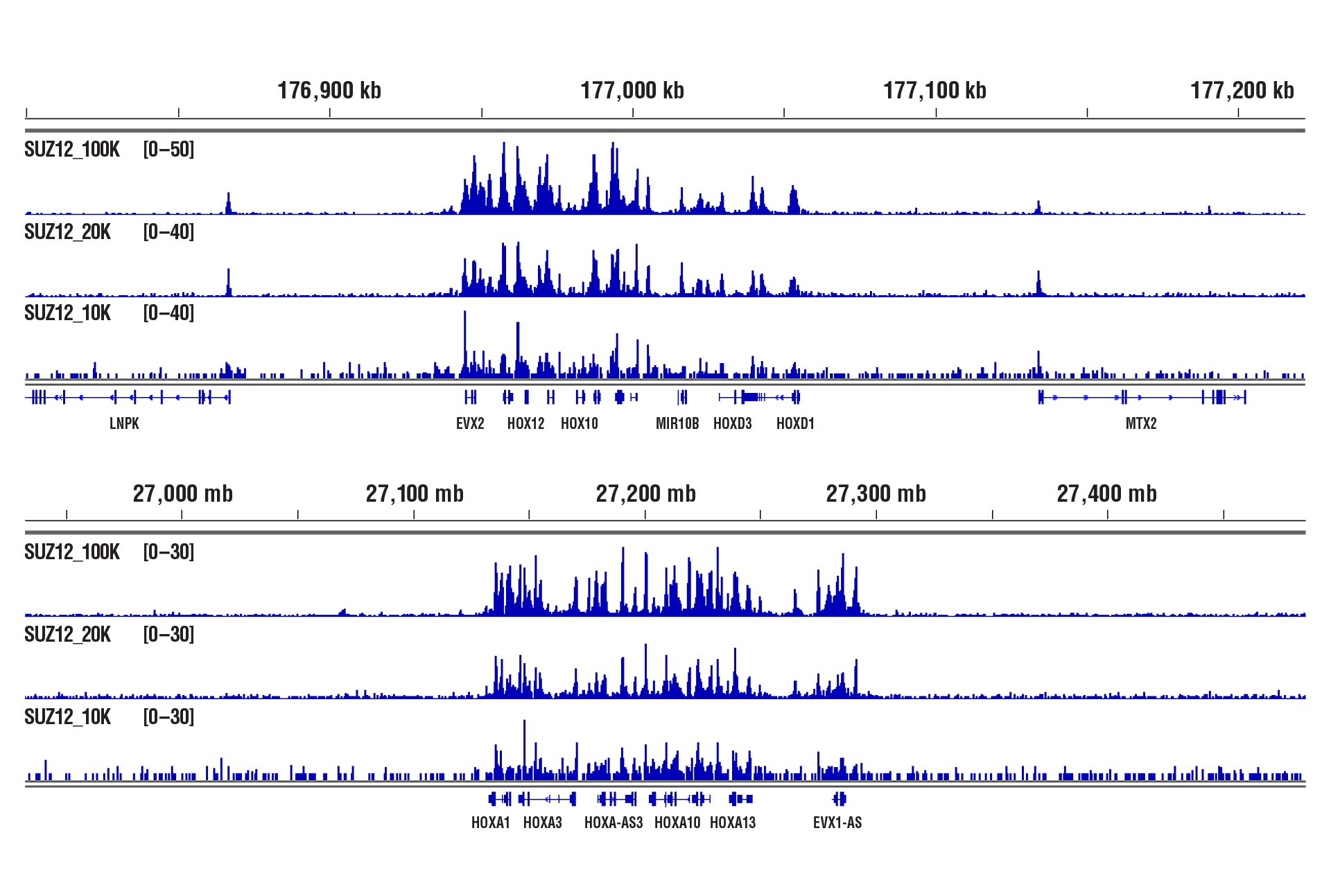
CUT&RUN Assay Kit #86652を用い、SUZ12 (D39F6) XP® Rabbit mAb #3737によるNCCIT細胞のCUT&RUNを行いました。図中に示したように、アッセイには100,000細胞、20,000細胞、10,000細胞を用いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、SUZ12の既知の標的遺伝子であるHoxD (上パネル) およびHoxA (下パネル) 遺伝子クラスター全体への結合を示しています。
社内検証で、ヒトとマウスの数十種類の接着細胞株、浮遊細胞株を試しています。現在のところ、異なる細胞株の間で、アッセイの効率に大きな違いはみられません。
接着細胞株の場合は、まず細胞をディッシュから剥離する必要があります。細胞の剥離には、トリプシンの使用を推奨しています。Accutaseも使用可能ですが、細胞ペレットの粘着性が弱くなるために、洗浄のステップで細胞を失いやすくなります。浮遊細胞の場合はより簡単です。血球計算盤または自動細胞数測定器で細胞をカウントし、必要数の細胞を回収してください。接着細胞と浮遊細胞の間で、アッセイの効率に大きな違いはみらません。
はい。壊れ易い (実験中に溶解し易い) 細胞を用いる場合、細胞の結合に用いるコンカナバリンAビーズが細胞のシグナル伝達経路に影響を及ぼす場合、標的タンパク質とDNAの結合が弱い場合、将来的に使用する細胞ペレットを凍結保存したい場合などには、細胞の固定を推奨します。固定した細胞でCUT&RUNを成功させるには、固定を軽く (0.1%ホルムアルデヒドで2分間) 行うことが非常に重要です。また、DNAからタンパク質を除去するため、アッセイの最後に脱クロスリンクが必要です。CSTの社内試験では、一般に固定した細胞より生細胞を用いた方がCUT&RUNで良好な結果が得られているため、上記したような場合を除き、なるべく生細胞を用いることを推奨します。
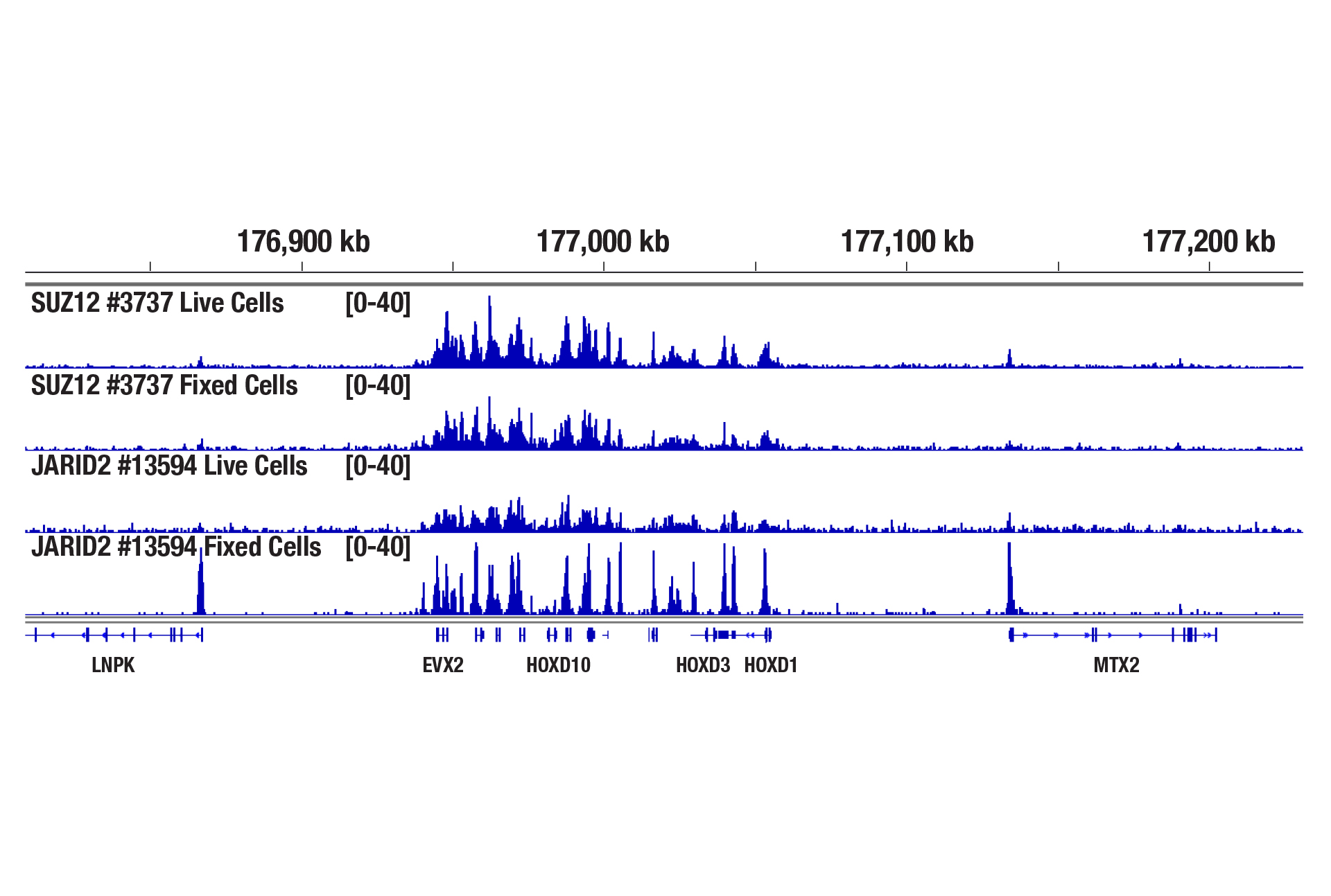
CUT&RUN Assay Kitを用い、SUZ12 (D39F6) XP® Rabbit mAb #3737またはJARID2 (D6M9X) Rabbit mAb #13594によるNCCIT細胞のCUT&RUNを行いました。アッセイには生細胞または固定した細胞を100,000細胞使用しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、HoxD遺伝子クラスター全体へのSUZ12とJARID2の結合を示しています。SUZ12 (PRC2複合体の中核成分) の場合は固定は不要ですが、JARID2 (PRC2複合体の副成分) の場合は固定によって濃縮が増加します。
はい。CSTのCUT&RUN Assay Kit #86652でCD8+ T細胞の生細胞100,000細胞を用い、エピジェネティクス制御因子や転写因子を解析できることを示しました (図参照)。
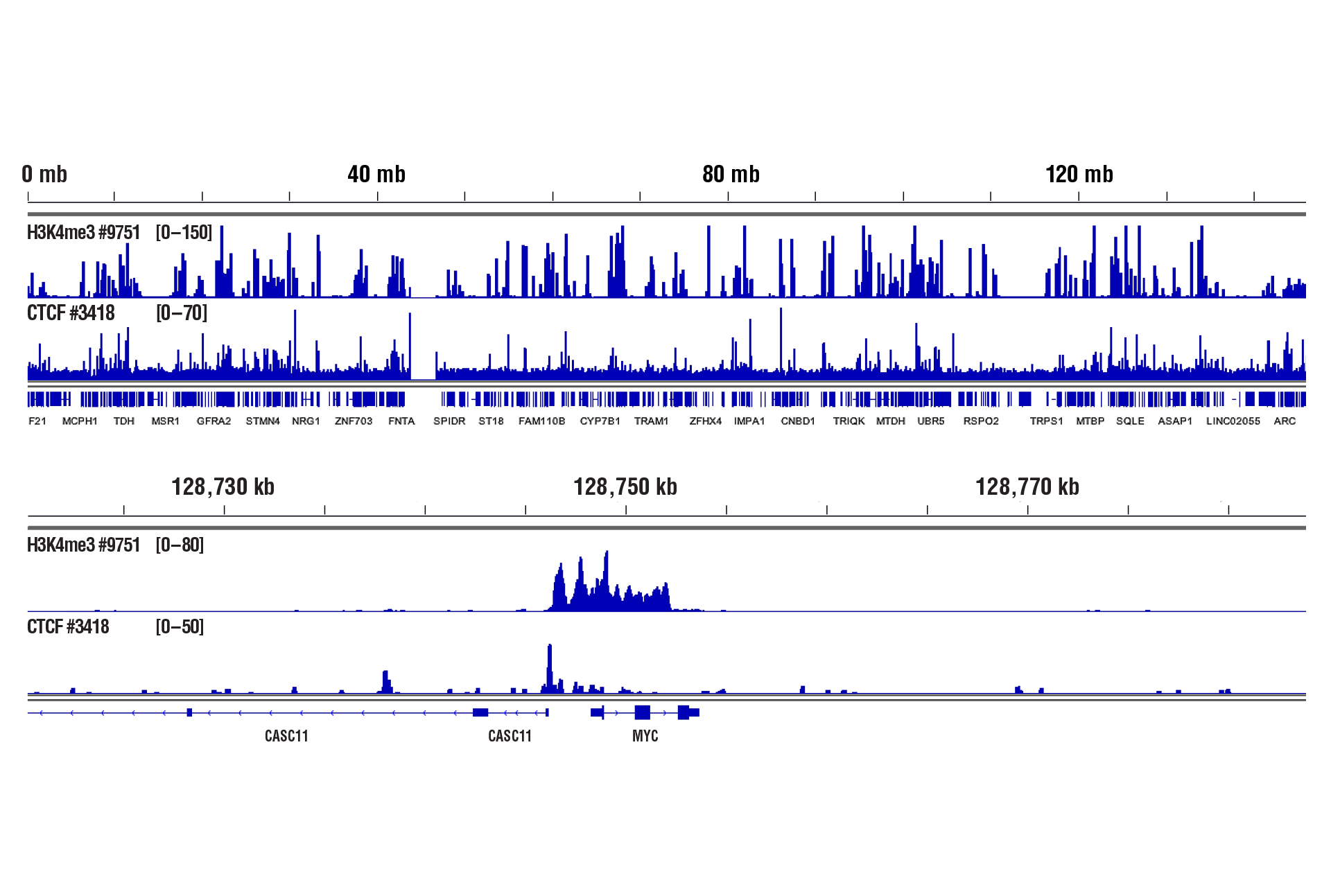
CUT&RUN Assay Kit #86652を用い、CTCF (D31H2) XP® Rabbit mAb #3418またはTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるCD8+ T細胞のCUT&RUNを行いました。図中に示したように、アッセイには生細胞を100,000細胞使用しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、8番染色体全体 (上) および、H3K4me3とCTCFの既知の標的遺伝子であるMYC (下) への結合を示しています。
はい。CSTのCUT&RUN Assay Kit #86652を用い、わずか1 mgのマウス肝臓、脳、心臓組織を解析できることを示しました。ヒストン修飾やCTCFなどの量の多い転写因子は新鮮な組織サンプルを用いて解析することができます。ただし、転写因子やコファクター、あるいは解離が難しい組織を解析する場合、軽い固定または中程度の固定 (0.1%ホルムアルデヒドで2-10分間) を行い、使用する組織の量を少し増やす (2.5-5 mg) ことで最適な結果が得られます (図参照)。CUT&RUNアッセイで使用するまで、固定化された組織は最大6ヵ月、凍結保存できます。
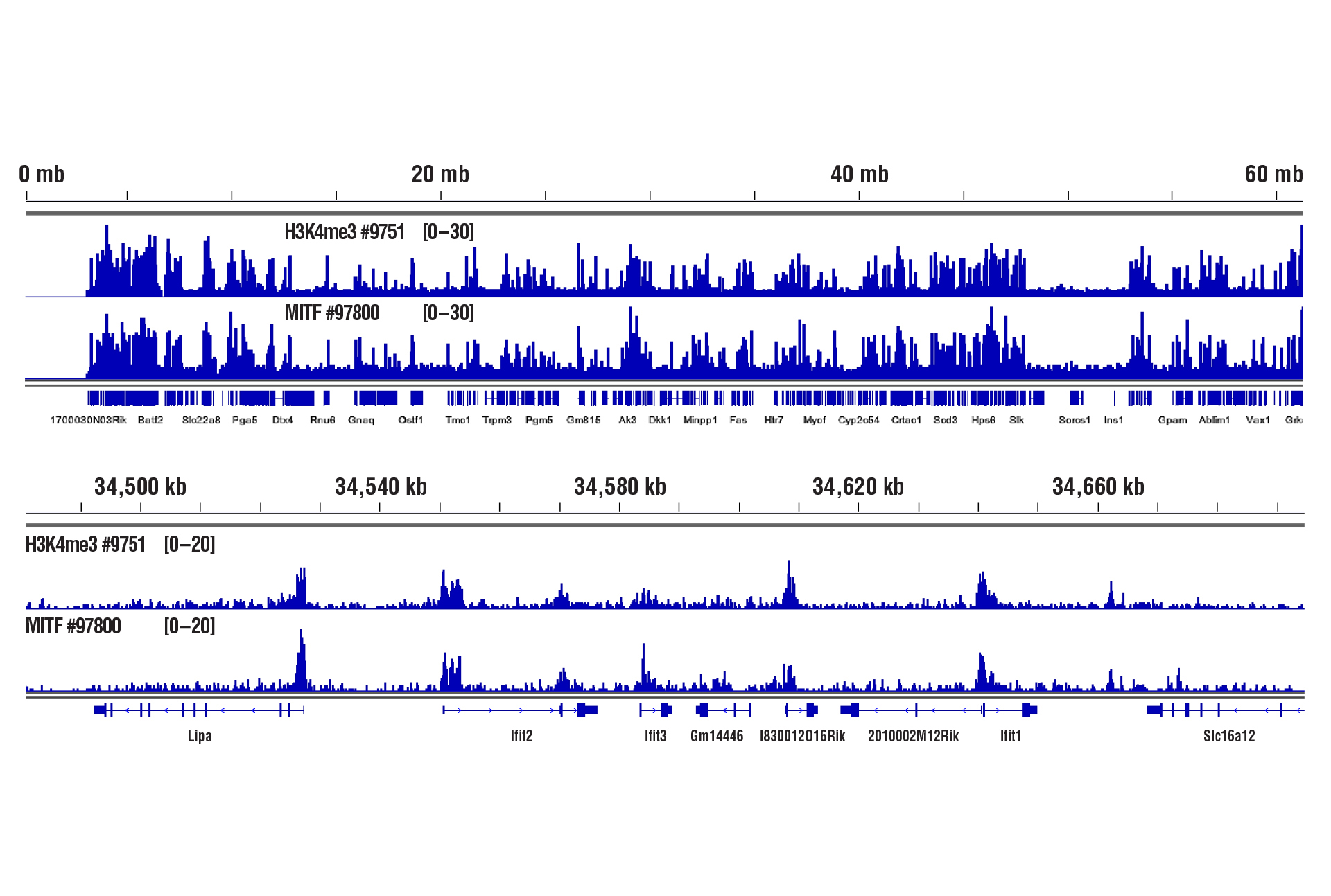
CUT&RUN Assay Kit #86652を用い、MITF (D3B4T) Rabbit mAb #97800またはTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるマウス心臓組織のCUT&RUNを行いました。アッセイには軽く固定した組織 (0.1%ホルムアルデヒドで2分間) を1 mg使用しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Ifit2遺伝子 (下パネル) およびこれを含む19番染色体全体への (上パネル) 、H3K4me3とMITFの結合を示しています。
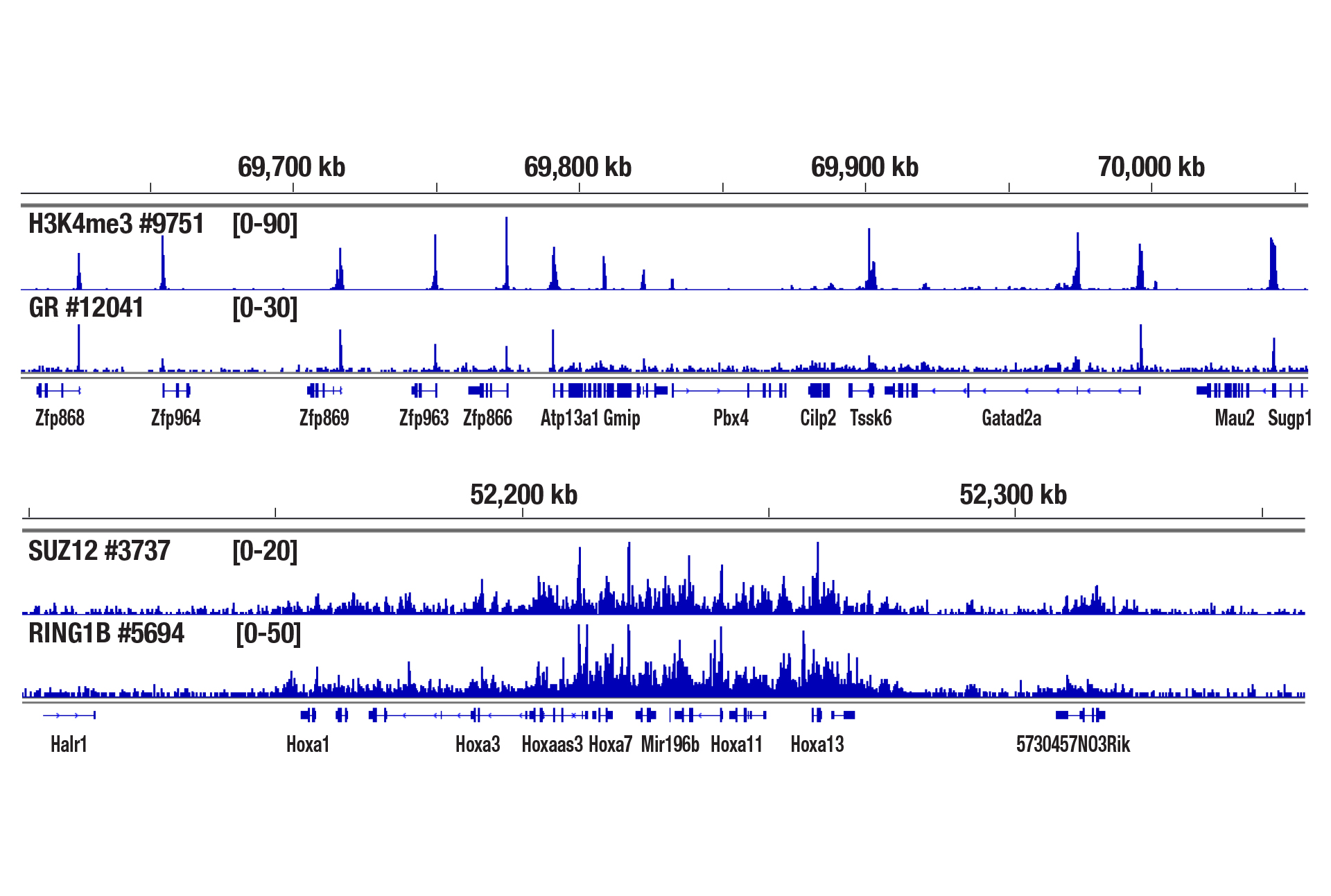
CUT&RUN Assay Kit #86652を用い、Glucocorticoid Receptor (D6H2L) XP® Rabbit mAb #12041、SUZ12 (D39F6) XP® Rabbit mAb #3737、RING1B (D22F2) XP® Rabbit mAb #5694、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるマウス肝臓組織のCUT&RUNを行いました。アッセイには中程度に固定した組織 (0.1%ホルムアルデヒドで10分間) を2.5 mg使用しました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Zfp866遺伝子周辺へのH3K4me3と糖質コルチコイド受容体の結合 (上パネル)、HoxA遺伝子クラスター全体へのSUZ12とRING1B (下パネル) の結合を示しています。
CSTは植物細胞でCUT&RUN Assay Kitの検証を行っていません。しかしながら、Xiao-Yu Zheng et al. (2019; PMID30719569)に、植物組織の前処理と、核を調製してCUT&RUNに用いる方法が記載されています。CSTのキットもこのステップを経れば、植物サンプルでもご使用いただける可能性はあります。この場合、ジギトニン処理は必要ないと考えられますが、 植物細胞が100%均質でない場合には、細胞膜の透過性を上げるためにジギトニン処理を行った方が良い可能性もあります。
Concanavalin Aビーズは、特に細胞が溶解された時に凝集しやすくなります。細胞の生存や増殖に異常が無いことを確認し、細胞洗浄の操作は穏やかに行ってください。細胞懸濁液とConcanavalin Aビーズの室温でのインキュベーション時間に注意し、5分間を超えないようにすることを推奨します。細胞とビーズの結合や抗体の結合、pAG-MNase酵素の結合を促進する方法として、インキュベーション中にチューブを回転または振盪させる代わりに、適切なインキュベーション温度でチューブを静置することもできます。このインキュベーション方法は、ビーズの凝集を軽減する効果があり、最終的な結果に悪影響を与えることはないとされています (下図参照)。とはいえ、CSTの社内試験では、ビーズの凝集は最終的な結果に悪影響を及ぼさないことが分かっています。
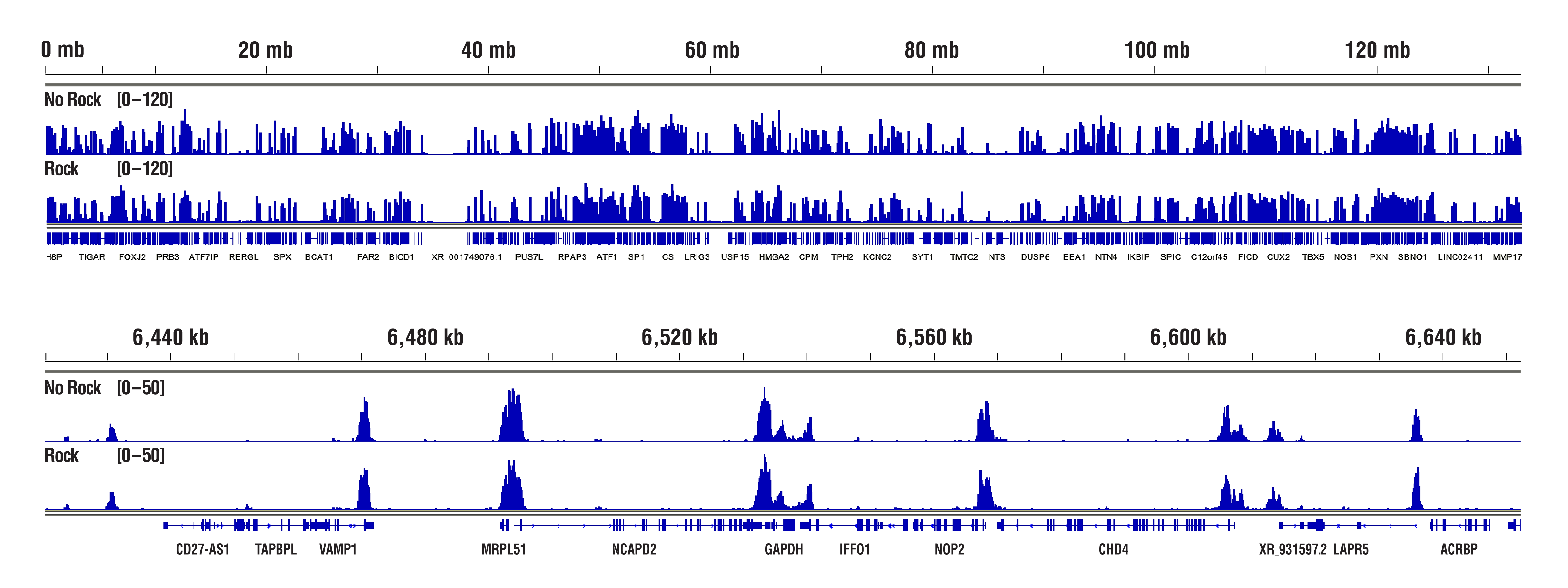
H3K4me3のデータから、「振盪していない」サンプルと「振盪した」サンプルで同等の結果が得られることがわかります。 CUT&RUN Assay Kit #86652を用い、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるCUT&RUNを行いました。アッセイには20,000細胞のHCT116細胞を使用しました。細胞とビーズの結合、抗体の結合、pAG-MNase酵素の結合のため、サンプルは適切な温度での静置 (振盪なし) またはローテーターを用いて (振盪あり) インキュベーションを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、12番染色体全体にわたる結合 (上パネル) と、これに含まれるH3K4me3の既知の標的遺伝子であるGAPDHへの結合 (下パネル) を示しています。振盪なしまたは振盪ありのプロトコールによる結果に、シグナル強度や結合パターンの有意な差はありません。
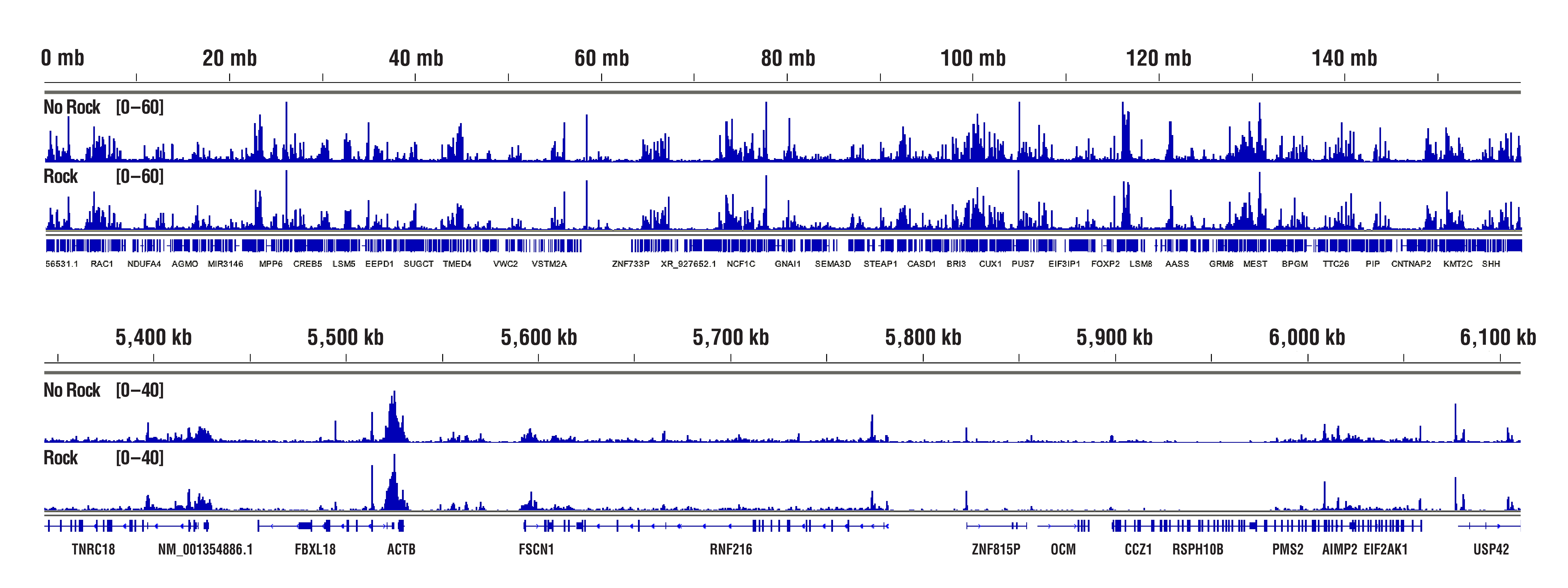
Rpb1のデータから、「振盪していない」サンプルと「振盪した」サンプルで同等の結果が得られることがわかります。 CUT&RUN Assay Kit #86652を用い、Phospho-Rpb1 CTD (Ser2) (E1Z3G) Rabbit mAb #13499によるCUT&RUNを行いました。アッセイには100,000細胞のHCT116細胞を使用しました。細胞とビーズの結合、抗体の結合、pAG-MNase酵素の結合のため、サンプルは適切な温度での静置 (振盪なし) またはローテーターを用いて (振盪あり) インキュベーションを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、Phospho-Rpb1の既知の標的遺伝子であるACTB (下パネル) および、これを含む7番染色体全体への結合 (上パネル) を示しています。振盪なしまたは振盪ありのプロトコールによる結果に、シグナル強度や結合パターンの有意な差はありません。
CSTが最適化したCUT&RUNアッセイの消化条件は、接着細胞、浮遊細胞を問わず複数の細胞株で機能します。また、pAG-MNaseや消化の時間を増減させても、クロマチン消化に大きな変化はみられません。Peter Skene et al. (2018; PMID29651053) にも、CSTと同様の見解が記載されています。とは言え、消化温度は重要です。社内検証では、転写因子やコファクターの解析で、0˚Cより4˚Cで消化した方がバックグラウンドは許容範囲に抑えられたままシグナル強度が増強することが分かっています。
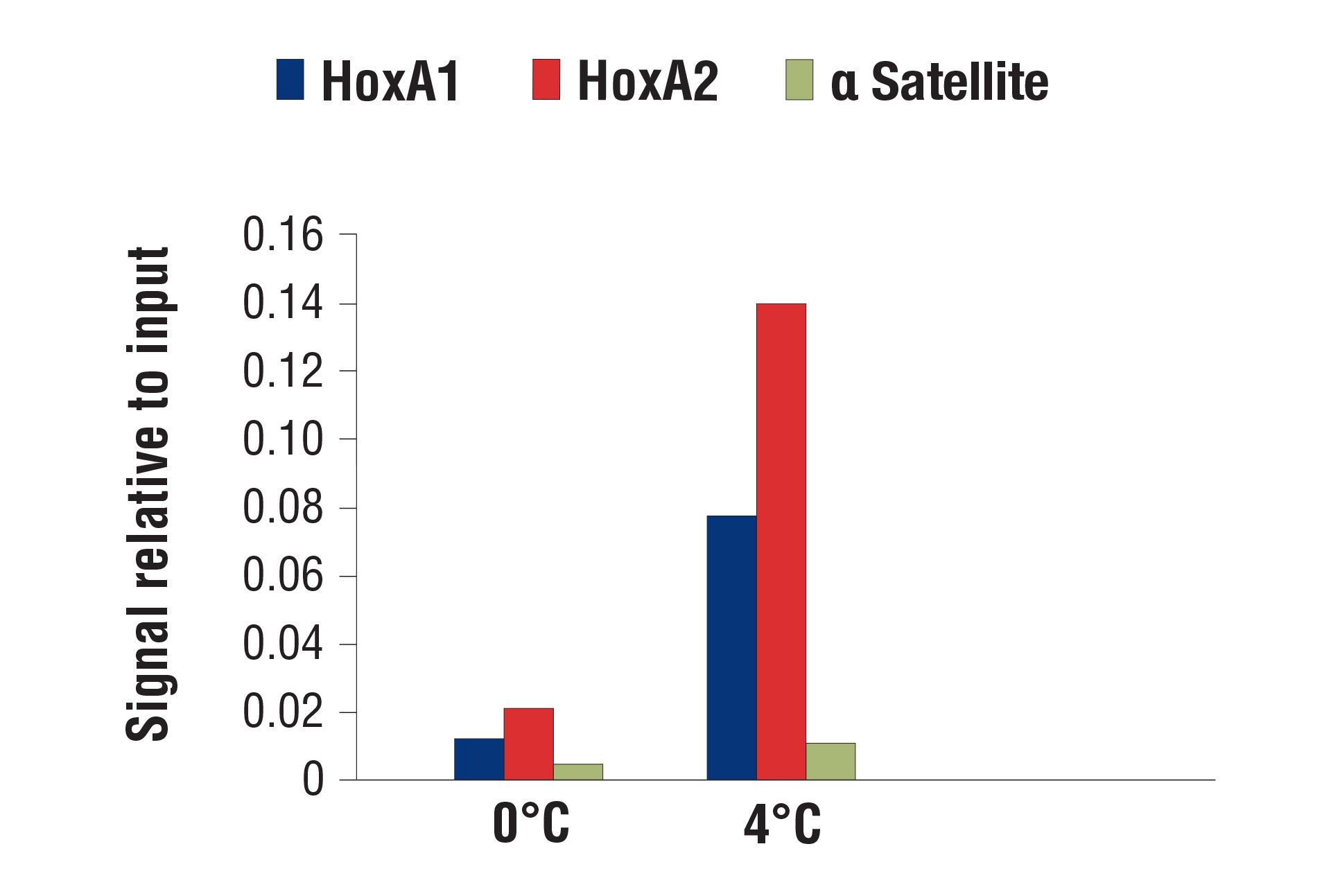
CUT&RUN Assay Kit #86652を用い、SUZ12 (D39F6) XP® Rabbit mAb #3737によるNCCIT細胞のCUT&RUNアッセイ行いました。図に示したように、DNA消化は0°Cまたは4°Cで行いました。SimpleChIP® HoxA1 Intron 1 Primers #7707、SimpleChIP® Human HoxA2 Promoter Primers #5517、SimpleChIP® Human α Satellite Repeat Primers #4486のプライマーセットでリアルタイムPCRを行い、濃縮されたDNAを定量解析しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。
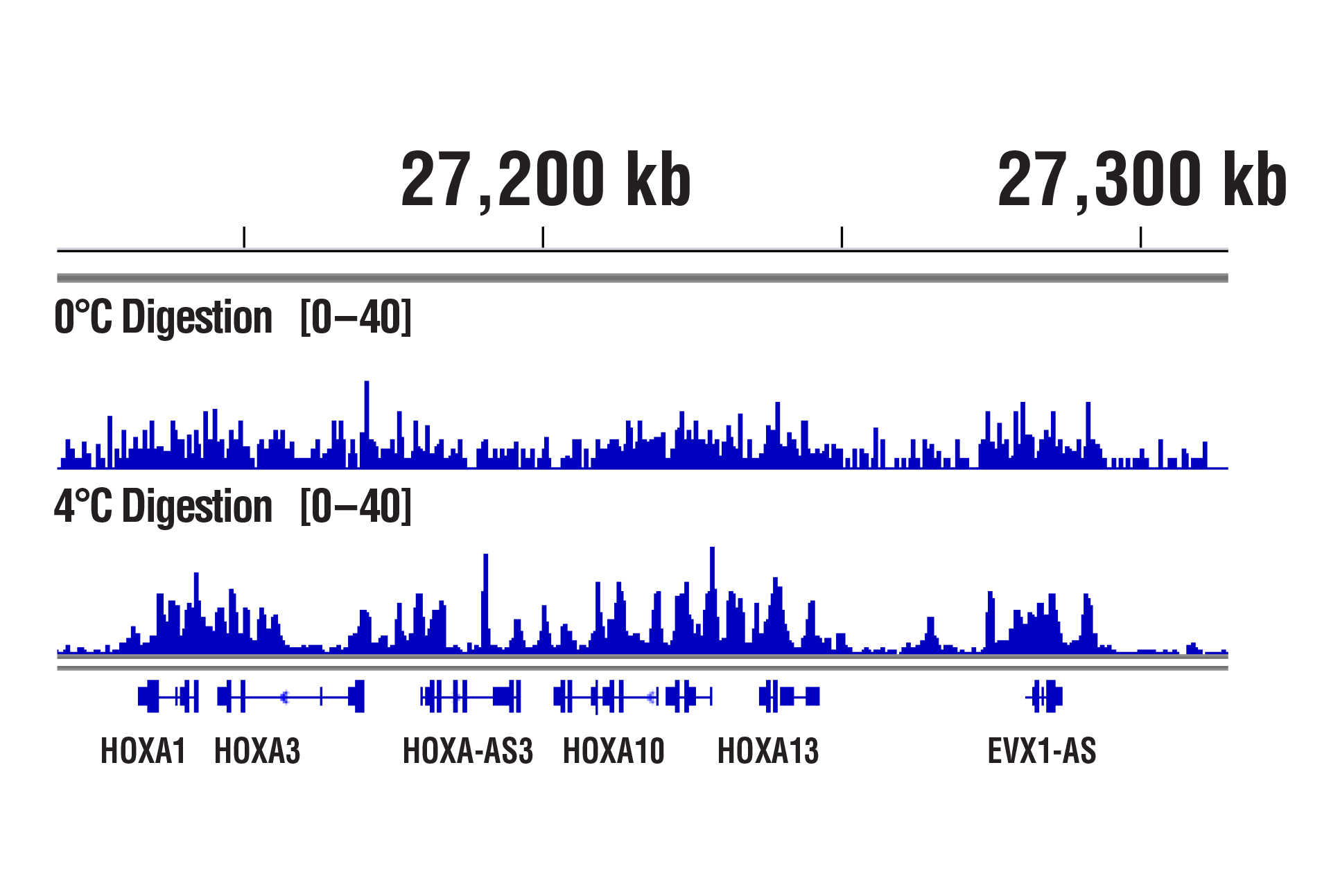
CUT&RUN Assay Kit #86652を用い、SUZ12 (D39F6) XP® Rabbit mAb #3737によるNCCIT細胞のCUT&RUNアッセイ行いました。図に示したように、DNA消化は0°Cまたは4°Cで行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、HoxA遺伝子全体へのSUZ12の結合を示しています。
はい。ジギトニンによる膜の透過化は可逆的であるため、実験に用いるすべてのバッファーにジギトニンを加えておく必要があります。バッファーからのジギトニンの除去や濃度の低下は、抗体やpAG-MNaseの核への進入に影響を及ぼす可能性があります。細胞は、ジギトニンの処理時間よりも、ジギトニンの濃度に高い感受性を示します。推奨ジギトニン濃度で細胞の広範な溶解がみられる場合は、CUT&RUN Assay KitプロトコールのAppendix Aの記載に従い、ジギトニン濃度を最適化してください。
CUT&RUN Assay Kitは全細胞で使用できるように最適化されており、接着細胞、浮遊細胞を問わず多くの細胞株で使用できます。通常、CUT&RUNでは核を予め単離する必要はありませんが、Concanavalin A ビーズは細胞表面と核表面の両方に存在する糖タンパク質に結合するため、CSTのキットは予め単離した核で使用することもできます。細胞壁をもつ酵母や植物細胞など、ジギトニンで効率的に透過化できない細胞の場合のみ、予め核を単離する必要があります。
いいえ。CSTの社内検証でユークロマチンやヘテロクロマチンに対するバイアスはみられません。CUT&RUNでは標的抗体に特異的抗体がpAG-MNaseをリクルートし、ユークロマチン、ヘテロクロマチンを問わず、抗体に直接隣接するクロマチンを消化します。この場合、pAG-MNaseを積極的に繋ぎ止めることで、アクセスが難しいヘテロクロマチンの消化も可能になります。CSTのCUT&RUN Assay Kit #86652は、アクセスし易いユークロマチンの転写活性化ヒストン修飾 (Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751参照)、転写を活性化する転写因子 (NF-kB p65 [D14E12] XP® Rabbit mAb #8242)参照) と同様に、アクセスし難いヘテロクロマチンの転写抑制ヒストン修飾 (Tri-Methyl-Histone H3 [Lys27] [C36B11] #9733参照)、不活性なヘテロクロマチンに結合するポリコーム抑制複合体タンパク質 (EZH2 [D2C9] XP® Rabbit mAb #5246およびRING1B [D22F2] XP® Rabbit mAb #5694参照) に対する抗体で使用できることが示されています。
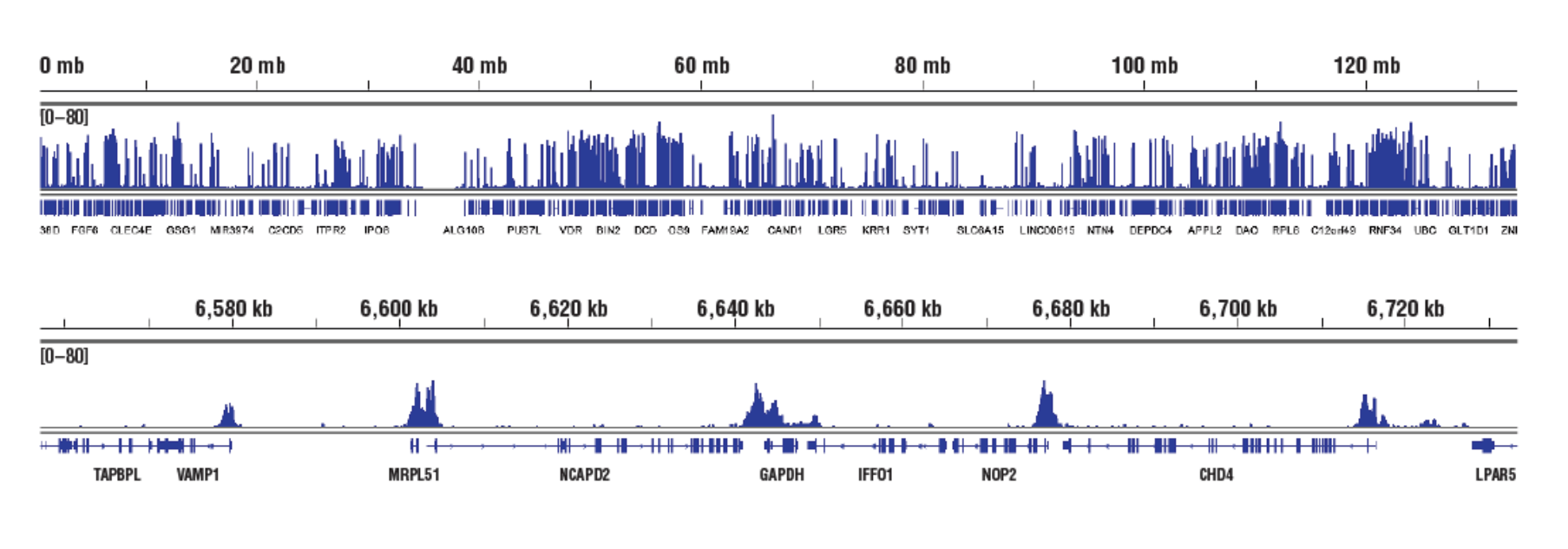
標的:H3K4me3 (アクセスし易いクロマチン): CUT&RUN Assay Kit #86652を用い、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるHCT116細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、12番染色体全体にわたる結合 (上パネル) と、これに含まれるH3K4me3の既知の標的遺伝子であるGAPDHへの結合 (下パネル) を示しています。
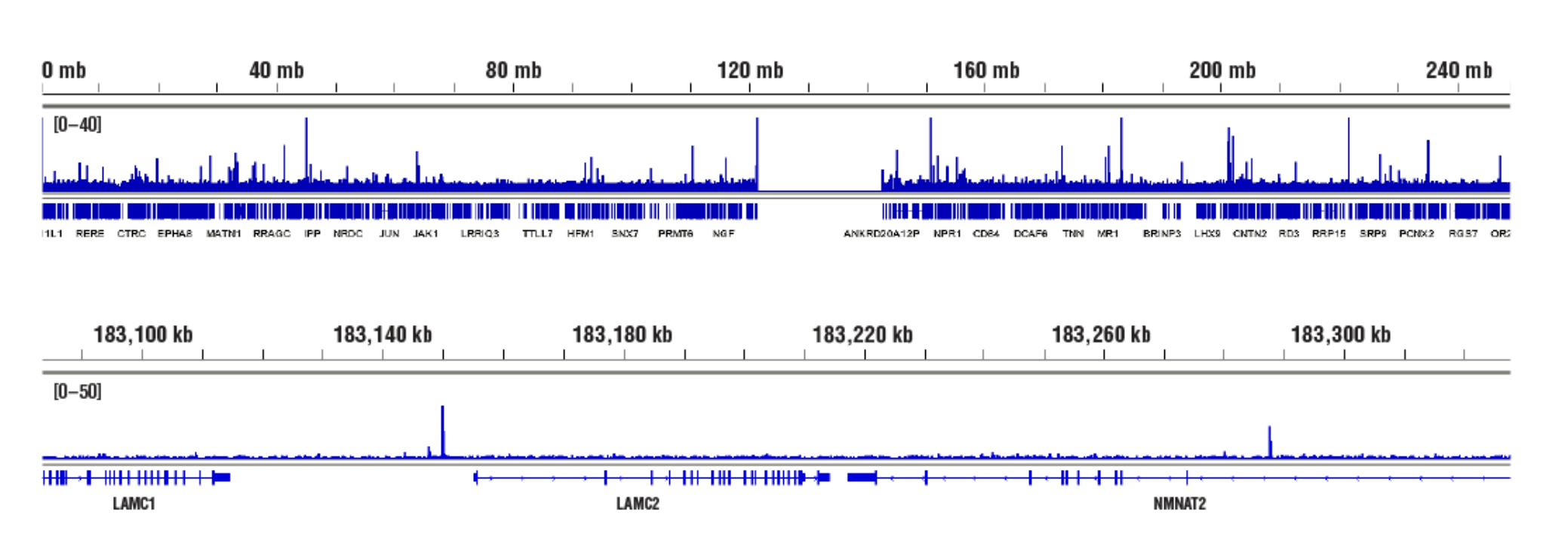
標的:NF-κB p65 (アクセスし易いクロマチン): CUT&RUN Assay Kit #86652を用い、NF-κB p65 (D14E12) XP® Rabbit mAb #8242によるhTNF-α #8902 処理 (30 ng/mL、1時間) したHeLa細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、NF-κB p65の既知の標的遺伝子であるLAMC2 (下パネル) および、これを含む1番染色体全体への結合 (上パネル) を示しています。
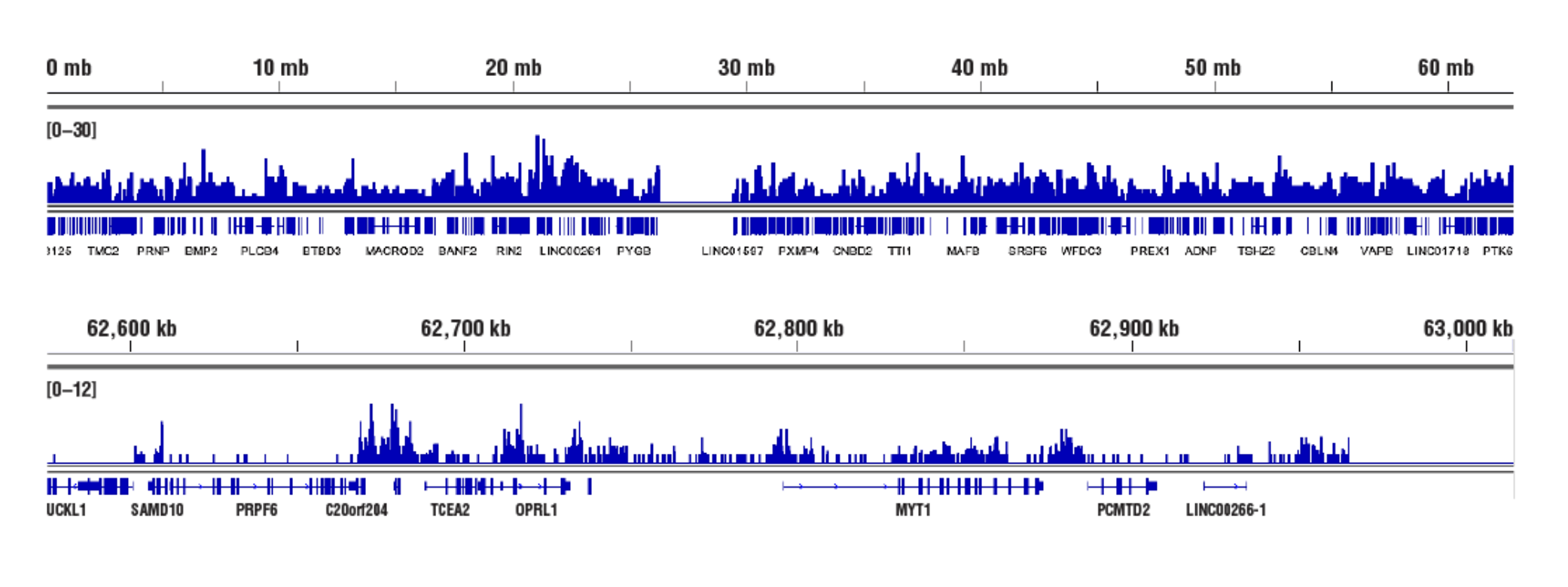
標的:Tri-Methyl-Histone H3 (アクセスし難いへテロクロマチン): CUT&RUN Assay Kit #86652を用い、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733によるHeLa細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、TCEA2 (下パネル) および、これを含む20番染色体全体への結合 (上パネル) を示しています。
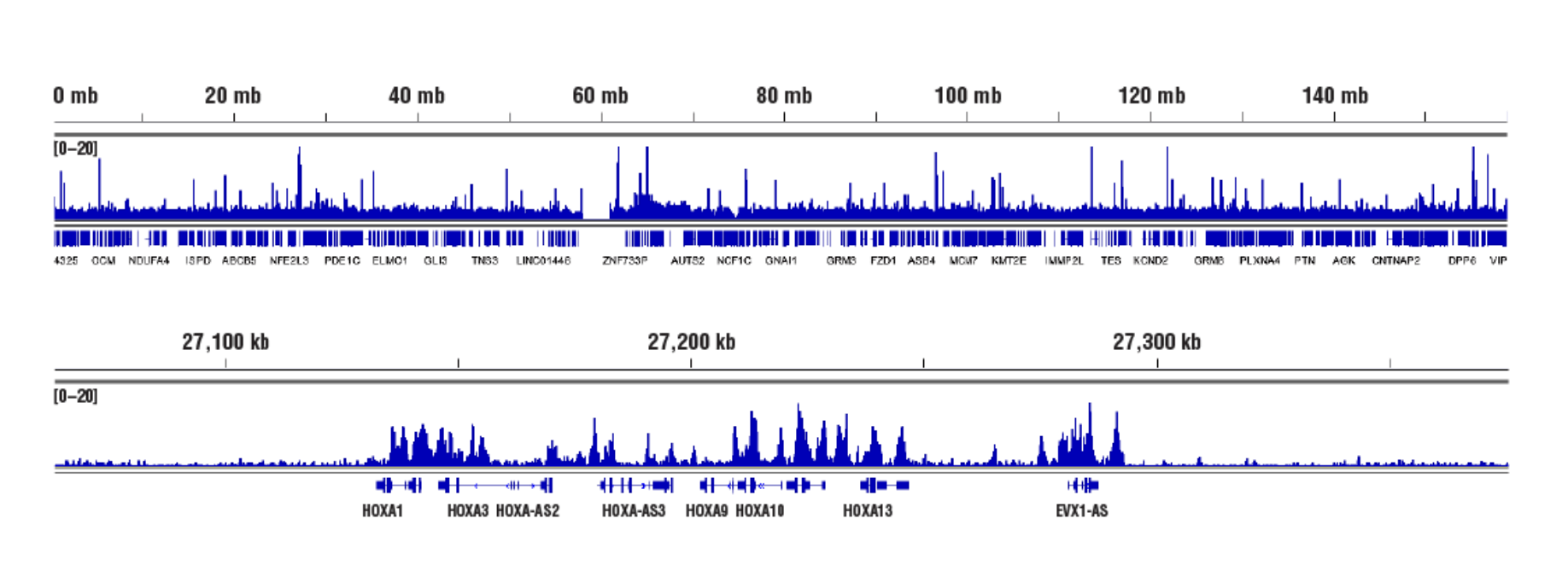
標的:Ezh (アクセスし難いヘテロクロマチン): CUT&RUN Assay Kit #86652を用い、Ezh2 (D2C9) XP® Rabbit mAb #5246によるNCCIT細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。図は、EZH2の既知の標的遺伝子であるHoxA (下パネル) および、7番染色体全体への結合 (上パネル) を示しています。
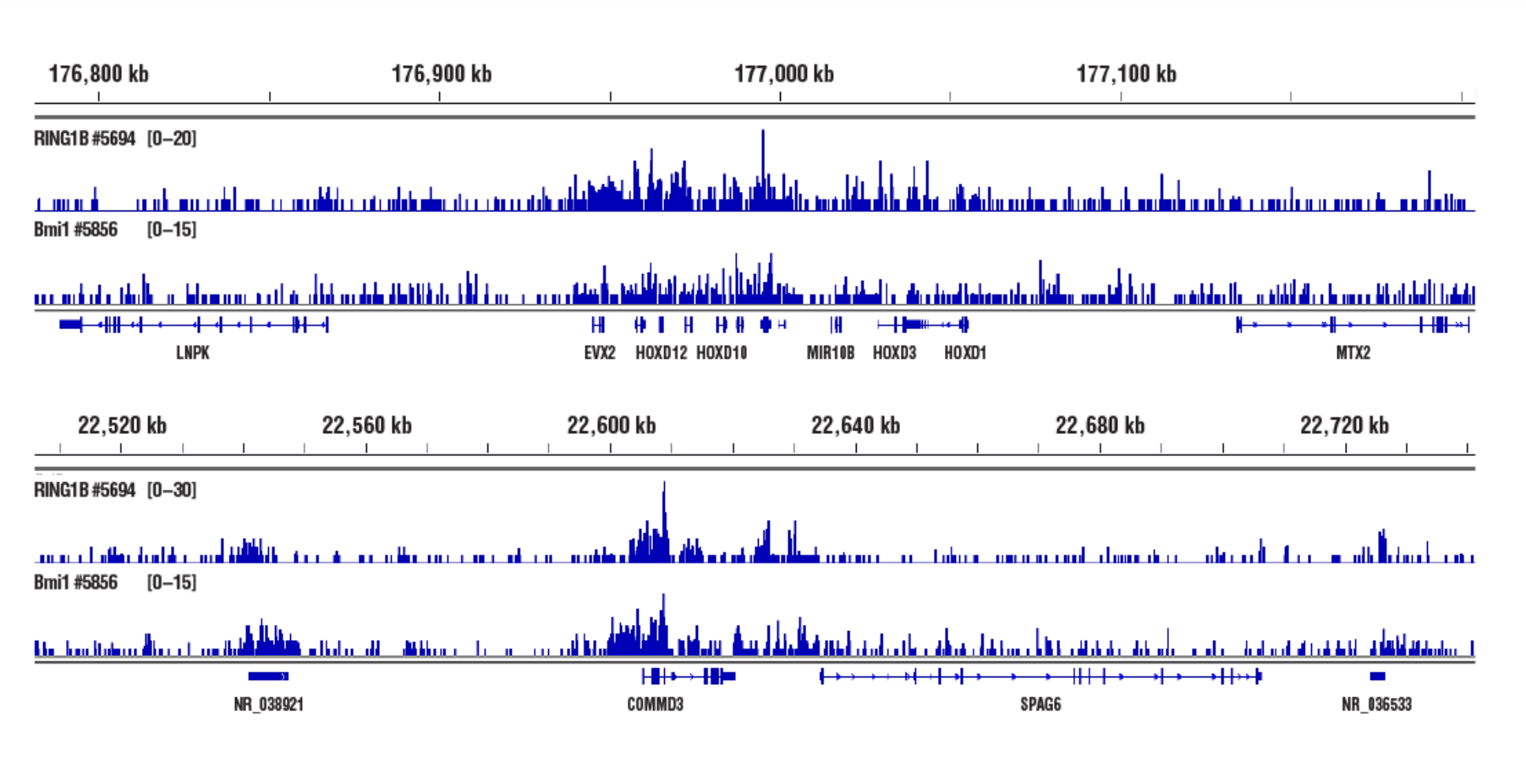
標的:RING1B (アクセスし難いヘテロクロマチン): CUT&RUN Assay Kit #86652を用い、RING1B (D22F2) XP® Rabbit mAb #5694またはBmi1 (D42B3) Rabbit mAb #5856によるNCCIT細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。RING1BとBmi1はともにPRC1の構成因子です。図は、HOXD (上パネル) 遺伝子および、COMMD3 (下パネル) 遺伝子への結合を示しています。
CUT&RUNの後、qPCRで解析する場合とNG-seqで解析する場合のどちらでも、CUT&RUNで検証済みのポジティブコントロール抗体 (Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751など) とネガティブコントロール抗体 (Rabbit (DA1E) mAb IgG XP® Isotype Control Antibody #66362など) を使用することをお勧めします。これらの抗体はSimpleChIP® Human RPL30 Exon 3 Primers #7014またはSimpleChIP® Mouse RPL30 Intron 2 Primers #7015と組み合わせて使用することで、ご希望の標的に特異的な抗体の性能に関わらず、CUT&RUNアッセイ自体が機能していることを確認できます。これら全てのコントロールはCUT&RUN Assay Kit #86652に同梱されています (図参照)。
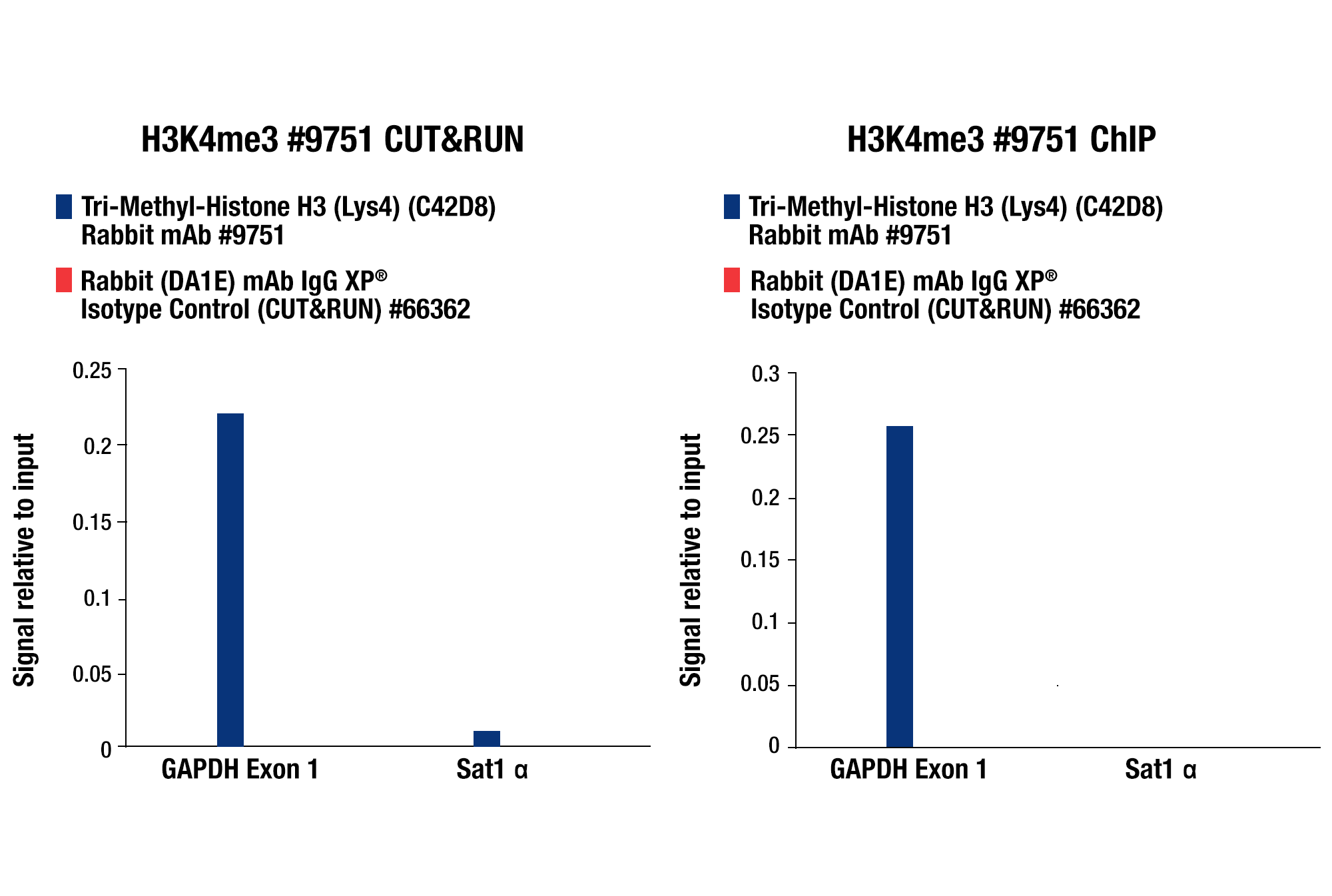
CUT&RUN qPCRコントロールデータ: CUT&RUN Assay Kit #86652またはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用い、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはRabbit (DA1E) mAb IgG XP® Isotype Control (CUT&RUN) #66362によるHCT116細胞のCUT&RUN (左パネル) およびChIPアッセイ (右パネル) を行いました。SimpleChIP® Human GAPDH Exon 1 Primers #5516およびSimpleChIP® Human α Satellite Repeat Primers #4486を用いたリアルタイムPCRにより、濃縮したDNAを解析しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。
CUT&RUN NGSコントロールデータ: CUT&RUN Assay Kit #86652またはSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005を用い、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるHCT116細胞のCUT&RUNおよびChIPアッセイを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。12番染色体の広域にわたる濃縮の比較を上パネルに、H3K4me3の既知の標的であるGAPDH遺伝子の濃縮の比較を下パネルに示しました。InputトラックにはCUT&RUNのInputサンプルから得られたデータを示しました。
ChIPまたはChIP-seq検証済み抗体から始めることを推奨しますが、ChIP検証済み抗体のすべてがCUT&RUNアッセイで機能するとは限らないことが分かっています。他の方法としてPeter J. Skene et al. (2017; PMID28079019)は、CUT&RUNにおける抗体の結合は無傷の核環境で行われ、免疫蛍光染色アッセイにおける抗体の結合条件に類似していることから、免疫蛍光染色アッセイ用の検証済み抗体を試すことを推奨しています。
pAG-MNaseに混入した大腸菌DNAをサンプルの標準化に利用するプロトコールもあります。しかし、pAG-MNaseに混入している大腸菌DNAの量はロットごとに異なるため、ロットが変わる際に問題になることがあります。CSTはこれとは別の、酵母Spike-In DNAサンプルを使用することを推奨しています。酵母Spike-In DNAサンプルは、CUT&RUN Assay Kit #86652およびCUT&RUN pAG-MNase and Spike-In DNA #40366に同梱されており、全てのロットでCUT&RUNに最適化した試験を行っています。この酵母Spike-In DNAは、標準化の作業においてロットごとの一貫性を向上させます。
酵素消化後、DNAを精製する前に、各CUT&RUN反応に予め断片化した酵母のゲノムDNAを一定量加えます。このSpike-In成分によって、DNA精製、qPCR、ライブラリー調製、シーケンシングのサンプルごとの効率の違いを標準化できます。CUT&RUN Assay Kitに同梱されているプロトコールに、qPCRとNG-seqそれぞれについてSpike-In DNAの詳細な使用法が記載されています。qPCRとNG-seqでは必要なSpike-In DNAの量が大きく異なりますので、プロトコールをよくご確認ください。
ヒストン修飾に対する抗体を用いた場合は、通常、150bp (モノヌクレオソームのサイズ) と、ヌクレオソーム・ラダーに相当するサイズ (150、300、450、600bp) のDNA断片がみられます。転写因子に対する抗体のを使用した場合には、様々なサイズのDNA断片がみられ、その大半は35 bp以上です (図参照)。
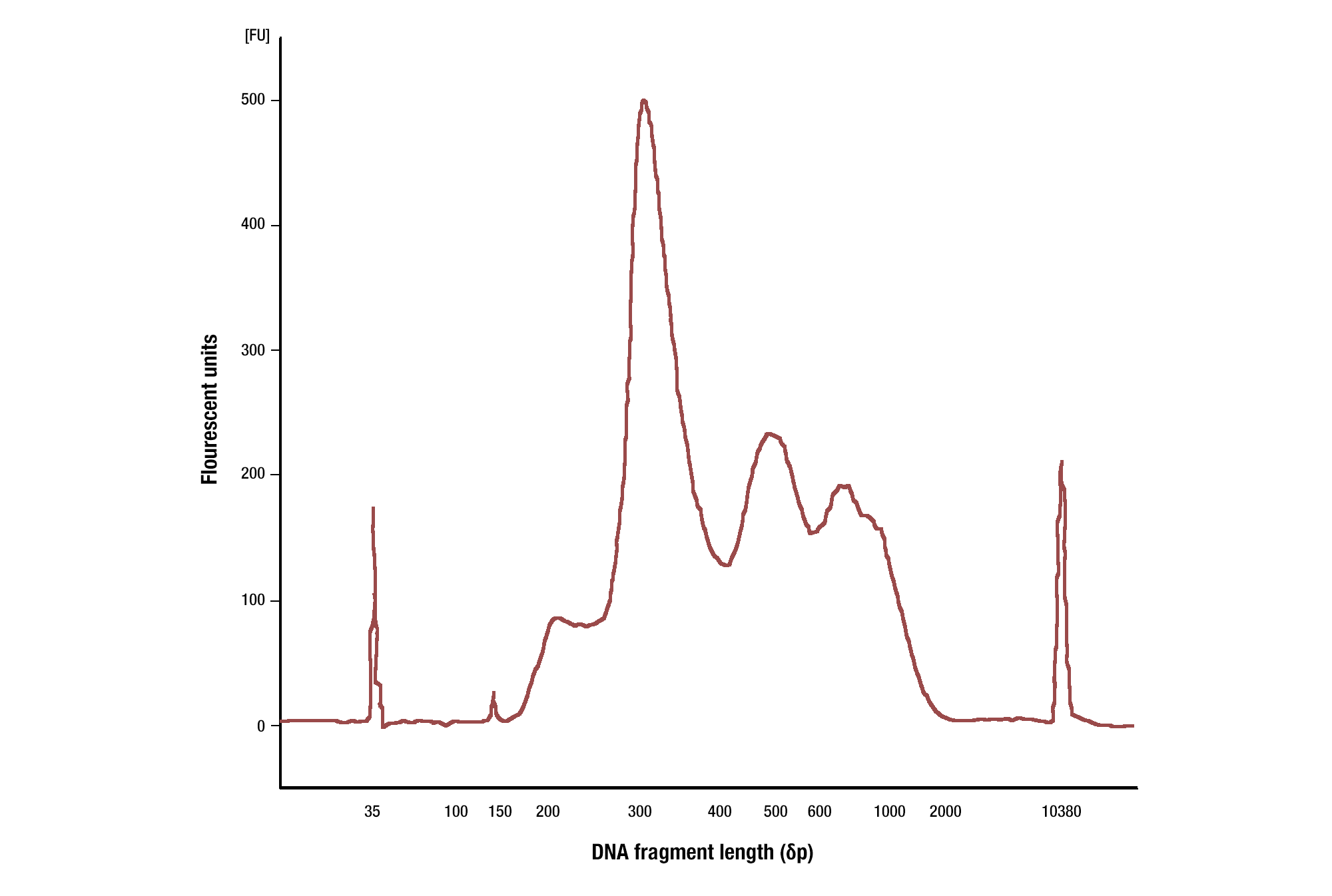
標的:H3K4me3: Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751によるHCT116細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。ライブラリーのDNA断片のサイズを、Agilent Bioanalyzerを用いて解析しました。ライブラリーの構築で付加されたアダプター配列とバーコード配列の断片の長さは140 bpです。こちらに示したように、切り出されたDNAにはモノヌクレオソーム単位の断片が高度に濃縮されています (ピークは約300 bp)。
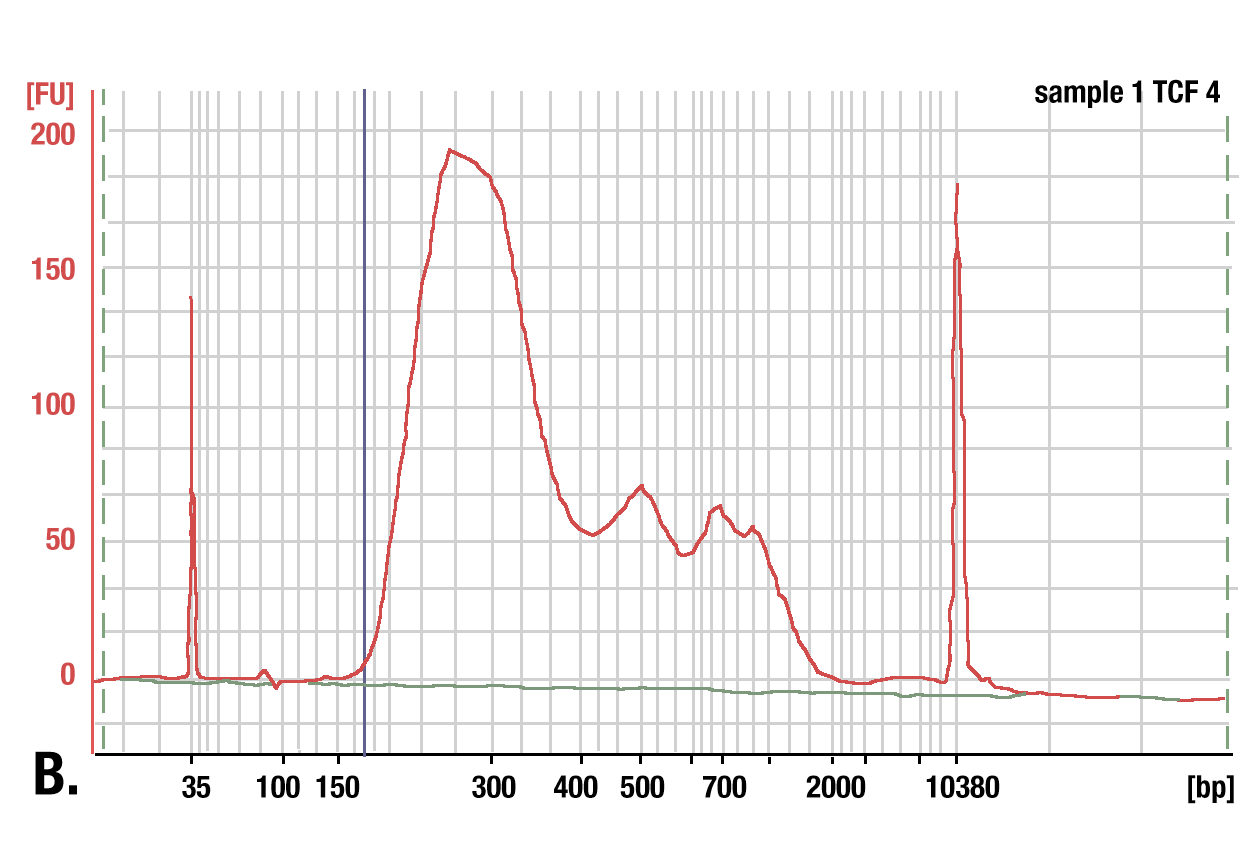
標的:TCF4/TCF7L2: TCF4/TCF7L2 (C48H11) Rabbit mAb #2569を用いCUT&RUNアッセイで得られたDNAを、フェノール/クロロホルム抽出とエタノール沈殿で精製しました。ライブラリーのDNA断片のサイズを、Bioanalyzer (Agilent Technologies) を用いて解析しました。ライブラリーの構築で付加されたアダプター配列とバーコード配列の断片の長さは140 bpです。
CUT&RUNで得られたDNAの精製には、CSTのDNA Purification Buffers and Spin Columns (ChIP & CUT&RUN) #14209の使用を推奨します。このスピンカラムを用いることで、CUT&RUNで得られたDNAを簡便かつ経済的に精製できます。対照比較実験で、CSTのDNAスピンカラムは35 bp以上のDNA断片を効率的に回収できることが分かっています。フェノール/クロロホルム抽出とエタノール沈殿で精製した場合は、より小さなDNA断片をより効率的に回収できますが、これらのサンプルから調製したDNAライブラリーを解析すると、CUT&RUNで得られるDNA断片全体の98%をDNAカラムで回収できることが分かりました。フェノール/クロロホルムで抽出しエタノールで沈殿させたサンプルに含まれるDNA断片のうち、サイズが35bp以下のものは約2%でした。したがってCSTのDNAスピンカラムは、CUT&RUNで得られるDNA断片を精製する簡便かつ経済的で、頑強な方法と言えます。すべてのスピンカラムで同様のサイズレンジのDNA断片を回収できるわけではないので、他社のDNAスピンカラムを使用する場合は、そのカラムで保持できるDNA断片の最小のサイズを確認してください。断片サイズのカットオフが大きい場合は、結果に悪影響を与える可能性があります (図参照)。
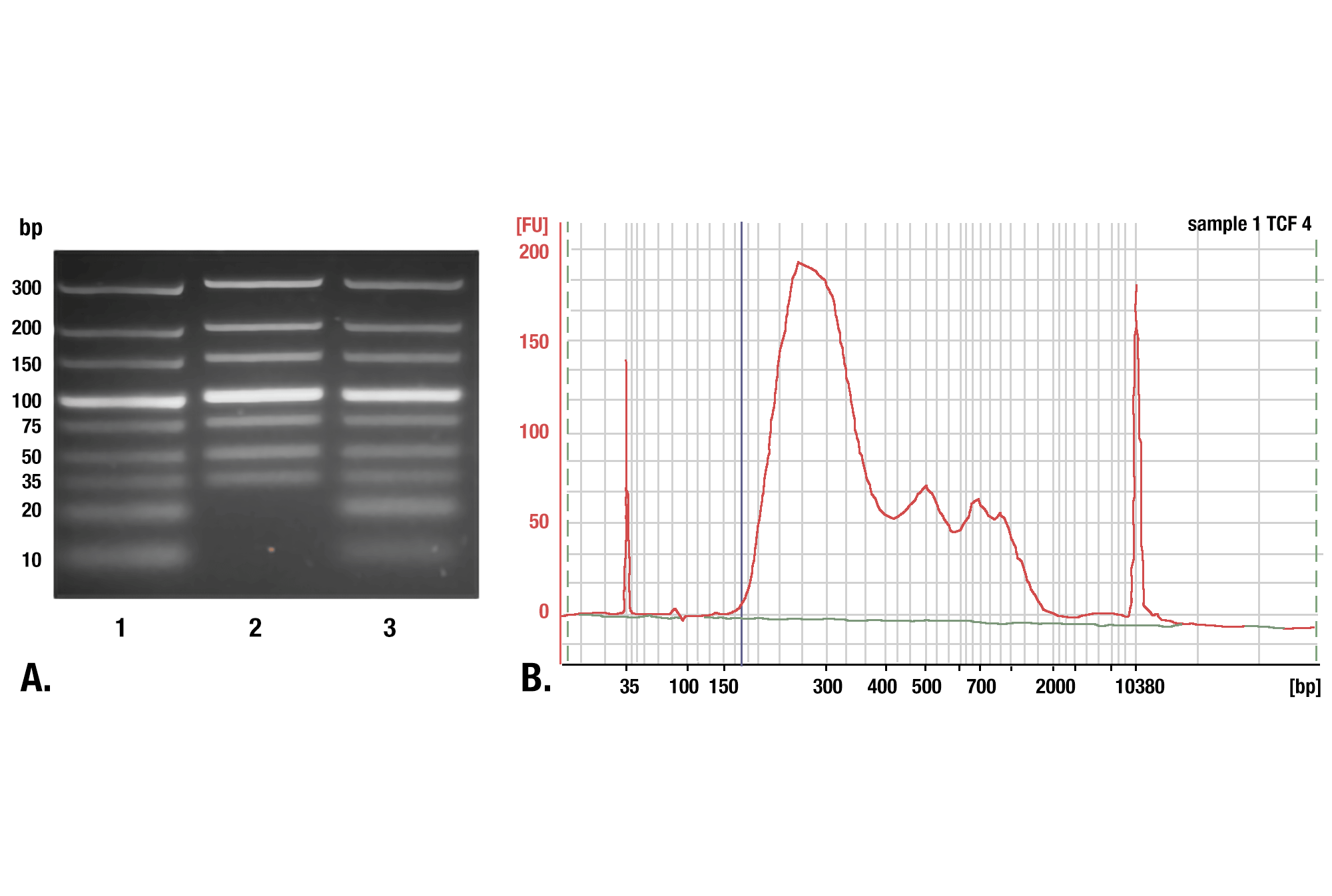
DNA精製におけるフェノール/クロロホルム抽出とスピンカラムの比較 DNA精製にスピンカラムを用いた場合と、フェノール/クロロホルム抽出とエタノール沈殿を用いた場合を比較しました。(A) 精製l前のlow range DNA ladder mix (Lane 1) と、DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN) #14209で精製したもの (Lane 2)、フェノール/クロロホルム抽出とエタノール沈殿で精製したもの (Lane 3) を、4%アガロースゲル電気泳動で分離しました。こちらに示したように、フェノール/クロロホルム抽出とエタノール沈殿は全てのサイズのDNA断片を効率的に回収しますが、DNAスピンカラムは35 bp以上のDNA断片を回収します。(B) TCF4/TCF7L2 (C48H11) Rabbit mAb #2569を用いたCUT&RUNアッセイで得られたDNAを、フェノール/クロロホルム抽出とエタノール沈殿で精製しました。ライブラリーのDNA断片のサイズを、Bioanalyzer (Agilent Technologies) を用いて解析しました。ライブラリーの構築で付加されたアダプター配列とバーコード配列の断片の長さは140 bpです。したがって、35 bpのDNA断片から始めると、ライブラリーの調製後には175 bpになります (図では青の垂直線で表示)。これにより、175 bp以下 (開始長は35bp以下) の断片はCUT&RUNにより濃縮された全DNA断片の2%以下であり、スピンカラムを用いたDNA精製は、CUT&RUNにより得られた全DNA断片の98%を得ることができることが分かります。
CSTのCUT&RUN Assay Kitは、下流のqPCR解析に適合します。キットのプロトコールには、CUT&RUNで得られたDNAをqPCRで定量する際に用いる、Inputの調製法とそれに必要な試薬が含まれています。CUT&RUNで得られるDNAをqPCRで解析する場合は、増幅するDNAの長さが60bpから80bpになるようにプライマーを設計することを推奨しています。
NG-seqの前に濃縮されたDNAをqPCRで解析することで、CUT&RUNのアッセイの品質を確認することができます。CSTのCUT&RUN Assay Kitは、下流のqPCR解析に適合します。
CUT&RUNの後にqPCRで解析する場合、正常IgGで濃縮されたサンプルをネガティブコントロールとして用い、標的に特異的な抗体でによる濃縮が、IgGバックグラウンドの何倍であるかを計算することもできます。しかし、通常はIgGサンプルに含まれるDNAはごく微量であり、また、使用した標的に特異的な抗体がアッセイでどの程度効率的に機能したかを示す良い指標にはなりません。この場合、インプットクロマチンDNAサンプルの作製と使用を推奨します。インプットクロマチンDNAサンプルの作製方法は、弊社のCUT&RUNキットプロトコールのセクションVをご覧ください。こうすることで、標的に特異的な抗体で濃縮されたDNAを総Input DNAのパーセントで表現し、標的に特異的な抗体がアッセイでどの程度効率的に機能したかを直接的に判断することができます。
BioanalyzerでDNA断片のサイズを解析する場合、最適の結果を得るには5-10 ngのDNAが必要になります。しかし、CUT&RUN実験が成功した場合でも、開始細胞から得られるDNAの収量が1 ng未満になることは少なくありません。特に転写因子やコファアクターの解析では、このように低収量になることがよくあります。CSTはこのように低収量な場合でも、堅牢なシーケンシングデータが得られることを確認しています。このような場合は、そのままDNAシーケンシングライブラリーの調製を進め、得られたDNAライブラリー (通常、濃度は10 - 40 ng/μLとなります) を用いて解析することをお勧めします。NG-seqの前にCUT&RUN実験で得られたDNAのQCを行う場合は、DNA収量に大きく依存する方法ではなく、qPCRで既知の標的遺伝子の濃縮をいくつか解析することをお勧めします。
これは、DNAライブラリー調製キットの感度に依存します。CUT&RUNで得られるDNAの反応ごとの典型的な収量は0.6から6 ngです。CSTのDNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795は、わずか0.5 ngのDNAからライブラリーを調製できます。ライブラリー調製に用いるDNA量が少ない場合は、ライブラリーを増幅するPCRのサイクル数を最大20サイクルまで増やすこともできます。
CUT&RUNやChIPにより得られるDNAのライブラリー調製には、CSTのDNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795の使用を推奨します。このキットは、弊社のMultiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538または#Multiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) #29580と組み合わせてお使いください。
いいえ、CUT&RUNやChIP-seqのライブラリー調製の際、DNAのサイズセレクションを推奨していません。CSTの経験とお客様からいただいたフィードバックを元に検証した結果、サイズセレクションによりライブラリーの多様性と収量が大きく損なわれることが分かっています。高分子DNA (> 1 kb) が気になる場合は、ライブラリー調製の際、アダプターを結合したDNAサンプルの増幅ステップでの伸長時間を短くしてください。伸長時間を10から15秒短縮するだけで、1 kb以上の大きなDNA断片の増幅を大きく低減し、CUT&RUNライブラリー調製中の必要なより小さいDNA断片の増幅には十分です。
CUT&RUNの後にNG-seqで解析を行う場合は、正常IgGで濃縮されたサンプルをネガティブコントロールとして使用することもできます。しかし、正常IgGから調製したDNAライブラリーの多様性は非常に低く、一見、CUT&RUNアッセイでゲノム上の特定の領域が濃縮されたように見えるため、NG-seqのデータ解析が複雑になるという情報を、外部の科学者から得ています。このため、CUT&RUNキットプロトコールのセクションVに記載されている通り、インプットクロマチンDNAサンプルの作製と使用を推奨しています。このInput DNAサンプルから調製したDNAライブラリーの多様性は高く、NG-seq解析により適したネガティブコントロールとして機能します。
ソニケーターを利用せずにNG-seq解析を行いたい場合は、CUT&RUN Input DNA by MNase protocolを用いた消化を行なってください。方法はプロトコールのページをご覧ください。ソニケーターを利用できない場合、qPCR解析にはインプットDNAを断片化せずに使用することができますが、断片化していないインプットDNAはサイズが大きすぎてDNAスピンカラムで精製できないので、フェノール/クロロホルム抽出とエタノール沈殿で精製する必要があります。酵素的消化したインプットDNAをqPCR解析に用いることもできます。
こちらはCSTで何百ものCUT&RUNサンプルを解析した結果に基づいたものです。シーケンシング深度が300万以下の場合、得られるピークの数は少なすぎ、シーケンシング深度を増加させるにつれてピーク数も増加します。必要に応じて、サンプルあたりのシーケンシング深度を1,000万リードまで増やすことができます。シーケンシング深度が1サンプル当たり1,500万以上の場合、リードの重複率が大幅に上昇します (図参照)。
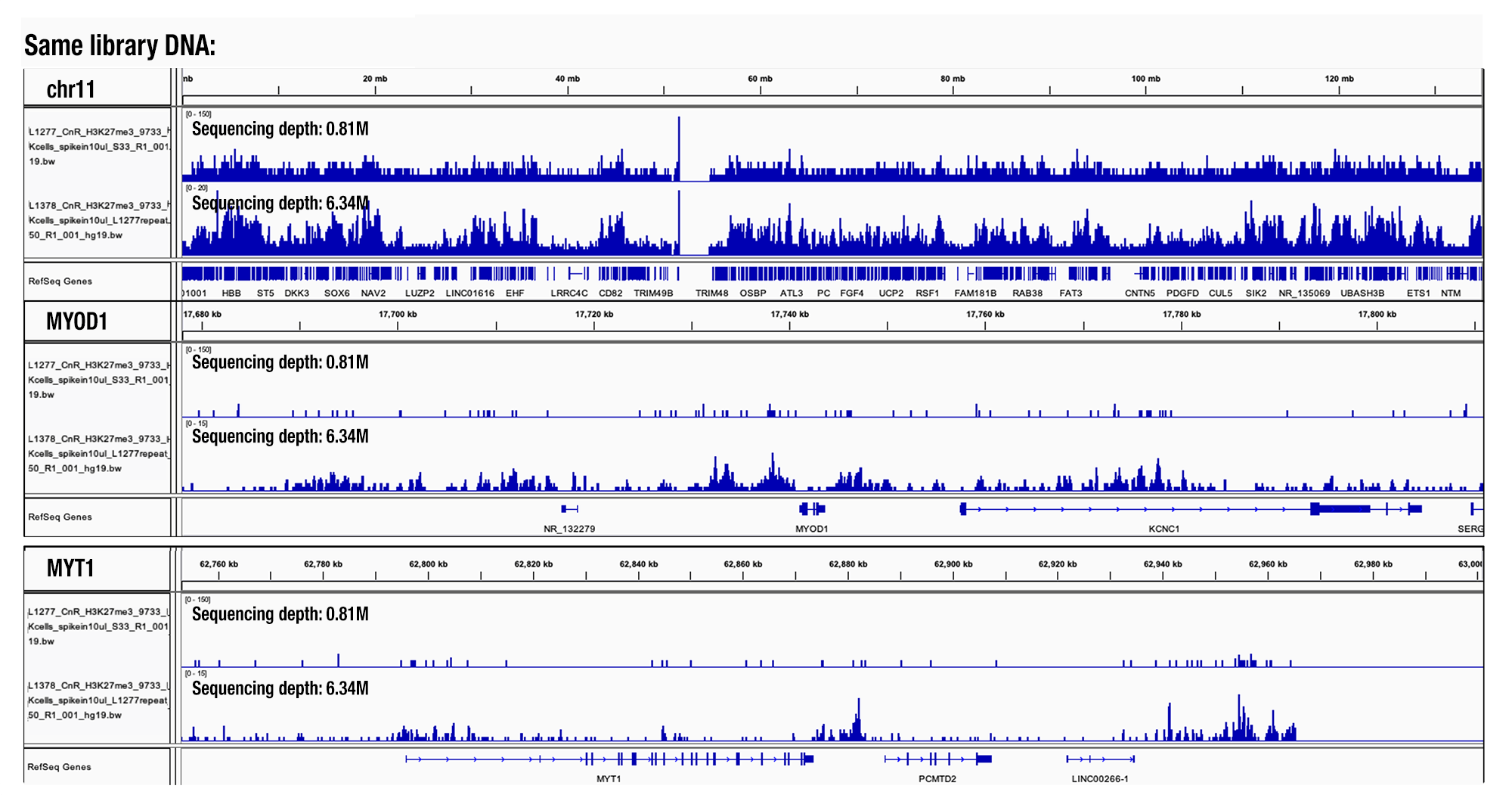
シーケンシング深度の比較 (標的:H3K27me3): CUT&RUN Assay Kit #86652を用い、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733によるHeLa細胞のCUT&RUNを行いました。DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795を用いて、DNAライブラリーを調製しました。同じライブラリーDNAを、図中に示した異なるシーケンシング深度でNGS解析しました。図は、11番染色体全体 (上トラック)、MYOD1遺伝子 (中トラック)、MYT1遺伝子 (下トラック) への結合を示しています。
ご不明点は、CSTのエピジェネティックスアプリケーショングループ [保護されたメール] まで直接お問い合わせください。
Christopher Fry, PhD
エピジェネティクス製品開発部長
Fang Chen, PhD
エピジェネティクスアプリケーションチーム グループリーダー
更新:2021年10月