View in English?
View in English?
社内検証では、複数のヒストン修飾、転写因子、コファクターにおいて、1アッセイにつき100,000個の細胞を用いることで標的遺伝子座を濃縮できることが分かっています。しかし、ビーズや抗体、酵素、バッファーをスケールアップすることなく、1アッセイにおける細胞数を250,000個まで増やすことができます。
CSTのCUT&Tag Assay Kit #77552では、わずか5,000 - 10,000個の細胞でヒストン修飾を、20,000個の細胞で転写因子やコファクターを解析できることが分かっています (下図を参照してください)。
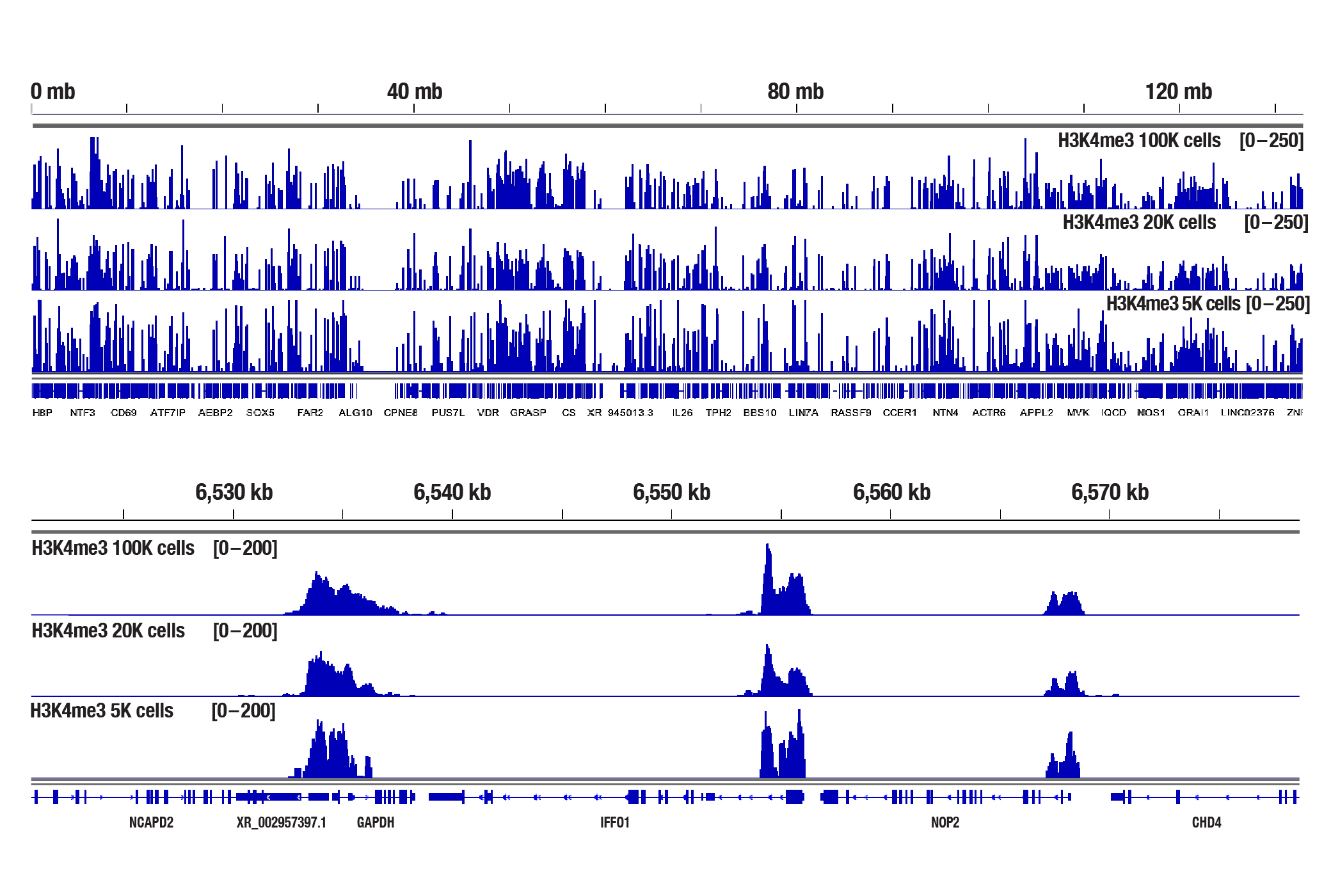
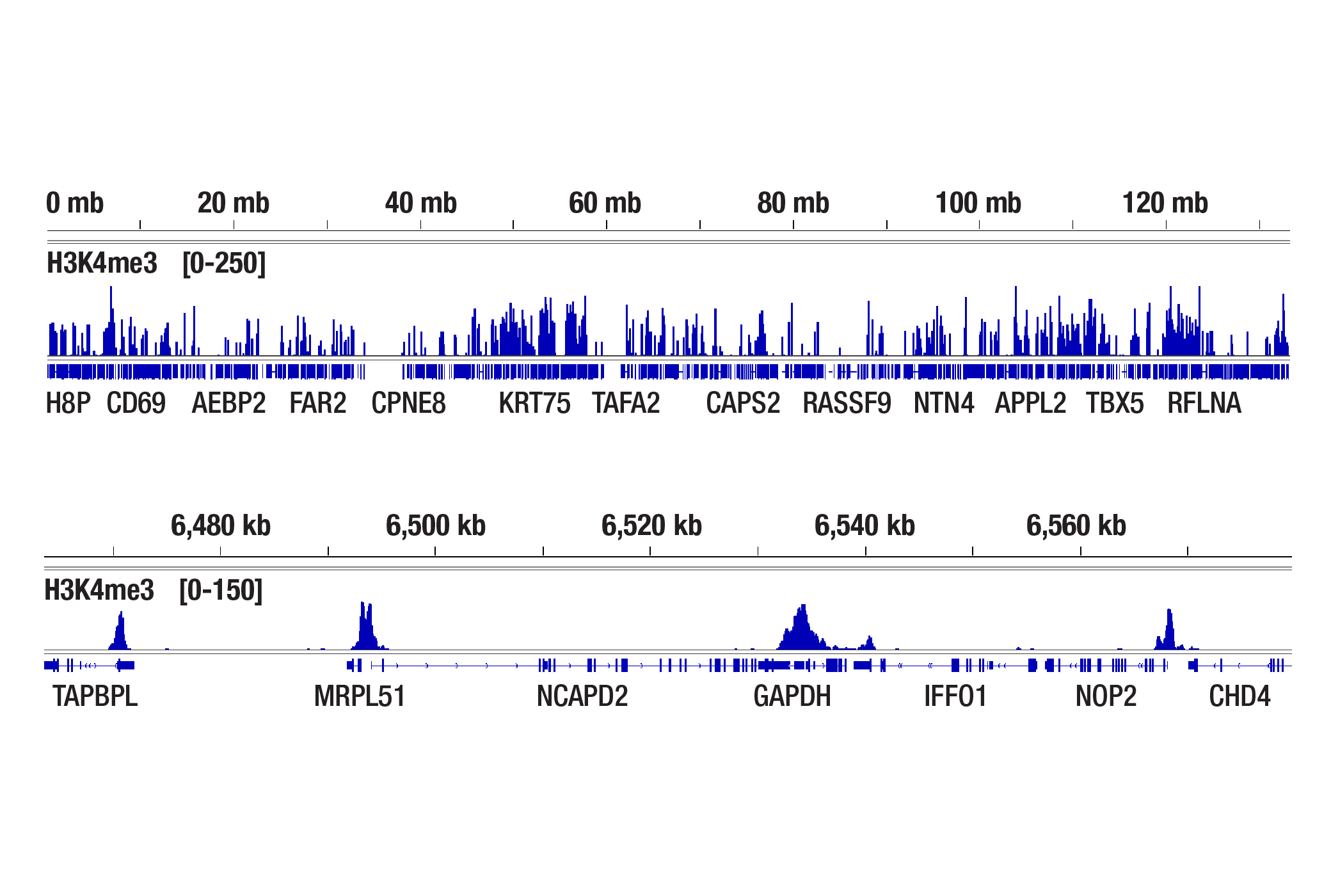
NCCIT細胞を、CUT&Tag Assay Kit #77552とTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751を用いてCUT&Tagアッセイで解析しました。アッセイには100,000個、20,000個、5,000個の細胞を使用しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。12番染色体全体にわたるH3K4me3の濃縮の比較 (上パネル) と、H3K4me3の既知の標的遺伝子であるGAPDHの濃縮の比較 (下パネル) を示しています。
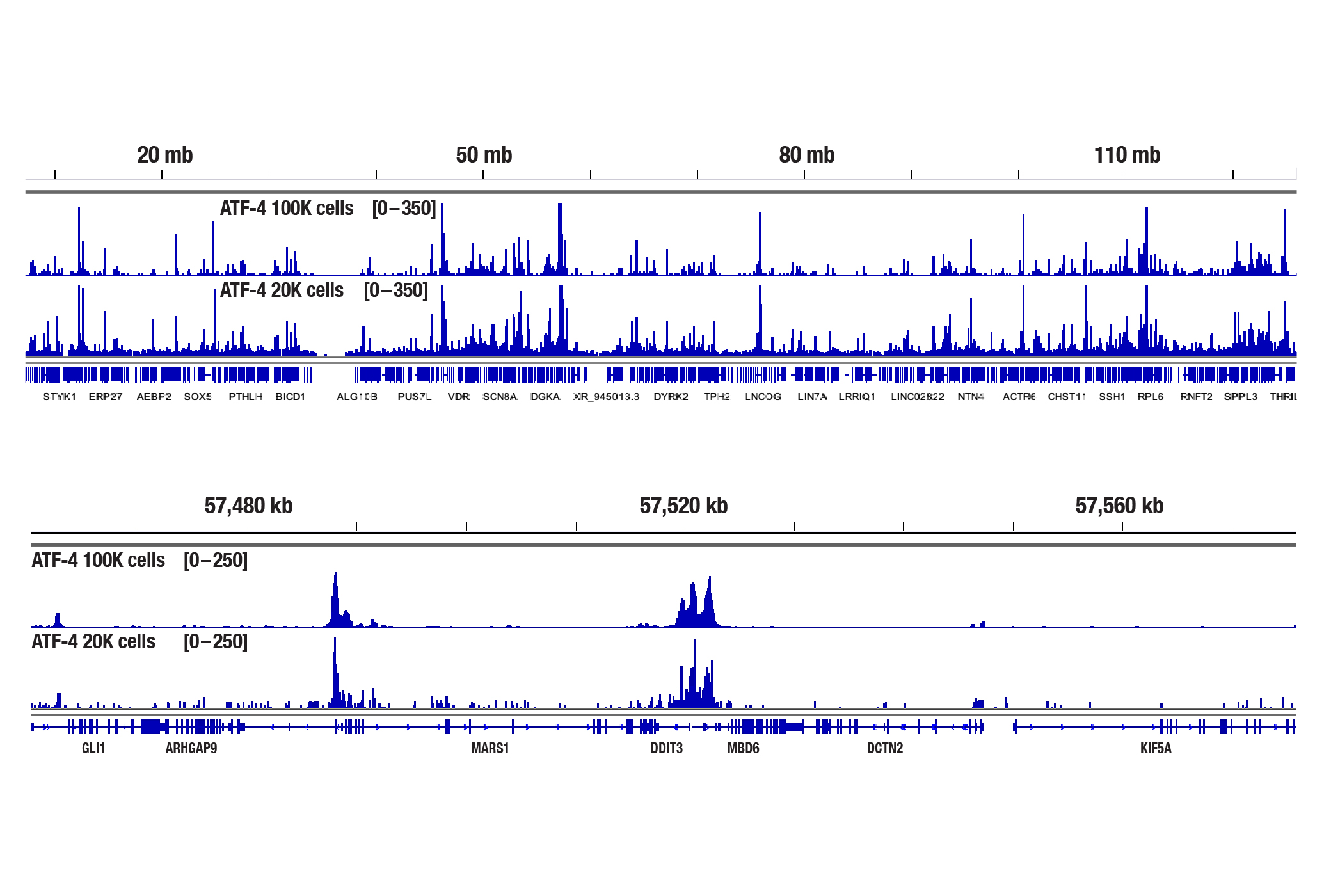
100,000個または20,000個のHep G2細胞をThapsigargin #12758 (300 nM) で4時間処理し、ATF-4 (D4B8) Rabbit mAb #11815とCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。12番染色体全体にわたるATF-4の濃縮の比較 (上パネル) と、ATF-4の既知の標的遺伝子であるDDIT3/CHOPの濃縮の比較 (下パネル) を示しています。
ヒトとマウスの数十種類の接着細胞株および浮遊細胞株を用いてCUT&Tagを行いました。現在のところ、異なる細胞株の間で、アッセイの効率に大きな違いはみられません。
接着細胞株の場合は、まず細胞をディッシュから剥離する必要があります。細胞の剥離には、トリプシンの使用を推奨しています。Accutaseも使用可能ですが、細胞ペレットの粘着性が弱くなるために、洗浄のステップで細胞を失いやすくなります。浮遊細胞の場合はより簡単です。血球計算盤または自動細胞数計測器で細胞をカウントし、必要数の細胞を回収してください。接着細胞と浮遊細胞の間で、アッセイの効率に大きな違いはみらません。
はい。壊れやすい (実験中に溶解しやすい) 細胞を用いる場合や細胞の結合に用いるConcanavalin Aビーズが細胞のシグナル伝達経路に影響をおよぼす場合、将来的に使用する細胞ペレットを凍結保存したい場合などには、細胞の固定を推奨します。固定が必要な場合は、CUT&Tag Assay KitのプロトコールのAppendix Aを参照して、CUT&Tag実験の前に細胞を軽く固定してください。CSTの社内試験では、固定した細胞より生細胞を用いた方がCUT&Tagで良好な結果が得られているため (下図を参照してください)、上記のような固定が必要な場合を除き、できる限り生細胞を使用することを推奨します。
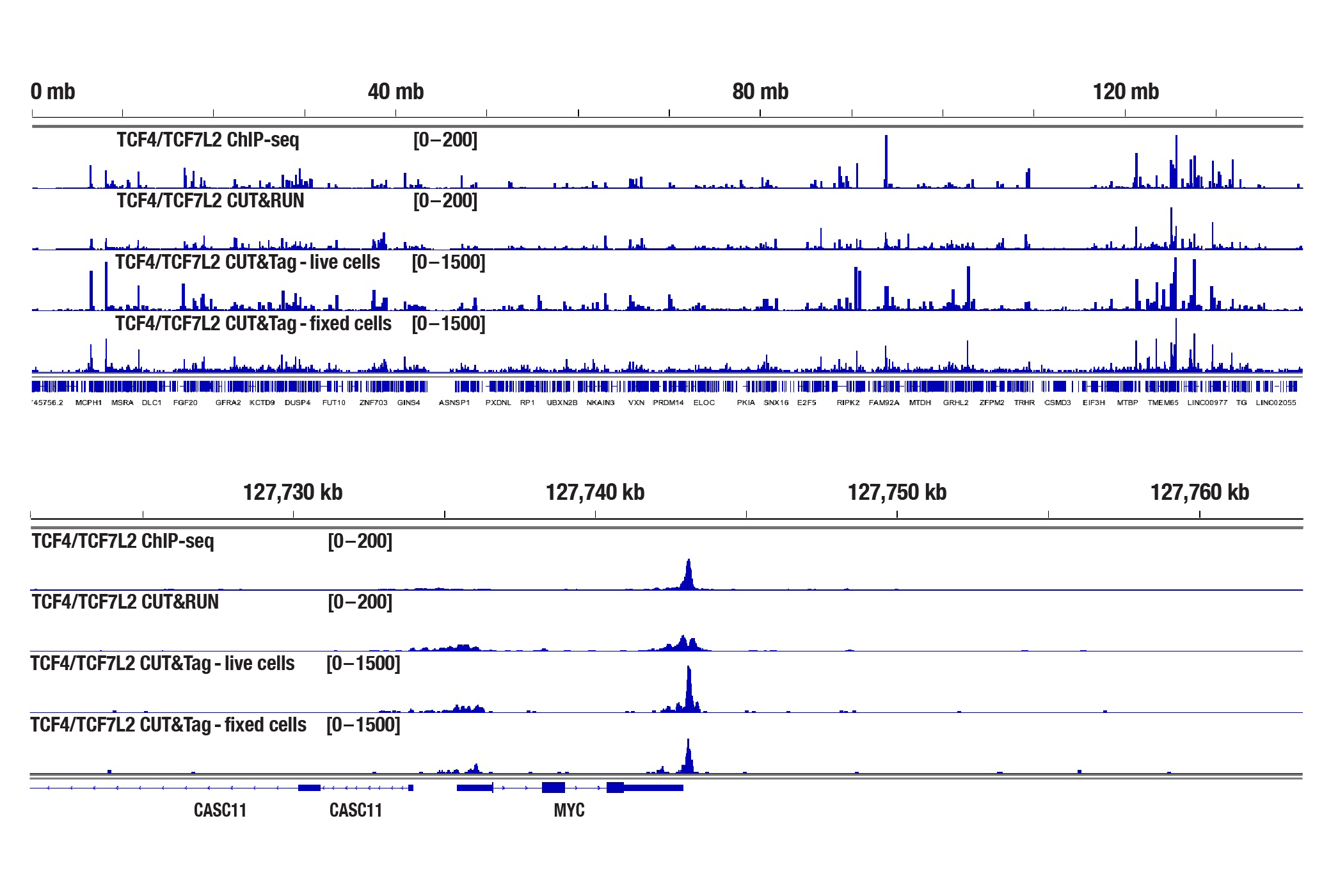
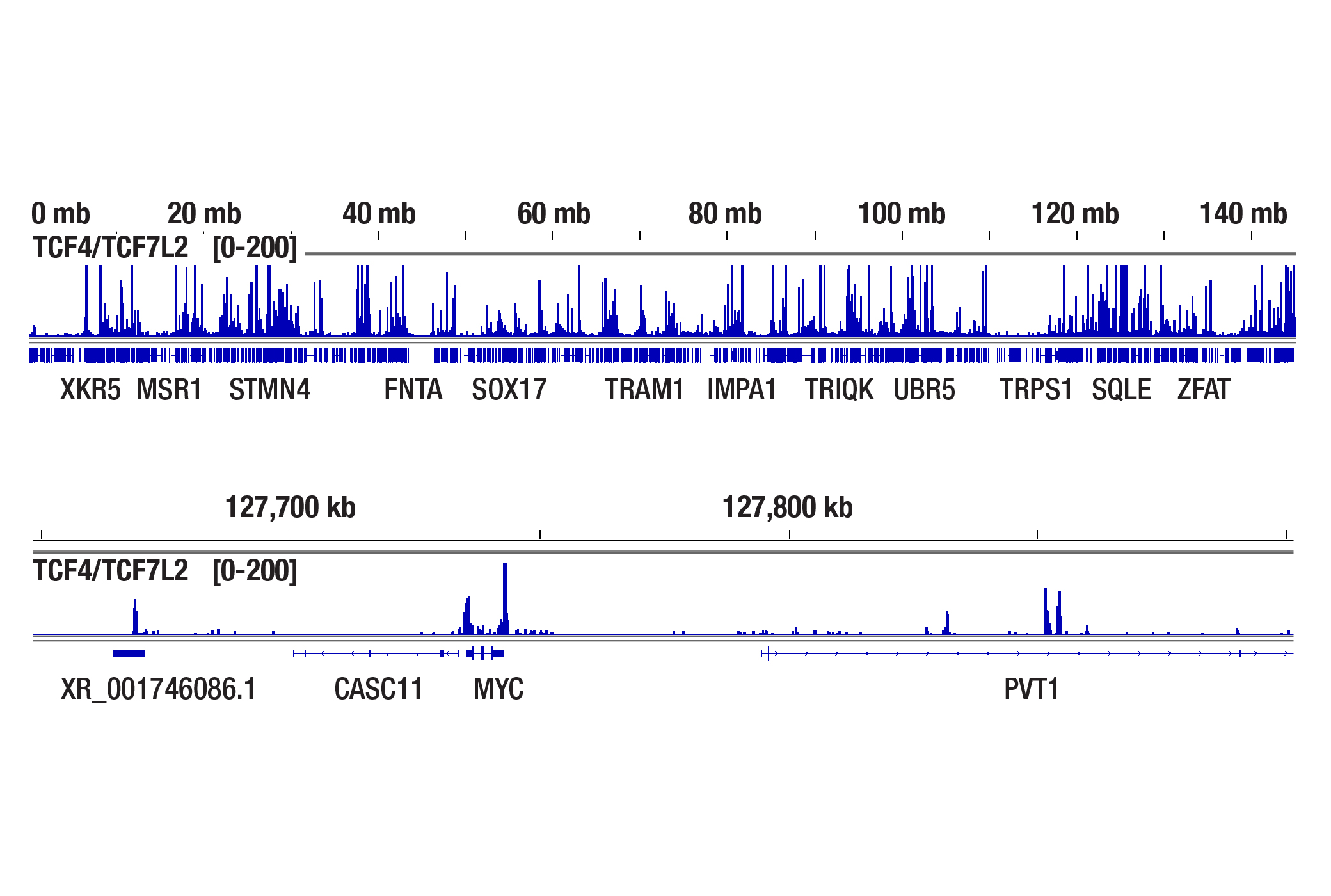
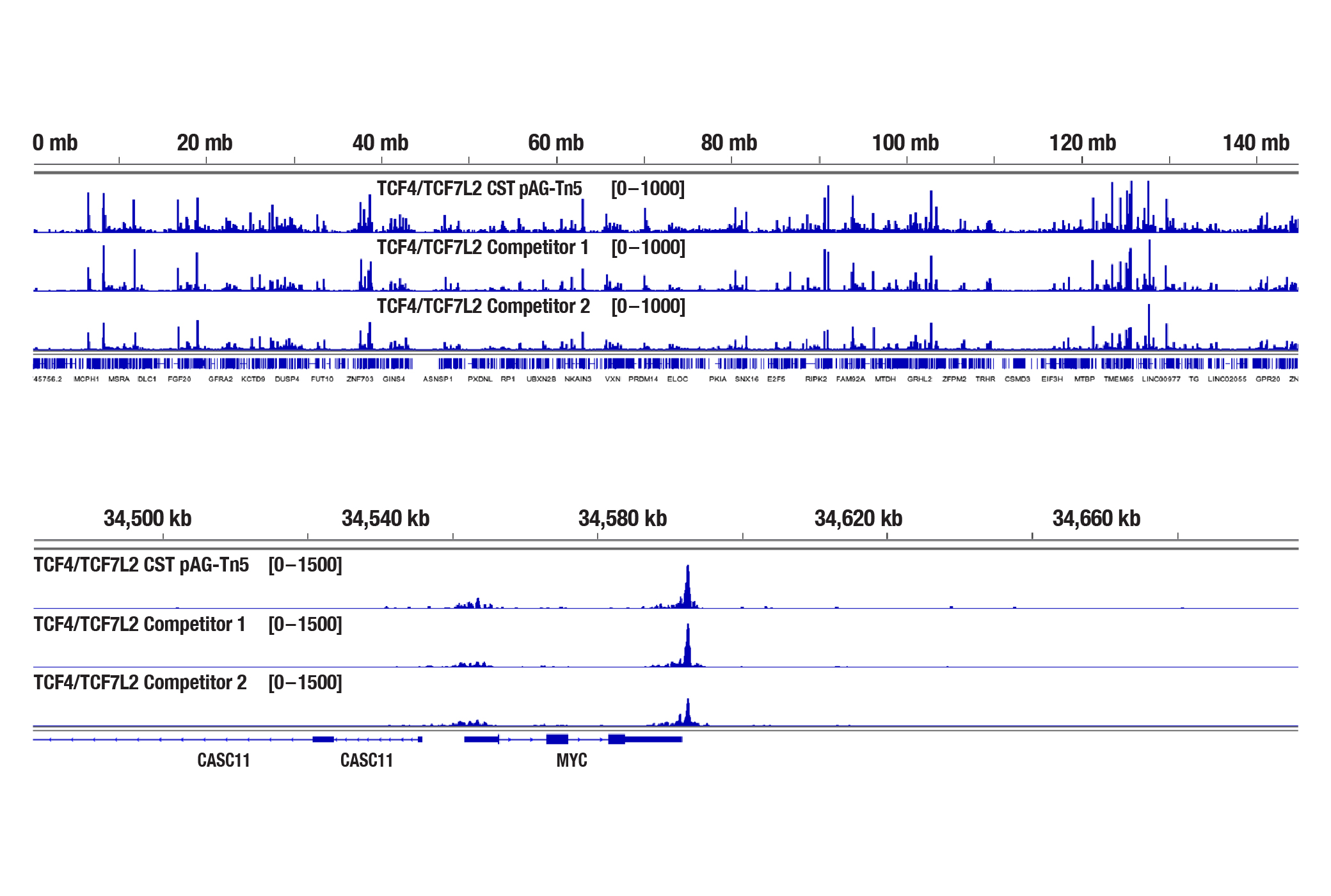
HCT 116細胞を、TCF4/TCF7L2 (C48H11) Rabbit mAb #2569を用いてChIP-seq、CUT&RUN、CUT&Tagの各アッセイを行いました。各アッセイは、それぞれSimpleChIP® Plus Enzymatic Chromatin IP Kit (Magnetic Beads) #9005、CUT&RUN Assay Kit #86652、CUT&Tag Assay Kit #77552を用いています。CUT&Tagアッセイでは、0.1%ホルムアルデヒドで2分間細胞を固定しました。ChIP-seqとCUT&RUNサンプルにはDNA Library Prep Kit for Illumina Systems (ChIP-seq, CUT&RUN) #56795、CUT&TagサンプルにはCUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いてDNAライブラリーを調製しました。図は、8番染色体全体にわたるTCF4/TCF7L2の濃縮 (上) と、TCF4/TCF7L2の既知の標的遺伝子であるMYCの濃縮 (下) を示しています。
はい。CSTのCUT&Tag Assay Kit #77552では、CUT&Tag 1反応あたりわずか1 mgのマウスの肝臓組織、脳組織、心臓組織で、ヒストンを標的とする解析が可能であることが分かっています (下図を参照してください)。ただし、組織内の転写因子やコファクターなどのヒストン以外の標的は、CUT&Tagアッセイでは十分に濃縮されていません。組織内の転写因子やコファクターの解析には、CUT&RUN Assay Kit #86652の使用を推奨します。通常、新鮮な組織では、固定組織と同等またはより強力なCUT&Tagシグナルが取得できます。固定が必要な場合は、CUT&Tag Assay KitのプロトコールのAppendix Bを参照して、CUT&Tag実験の前に細胞を軽く固定してください。固定した組織は、CUT&Tagアッセイで使用するまで最大6か月間凍結保存できます。
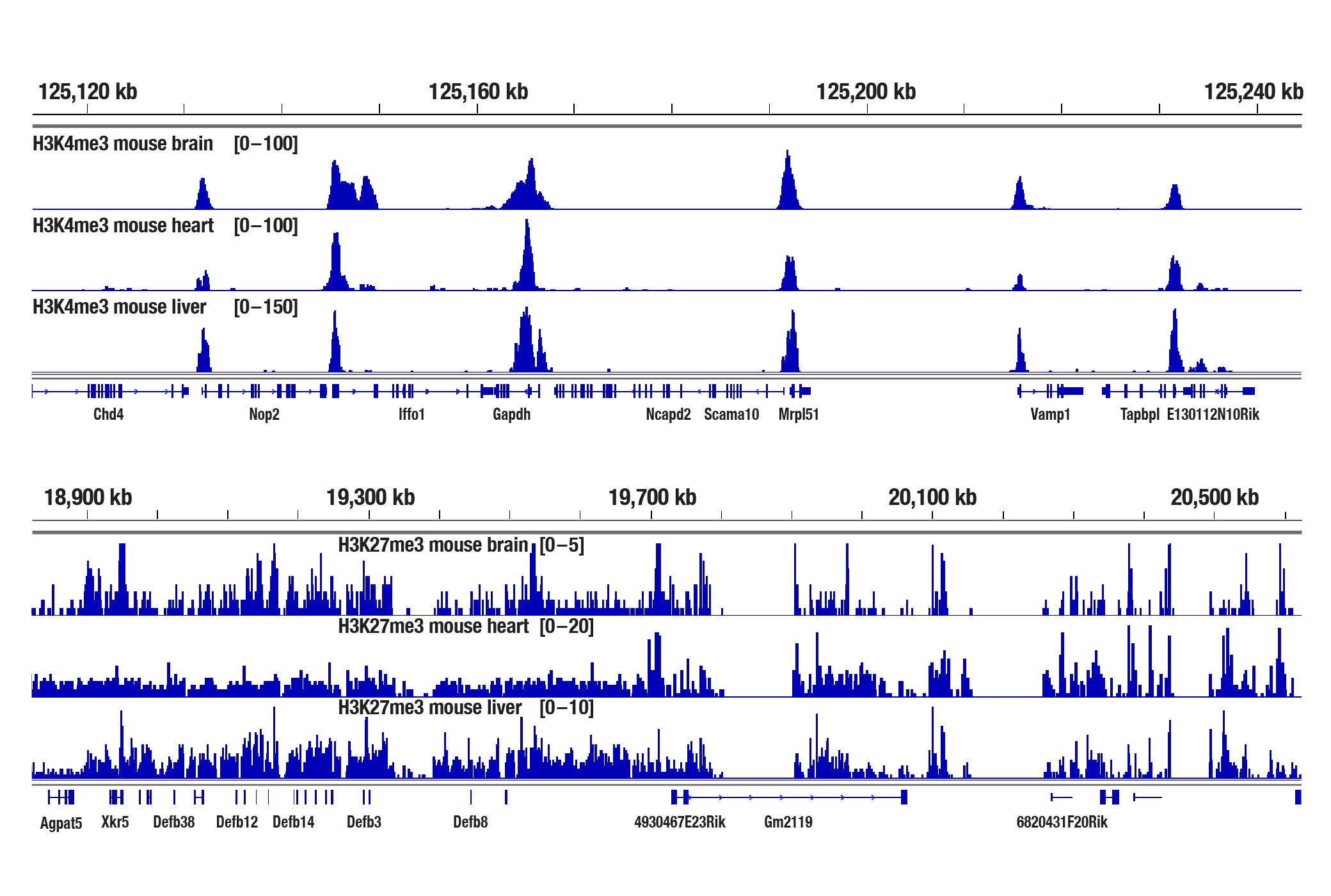
図に示すとおり、1 mgの新鮮なマウス脳組織、心臓組織、肝臓組織を、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはTri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagアッセイで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。図は、Defb遺伝子周囲のH3K27me3の濃縮 (上パネル) と、H3K4me3の既知の標的遺伝子であるGAPDHの濃縮 (下パネル) を示しています。
はい、CUT&Tag Assay Kitでは、瞬間凍結した組織サンプルまたは-80℃で直接凍結した組織サンプルが使用できます。解凍後に、組織サンプルを固定することもできます。固定により、CUT&Tagシグナルの強度が向上するという結果は得られていません (下図を参照してください)。
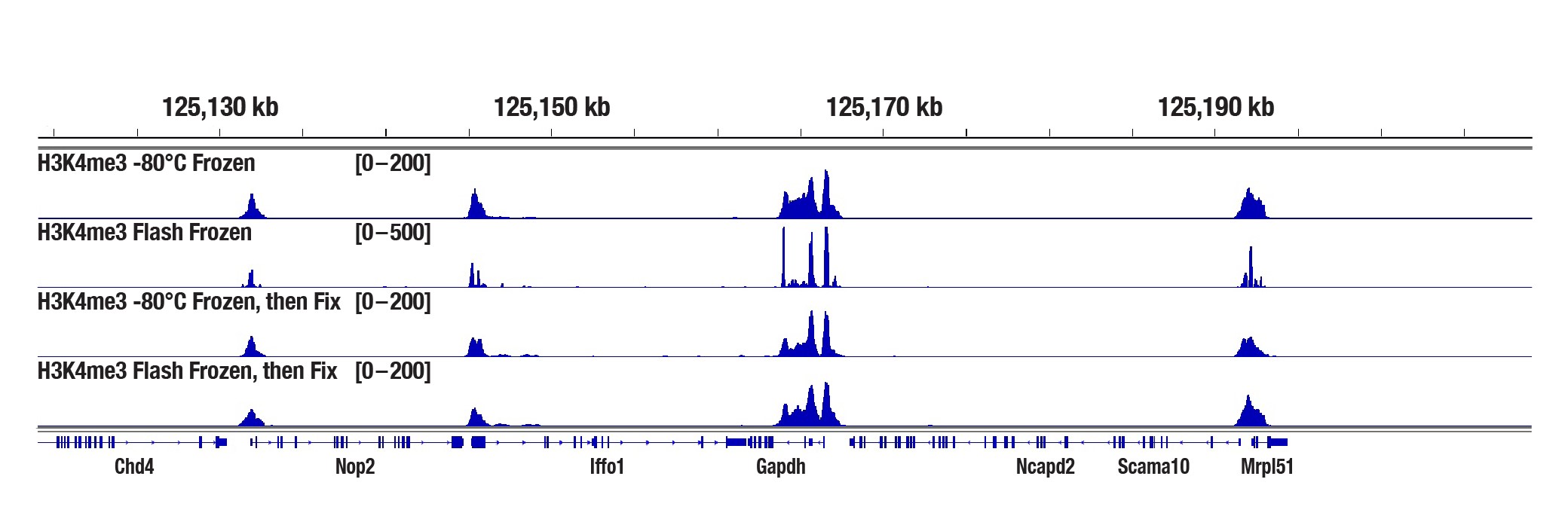
マウス肝臓組織を-80℃で凍結、または2分間液体窒素内で凍結させて-80℃で保存し (瞬間凍結)、図に示すとおり、固定しないまたは0.1%ホルムアルデヒドで10分間固定したサンプルを作成しました。2.5 mgのこれらの組織を、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751とCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。H3K4me3の既知の標的遺伝子であるGAPDH遺伝子全体にわたる結合をパネルに示しています。
Concanavalin Aビーズは、特に細胞が溶解された時に凝集しやすくなります。細胞の生存や増殖に異常が無いことを確認し、細胞洗浄の操作は穏やかに行ってください。細胞懸濁液とConcanavalin Aビーズの室温でのインキュベーション時間に注意し、5分間を超えないようにすることを推奨します。細胞とビーズの結合、抗体の結合、pAG-Tn5酵素の結合の各ステップ中におけるインキュベーション中は、チューブを回転または振盪させる代わりに、適切なインキュベーション温度でチューブを静置すると、ビーズの凝集を軽減できます。シーケンシング深度や得られるピーク数、結合パターンに悪影響を与えることはありません (下図を参照してください)。しかしながら、CSTの社内試験では、ビーズの凝集は最終的な結果に悪影響をおよぼさないことが分かっています。
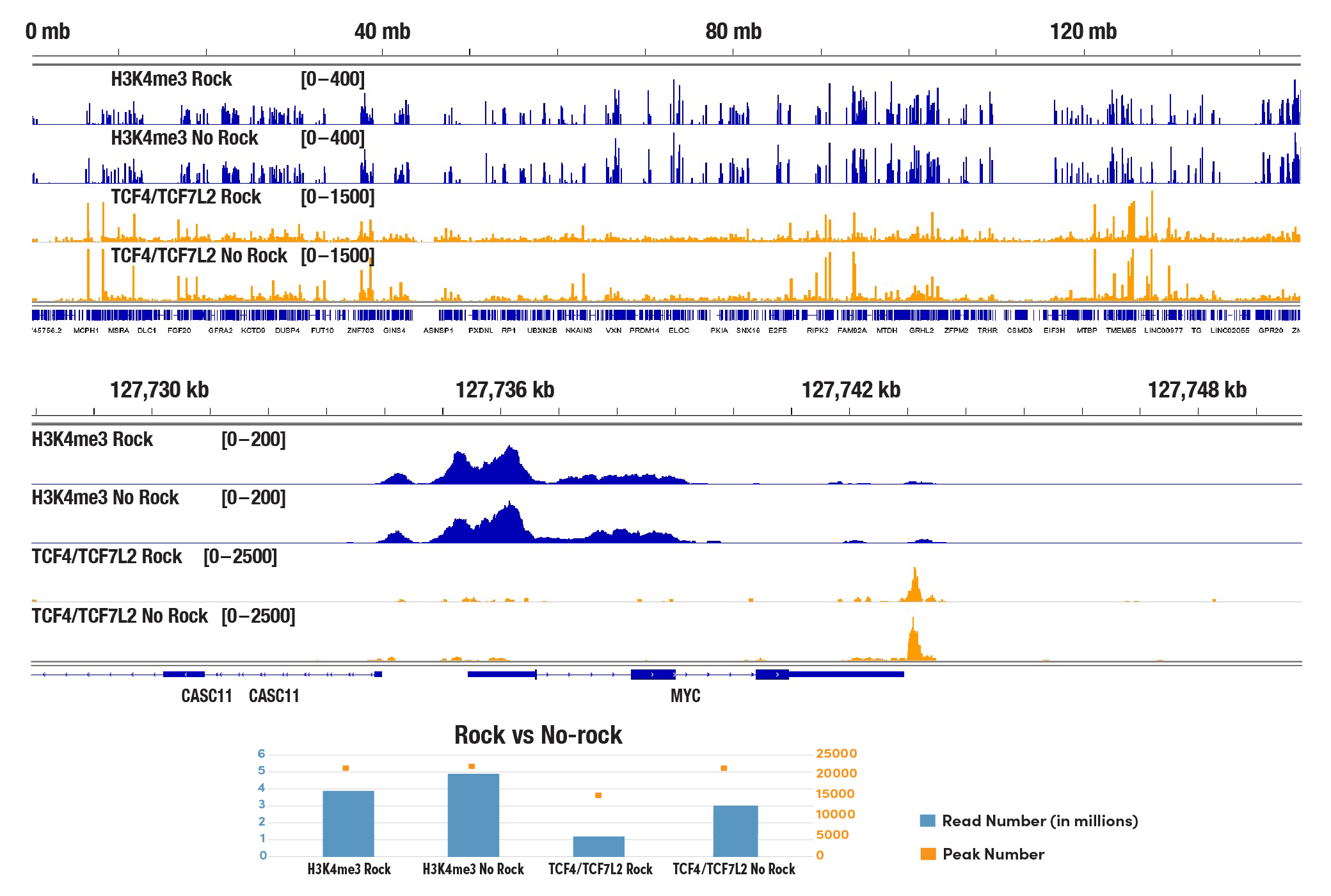
100,000個のHCT 116細胞を、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはTCF4/TCF7L2 (C48H11) Rabbit mAb #2569のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。細胞-ビーズの結合、抗体の結合、タグメンテーションの各ステップにおけるインキュベーション中に振盪したサンプルチューブ (Rock) と、これらのすべてのステップ中に適切な温度でチューブラック上に静置したサンプルチューブ (No-Rock) を用意しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。これら4つのサンプルをプールし、一緒にシーケンスしました。下のパネルは、各サンプルから取得されたリード数と、各データセットで特定されたピーク数を示しています。図は、8番染色体全体にわたるH3K4me3およびTCF4/TCF7L2の濃縮 (上パネル) と、H3K4me3およびTCF4/TCF7L2の既知の標的遺伝子であるMYCの濃縮 (下パネル) を示しています。
CSTが最適化済みの、CUT&Tagアッセイにおけるタグメンテーションの条件は、接着細胞、浮遊細胞を問わず様々な細胞株で機能します。50 μLの各タグメンテーション反応につき、2 μLのCUT&Tag pAG-Tn5 (Loaded) #79561の使用を推奨します。CSTの社内試験では、各反応で使用するpAG-Tn5を増やしても、リード数やピーク数、シグナル対ノイズ比の増加はみられませんでした (下図を参照してください)。実際、CUT&Tagアッセイの成功には、使用するpAG-Tn5の量よりもpAG-Tn5の活性の度合いが重要です。pAG-Tn5酵素は、-20℃で適切に保存し、時間の経過とともに活性が低下するため使用期限の切れたpAG-Tn5は使用しないでください。CUT&Tag pAG-Tn5 (Loaded) #79561の使用期限は6か月です。また、効率的な標的に特異的なタグメンテーションを確保し、オープンクロマチン領域での非特異的なタグメンテーションを制限するために、タグメンテーションバッファーの塩濃度を最適化しています。
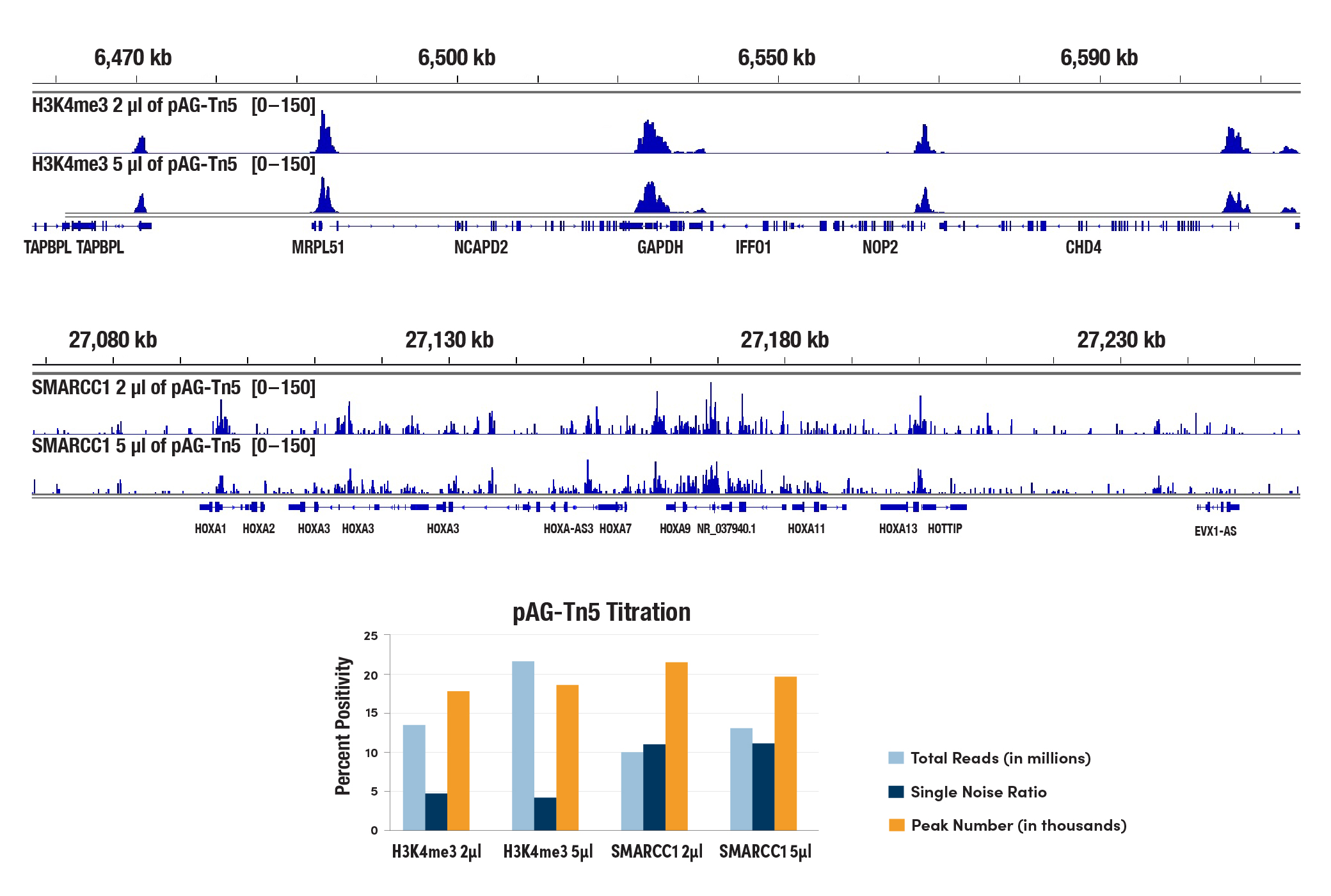
100,000個のHCT 116細胞を、2 µLあるいは5 µLのpAG-Tn5 (Loaded) #79561、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751またはSMARCC1/BAF155 (D7F8S) Rabbit mAb #11956のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。これらの4つのサンプルをプールし、一緒にシーケンスしました。下のグラフは、リード数やゲノム全体にわたるシグナル対ノイズ比、各サンプルで得られたピーク数が、異なる2つの量のpAG-Tn5間で同等であることを示しています。また、上部の図では、H3K4me3の既知の標的遺伝子であるGAPDH全体にわたるH3K4me3の結合 (上パネル) と、SMARCC1の既知の標的遺伝子であるHoxA遺伝子クラスターにわたるSMARCC1の結合 (下パネル) を示しています。
はい。ジギトニンによる膜の透過化は可逆的であるため、実験に用いるすべてのバッファーにジギトニンを加えておく必要があります。バッファーからジギトニンを除去したり、ジギトニン濃度が低下すると、抗体やpAG-Tn5の核への進入に影響をおよぼす可能性があります。細胞は、ジギトニンの処理時間よりも、ジギトニンの濃度に高い感受性を示します。推奨ジギトニン濃度で細胞の広範な溶解がみられる場合は、CUT&Tag Assay KitのプロトコールのAppendix Cの記載に従い、ジギトニン濃度を最適化してください。
CUT&Tag Assay Kitは全細胞で使用できるように最適化されており、接着細胞、浮遊細胞を問わず様々な細胞株で使用できます。Concanavalin Aビーズは、細胞表面と核表面の両方に存在する糖タンパク質に結合するため、弊社のキットは予め単離した核でも機能しますが、予め単離した核を使用することによりCUT&Tagシグナルが増加するという結果は得られていません (下図を参照してください)。細胞壁をもつ酵母や植物細胞など、ジギトニンで効率的に透過化できない細胞の場合のみ、予め核を単離する必要があります。
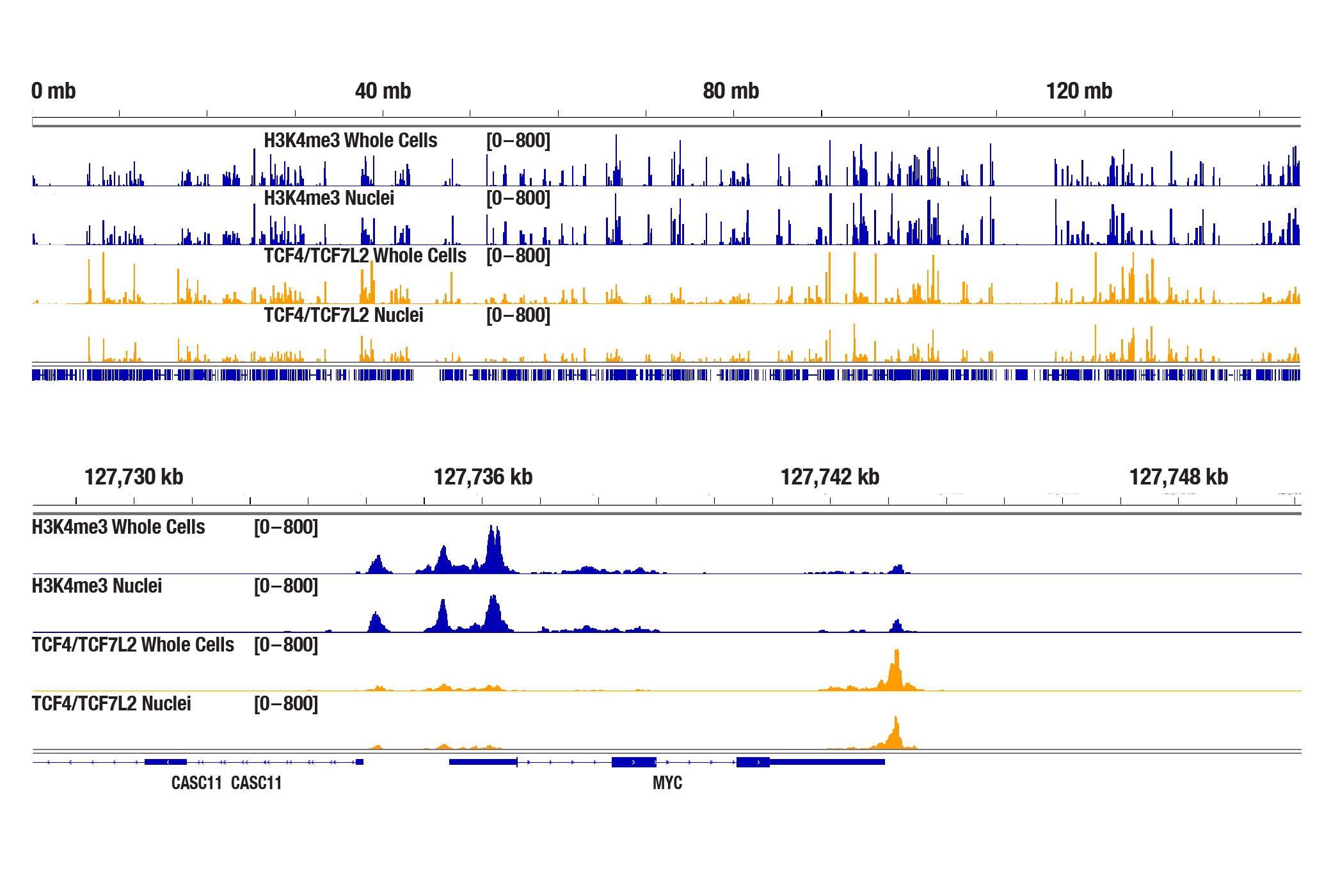
100,000個のHCT 116細胞の全細胞あるいは予め単離した核を、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb#9751またはTCF4/TCF7L2 (C48H11) Rabbit mAb #2569のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。図は、H3K4me3および8番染色体全体でのTCF4/TCF7L2の濃縮 (上) および、H3K4me3およびTCF4/TCF7L2の既知の標的遺伝子であるMYCの濃縮 (下) を示しています。
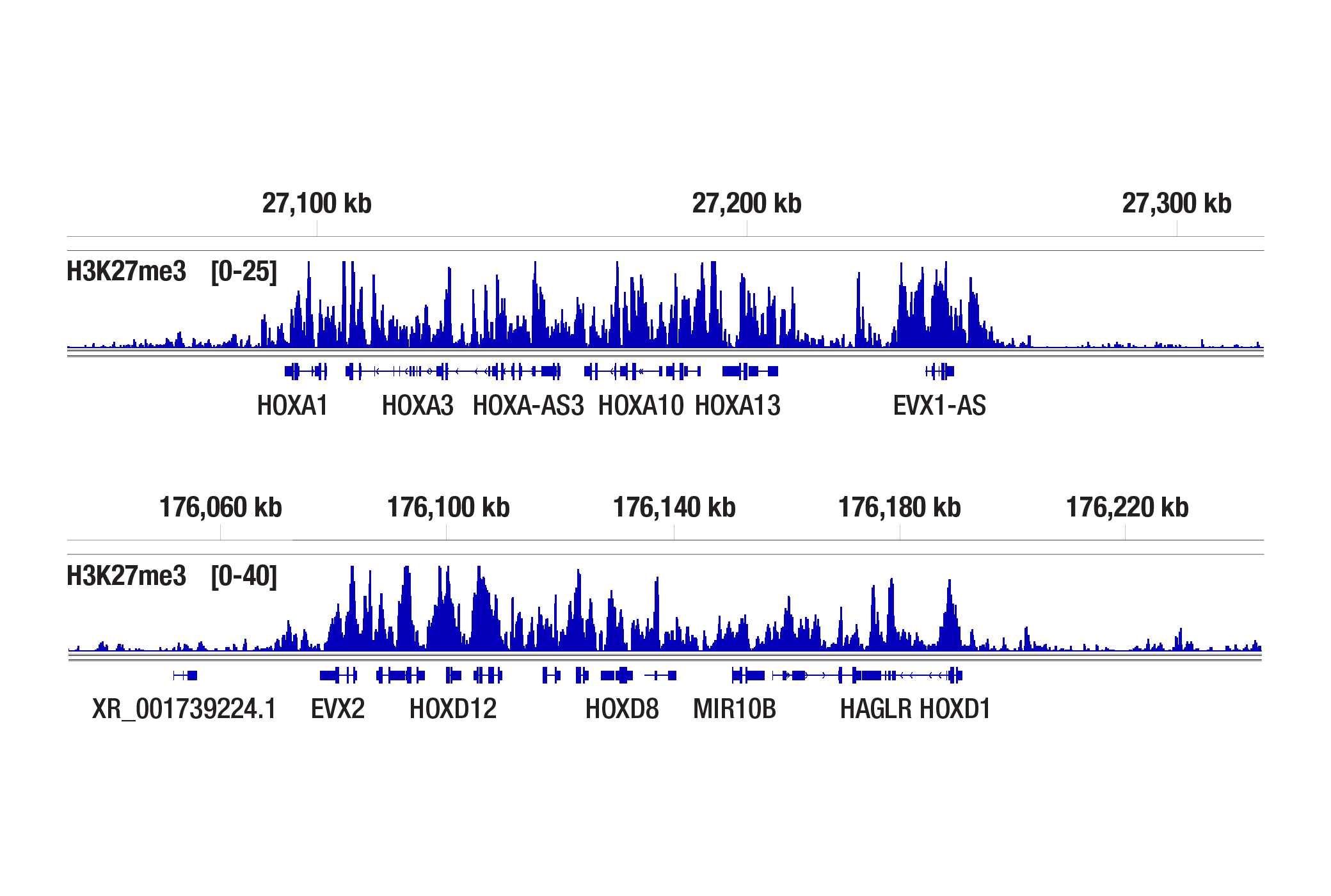
CSTの社内検証では、タグメンテーションバッファーの塩濃度が適切であれば、ユークロマチンやヘテロクロマチンに対するバイアスはみられません。CUT&Tagでは、標的に特異的な抗体および二次抗体がpAG-Tn5をリクルートし、ユークロマチン、ヘテロクロマチンを問わず、標的に特異的な抗体に直接隣接するクロマチンにタグメンテーションを行います。pAG-Tn5は、能動的にクロマチンに繋ぎ止められるため、アクセスが難しいヘテロクロマチン領域のタグメンテーションも可能です。CSTの科学者がCUT&Tagの抗体を検証する際に、オープンクロマチン領域における非特異的なタグメンテーションを観察した場合、その抗体はCUT&Tagアッセイでの使用は不合格となります。CSTのCUT&Tag Assay Kit #77552は、活性化しているアクセス可能なユークロマチンにおけるヒストン修飾 (Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751参照) や活性化している転写因子 (TCF4/TCF7L2 (C48H11) Rabbit mAb #2569参照) だけではなく、活性が抑制されているアクセスが難しいヘテロクロマチンにおけるヒストン修飾 (Tri-Methyl-Histone H3 (Lys27) (C36B11) #9733参照) やこれらのクロマチンに関連するポリコーム抑制複合体タンパク質 (EZH2 (D2C9) XP® Rabbit mAb #5246参照) に対する抗体と共に使用した場合も、うまく機能することが分かっています (下図を参照してください)。
ご希望の標的に特異的な抗体の性能に関わらず、CUT&Tagアッセイ自体が機能していることを確認するために、CUT&Tagで検証済みのポジティブコントロール抗体 (Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751など) を実験で使用することを推奨します。CUT&Tagアッセイの場合、ネガティブコントロールには、使用するピーク抽出アルゴリズムの要件に応じてIgG抗体またはインプットサンプルのいずれかを使用できます。どちらのネガティブコントロールにも利点と制限があります。インプットDNAサンプルは、Tn5によりタグメンテーションされていない断片化されたゲノムDNAであるため、CUT&Tagサンプルと同じ実験で比較するためには、ライブラリー調製のための超音波による断片化やin vitroアダプターライゲーションステップなどの、追加の作業が必要となります。インプットDNAサンプルは常に、データの解析に十分なライブラリー収量とシーケンシングリードを生成します。CUT&Tagシグナルは非常に強力ですが、インプットDNAサンプルをネガティブコントロールとして使用する場合に、バックグラウンドシグナルの一部がピークとしてカウントされる場合があります。この問題は、IgGコントロールを使用することにより回避できます。IgGは、合理的で信頼性の高いピーク抽出において、優れた性能を発揮します (下図を参照してください)。ただし、IgGコントロールを使用すると、ライブラリー収量が低下し、データ解析の際のシーケンシングリードが大幅に少なくなる恐れがあります。研究室によっては、ネガティブコントロールは全く使用せずにCUT&Tagピークを解析するところもあります。CUT&Tag Assay Kit#に77552は、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb#9751、Normal Rabbit IgG #2729、Normal Mouse IgG #68860のすべてのコントロール抗体が含まれた便利なキットです。
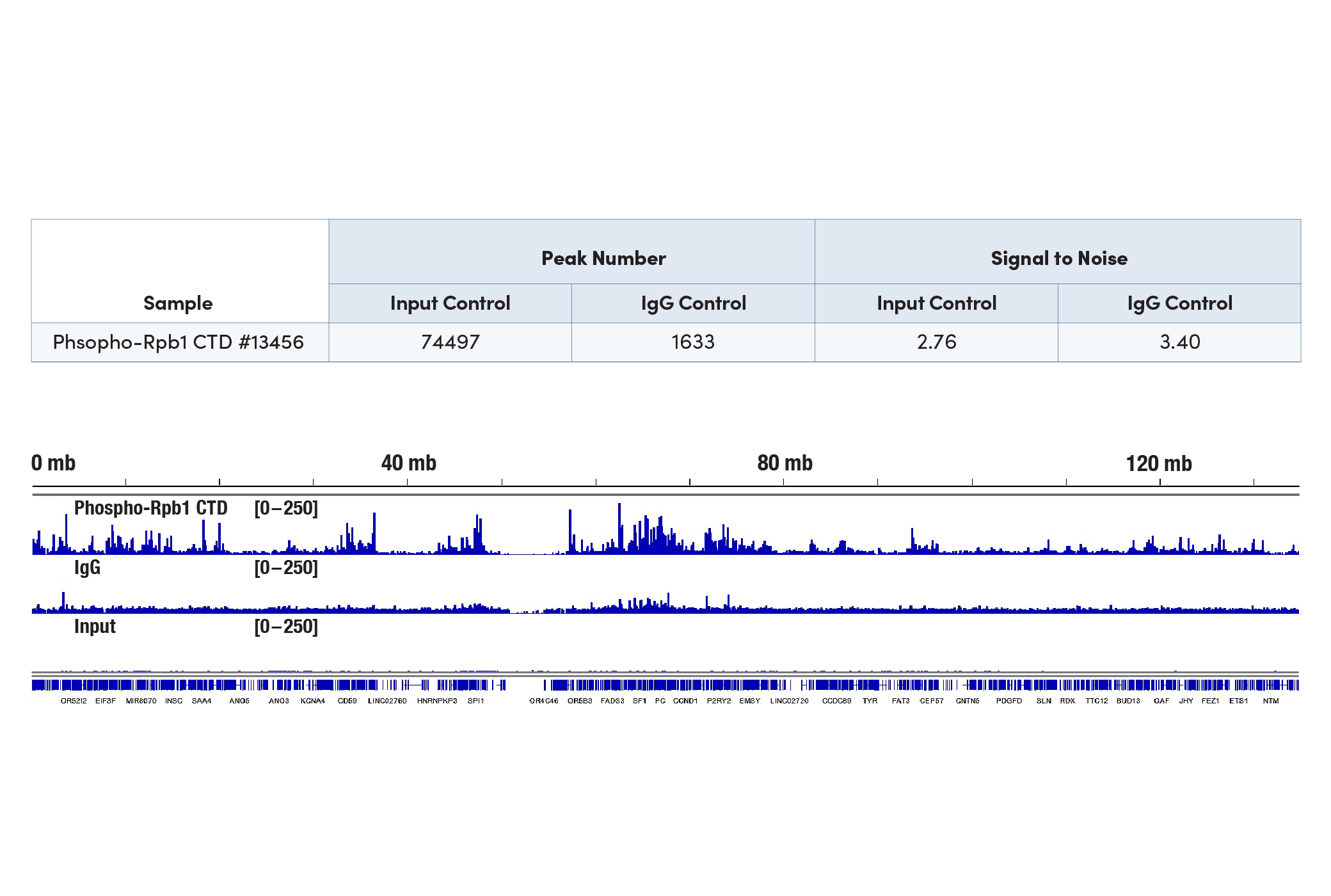
100,000個のHeLa細胞を、Phospho-Rpb1 CTD (Ser2/Ser5) (D1G3K) Rabbit mAb #13546またはNormal Rabbit IgG #2729のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAのインプットサンプルは、CUT&RUNのプロトコールのSection V (CUT&RUN Assay Kit #86652) に従い調製しました。Rpb1とIgGサンプルにはCUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415、インプットDNAサンプルにはDNA Library Prep Kit for Illumina Systems (ChIP-seq, CUT&RUN) #56795を用いてDNAライブラリーを調製しました。図は、11番染色体全体にわたる濃縮を示しています。IgGサンプルは、インプットサンプルよりも多くのバックグラウンドシグナルを示し、それに伴い、より妥当な数のピークを生成することが分かります (左図を参照してください)。
CSTの科学者は、オープンクロマチン領域における非特異的なタグメンテーション (CUT&Tagアッセイ特有の問題) を示す傾向のある抗体を除外するために精力的に社内試験を実施しています。そのため、弊社はCSTのCUT&Tag検証済み抗体を用いてアッセイを始めることを常に推奨しています。もしも、CUT&Tag検証済み抗体がない場合は、CUT&RUN検証済み抗体から開始することを推奨します。他の方法として、ChIPやChIP-seq、免疫蛍光染色アッセイ検証済み抗体を試すことを推奨します。CUT&Tagにおける抗体の結合は無傷の核内で行われるため、免疫蛍光染色アッセイにおける抗体の結合条件に類似しています。CUT&Tagで検証されていない抗体を使用する場合は、非特異的なタグメンテーションがないことを確認することが不可欠です。この確認を行わない場合、結果の信頼性が欠ける可能性があります。
CUT&Tag DNAを精製するときは、CST DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN, CUT&Tag) #14209の使用を推奨します (CUT&Tag Assay Kit #77552にも含まれます)。フェノール/クロロホルム抽出に続いてエタノール沈殿を行う方法では、あらゆる大きさのDNA断片を回収できますが、CSTのDNAスピンカラムは35 bp以上または同等のDNA断片を効果的に回収します。一方タグ付けされたゲノムDNA断片の大きさは、70 bp以上です。したがって、CSTのDNAスピンカラムは、CUT&Tagで得られるDNA断片を精製するための簡便かつ経済的で、確かな方法と言えます。
qPCR解析をご希望の場合は、CUT&RUNの実施を推奨します。CUT&Tag DNAはqPCR解析に適していません。CUT&Tagプロトコールにおいて、タグ付けされたDNAを可溶化するために必要な58℃でのサンプルのインキュベーションステップは、核膜を破壊するため、最終的なCUT&Tag DNAサンプルは、標的となるタグ付けされたDNAとバックグラウンドとなるゲノムDNAの混合物となります。最終的なCUT&Tag DNAサンプルで標的遺伝子に対するプライマーを使用したqPCRを行っても、CUT&Tagシグナルとバックグラウンドシグナルを区別する方法はありません。しかし、タグ付けされたDNAは、ライブラリーの増幅中に選択的に濃縮される一方、ゲノムDNAは希釈されるため、CUT&Tag DNAライブラリーのqPCR解析は可能です。そのため、NGSの前の品質チェックステップとして、CUT&Tag DNAライブラリーのqPCR解析を行うことができます。CUT&Tag DNAライブラリーの調製には、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415の使用を推奨します。
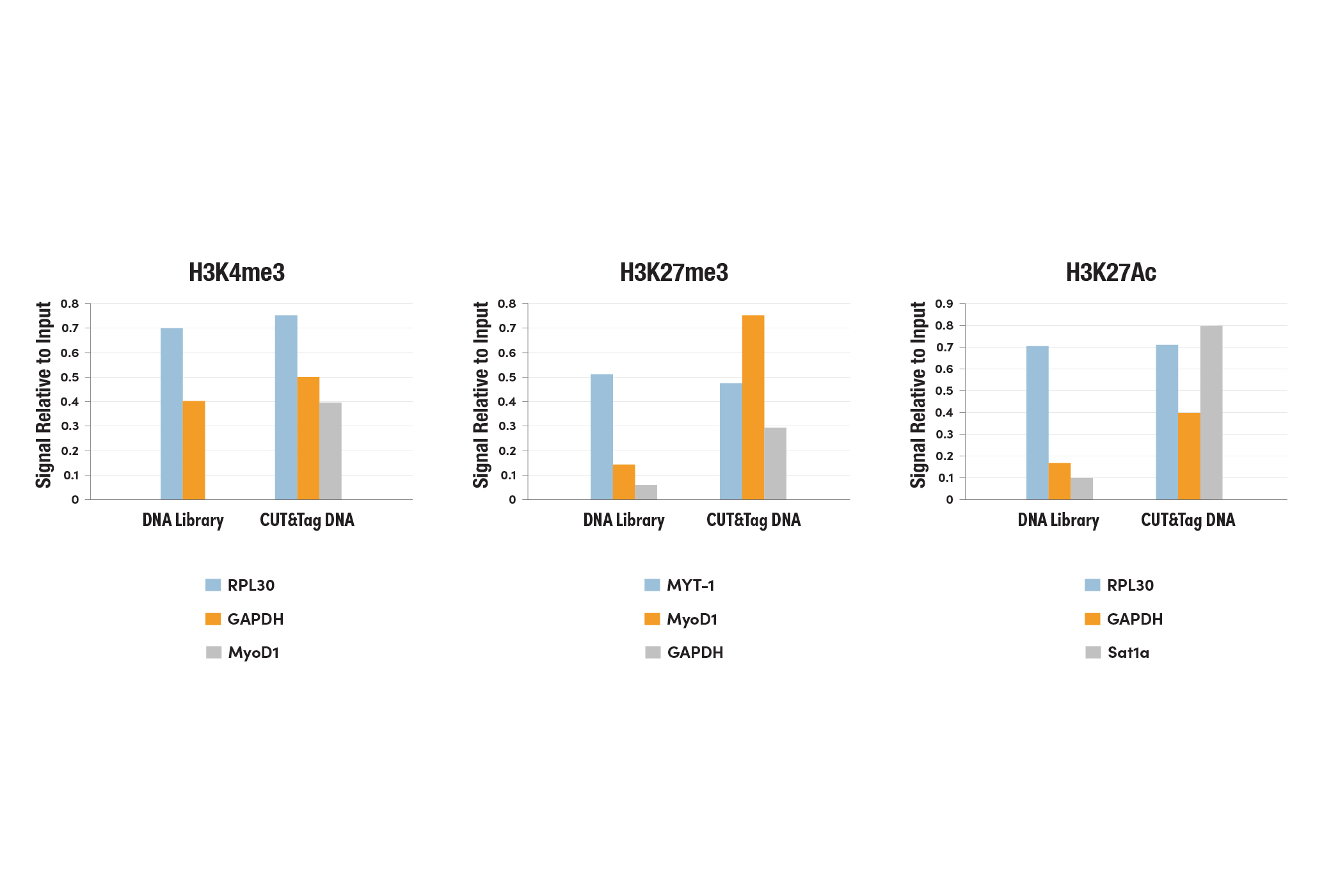
左パネル: HeLa細胞を、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751とCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。ポジティブプライマーセットとしてSimpleChIP® Human RPL30 Exon 3 Primers #7014 and human GAPDH exon 1 primers、ネガティブプライマーセットとしてSimpleChIP® Human MyoD1 Exon 1 Primers #4490を用いて濃縮したDNAをリアルタイムPCRで定量しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。
中央パネル: HeLa細胞を、Tri-Methyl-Histone H3 (Lys27) (C36B11) Rabbit mAb #9733とCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。ポジティブプライマーセットとしてSimpleChIP® Human MyoD1 Exon 1 Primers #4490 and human MYT-1 exon 1 primers、ネガティブプライマーセットとしてhuman GAPDH exon 1 primersを用いて濃縮したDNAをリアルタイムPCRで定量しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。
右パネル: HeLa細胞を、Acetyl-Histone H3 (Lys27) (D5E4) XP® Rabbit mAb #8173とCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。ポジティブプライマーセットとしてSimpleChIP® Human RPL30 Exon 3 Primers #7014 and human GAPDH exon 1 primers、ネガティブプライマーセットとしてSimpleChIP® Human α Satellite Repeat Primers #4486を用いて濃縮したDNAをリアルタイムPCRで定量しました。インプットクロマチンの総量を1とし、各サンプルで得られたDNA量の相対値を示しています。
増幅されたCUT&Tag DNAライブラリーの予想収量は、使用するDNA定量システムによって様々です。NanoDropまたはQIAxpert Systemを使用する場合に予想される一般的なライブラリー濃度は、ヒストン標的については10-20 ng/µL、ヒストン以外の標的については5-12 ng/µLです。NanoDropまたはQIAxpert Systemで測定したライブラリー濃度が3 ng/µL以下である場合は、サンプルをシーケンシングする前にトラブルシューティングガイドを参照してください。Qubit蛍光定量システムまたはPicogreenアッセイを使用する場合に予想される一般的なライブラリー濃度は、ヒストン標的については3-10 ng/µL、ヒストン以外の標的については1 ng/µL以下です。収量の低さが、必ずしもCUT&Tag実験の失敗を意味するわけではありません。ポジティブコントロールのTri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751で予想されるライブラリー収量が得られる、またはDNAライブラリーのqPCRによる品質チェックで良好なシグナル対ノイズ比が得られる場合は、NGSに進むことを推奨します。収量が低いという理由で、NGSに進むのを断念する必要はありません。
CUT&Tag DNAの収量は一般的に低いことが予想されるため、ライブラリーの増幅にCUT&Tag DNAサンプル30 µL (CUT&Tagプロトコール#77552のセクションVI) をすべて使用することを推奨します。ヒストン修飾の解析用に調製されたCUT&Tag DNAライブラリーは通常、Agilent社のBioanalyzerまたはTapeStationシステム上である程度のシグナルを示しますが、転写因子やコファクターなどのヒストン以外のタンパク質の解析用に調製されたライブラリーは、多くの場合、BioanalyzerまたはTapeStationシステム上で非常に弱いシグナルを示すか、シグナルが全くみえない場合があります。その場合でも、高いマッピング率、多数の結合ピーク、ゲノム全体にわたる適切なシグナル対ノイズ比を示すNGS結果を作成作成できます (下図を参照してください)。BioanalyzerまたはTapeStationシステムがライブラリーで断片の平均的なサイズを特定できないCUT&Tagライブラリーの場合は、900 bpの大きさの、通常の収量のライブラリーよりも低い収量のライブラリーを意図的にプールすることを推奨します。
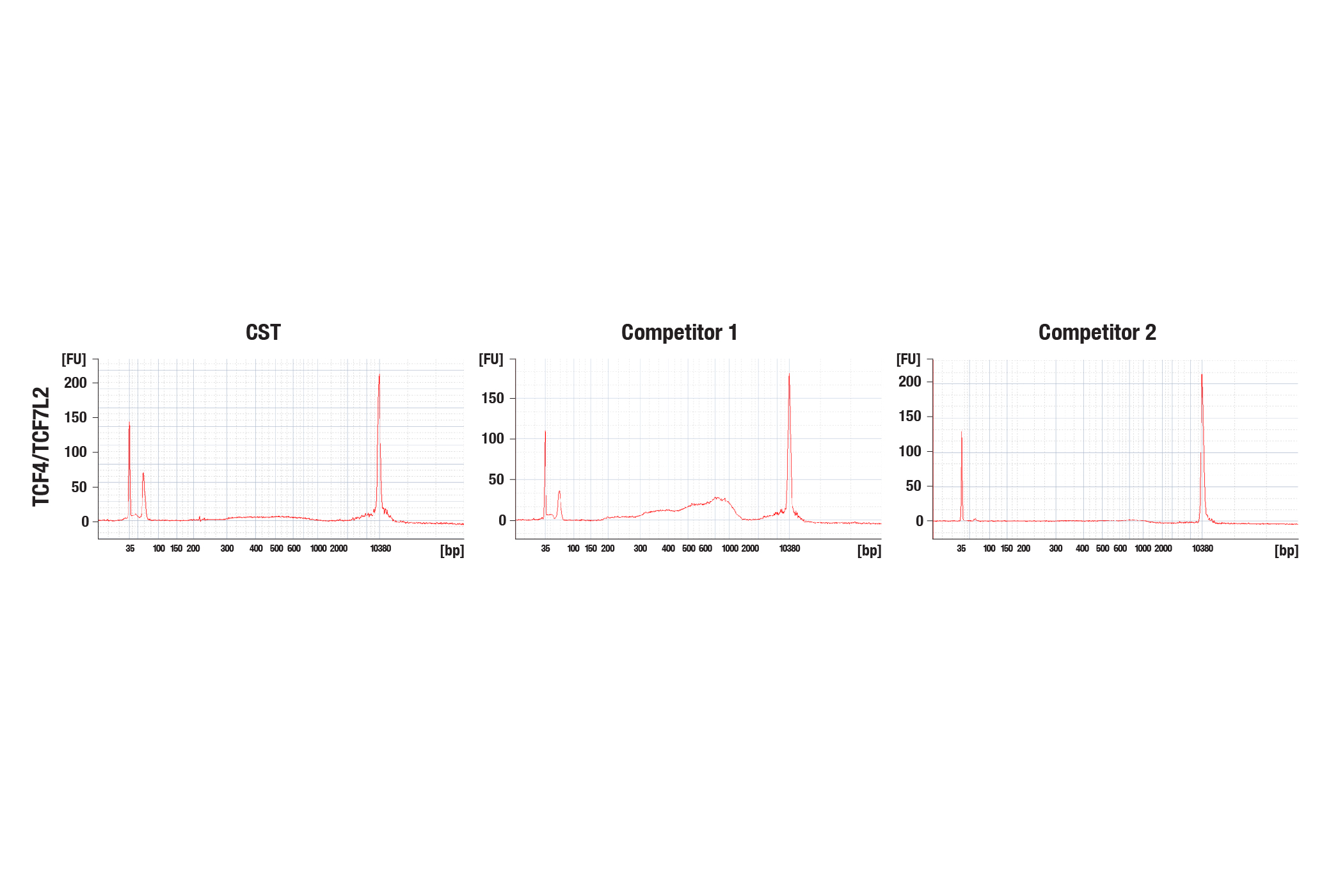
HCT 116細胞を、TCF4/TCF7L2 (C48H11) Rabbit mAb #2569と他社製品を含むいくつかのpAG-Tn5を用いてCUT&Tagで解析しました。各酵素の使用量は、メーカーの推奨量に基づいています。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina #47415を用いて調製しました。上の図は、Bioanalyzerシステムで得られたCUT&TagライブラリーDNAのプロファイルを示しています。
下の図は、Bioanalyzerシステムで解析したものと同じDNAライブラリーをシーケンシングし、得られたNGSトラックを示しています。NGSの結果から、8番染色体全体への結合 (上) およびTCF4/TCF7L2の既知の標的遺伝子であるMYCへの結合 (下) において、同等の結果が得られたことが分かります。
通常、ヒストンを標的とするCUT&Tag DNAライブラリーは、ヒストン以外を標的とするDNAライブラリーよりも濃度が高くなります。弊社では、プールする目的で各ライブラリーサンプルを同じ濃度 (nM) に希釈する前に、次の式を使用してライブラリー濃度をng/µLからnMに変換しています:濃度 (nM) = 1,000,000 X 濃度 (ng/µL) / ライブラリーの平均サイズ (bp) / 660。BioanalyzerまたはTapeStationシステムで検出できないライブラリーサンプルに対してより大きなサイズのライブラリーをプールするだけでなく (上記の質問への回答も参照してください)、BioanalyzerまたはTapeStationシステムで正常なサイズのピークを示すライブラリーよりも5-10倍多くのフラットなシグナルを持つライブラリーをプールすることも推奨します。これにより、すべてのサンプル間で読み取り数が均等に分散されます。通常、NGS用であれば、プールしたDNAライブラリーの濃度は2 nMで十分ですが、濃度はより高いに越したことはありません。
CUT&Tag DNAを用いたDNAライブラリーの調製には、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を推奨します。CSTの、DNA Library Prep Kit for Illumina (ChIP-seq, CUT&RUN) #56795、それと組み合わせて用いるMultiplex Oligos for Illumina (Dual Index Primers) (ChIP-seq, CUT&RUN) #47538またはMultiplex Oligos for Illumina (Single Index Primers) (ChIP-seq, CUT&RUN) #29580は、CUT&Tag DNAサンプルには適合していません。
いいえ。CUT&Tagのライブラリー調製の際の、DNAのサイズセレクションは推奨していません。CSTの経験とお客様からいただいたフィードバックを元に検証した結果、サイズセレクションによりライブラリーの多様性と収量が大きく損なわれることが分かっています。CUT&Tagアッセイにおける一般的なDNAの収量を考えると、これはCUT&Tagライブラリーにとって非常に大きな問題となります。大きなバックグラウンドDNA断片 (> 1 kb) によるNGSへの悪影響が懸念される場合は、ライブラリー調製時の、タグ付けされたDNAを増幅するための伸長時間を短縮してください。伸長時間を10-15秒短縮するだけで、1 kb以上の大きなDNA断片の増幅を大きく低減します。上記の時間を短縮しても、必要とするより小さなDNA断片の増幅に問題はありません。
CUT&Tag Assay Kit #77552には、慎重に検討して梱包された、室温や4℃、-20℃のいずれかで保存する必要がある各構成品が含まれています。適切な機能を確保するためには、各試薬を適切な温度で保管することが重要です。誤ってキット全体を-20℃で保存してしまうと、Concanavalin Aビーズの活性が一部失われます (下図を参照してください)。このような場合には、次の実験 (特に、存在量が少ない標的または結合力の弱いDNA結合タンパク質でのアッセイ) 用に、弊社がカスタム販売しているConcanavalin A Magnetic Beads and Activation Buffer #93569を購入されることを推奨します。また、DNAスピンカラムは、ゆっくり解凍し (4℃で解凍してから室温に戻すなど)、カラム内に結露がないことを確認してから使用してください。必要に応じて、DNA Purification Buffers and Spin Columns (ChIP, CUT&RUN, CUT&Tag) #14209も単体でご購入いただけます。キット内のその他の試薬はすべて、解凍後でもご使用いただけます。キット全体を室温で放置した場合、1週間未満であれば、pAG-Tn5 (Loaded) およびConcanavalin Aビーズを含む大部分の試薬は引き続き使用可能です。ただし、スペルミジン溶液とプロテアーゼ阻害剤カクテルは、性能が損なわれる可能性があります。100X Spermidine #27287およびProtease Inhibitor Cocktail (200X) #7012は、どちらも単体でご購入いただけます。
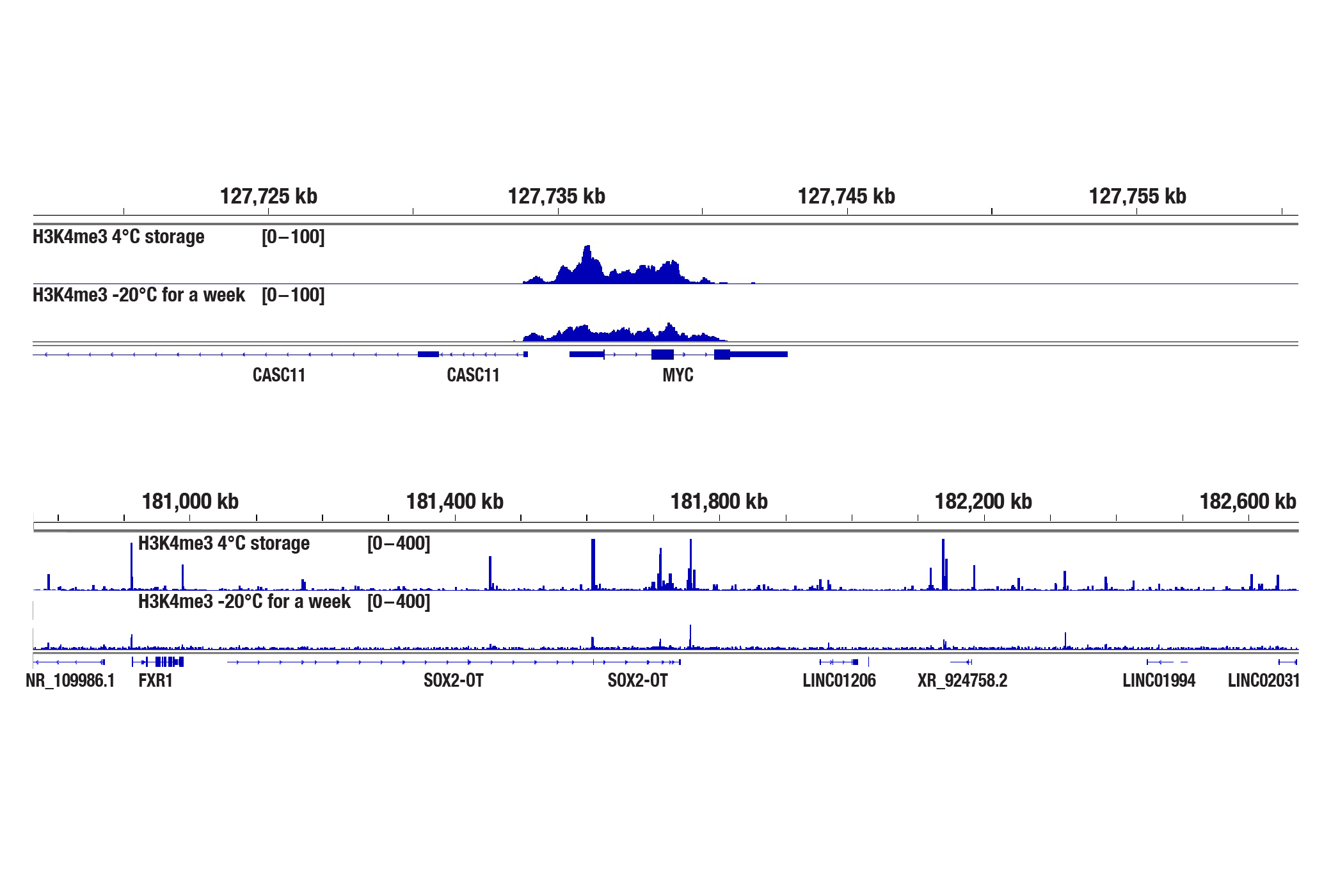
100,000個のNCCIT細胞を、4℃で1週間保存あるいは-20℃で1週間凍結保存したConcanavalin Aビーズと、Tri-Methyl-Histone H3 (Lys4) (C42D8) Rabbit mAb #9751 (上パネル) またはSMARCC1/BAF155 (D7F8S) Rabbit mAb #11956 (下パネル) のいずれかとCUT&Tag Assay Kit #77552を用いてCUT&Tagで解析しました。DNAライブラリーは、CUT&Tag Dual Index Primers and PCR Master Mix for Illumina Systems #47415を用いて調製しました。図は、H3K4me3の既知の標的遺伝子であるMYC全体にわたるH3K4me3の結合 (上パネル) と、BAF複合体の既知の標的遺伝子であるSox2全体にわたるSMARCC1の結合 (下パネル) に示しています。
CST, Cell Signaling Technology, and SimpleChIP are registered trademarks of Cell Signaling Technology, Inc. All other trademarks are the property of their respective owners. Visit cellsignal.com/trademarks for more information.
CUT&Tag provided under a license from Active Motif, Inc. under U.S. Patent No. 10,689,643 and 9,938,524, foreign equivalents, and child patents deriving therefrom. For purchaser's internal research use only. May not be used for resale, services, or other commercial use.
U.S. Patent No. 11,733,248, foreign equivalents, and child patents deriving therefrom.
U.S. Patent No. 7,429,487, foreign equivalents, and child patents deriving therefrom.